2. 浙江省海洋食品品质及危害物质控制技术重点实验室, 杭州 310018
2. Key Laboratory of Marine Food Quality and Hazard Controlling Technology of Zhejiang Province, Hangzhou 310018
抗生素可用于治疗各种细菌性感染疾病, 并且在低剂量情况下可以促进水生动物生长能显著提高养殖的经济效益, 因此抗生素在水产动物养殖中广泛使用并且滥用.这种情况在全球范围内普遍存在(Cabello, 2006), 而我国对于水产养殖中抗生素使用管理不够规范, 更加剧了抗生素滥用的情况(梁惜梅等, 2013).抗生素水体残留消除已成为棘手难题, 而抗生素残留诱导细菌的产生ARGs又成为了新型的污染物(罗义等, 2008;沈怡雯等, 2015).
耐药微生物不仅能够在环境中持续性残留, 在不同的环境介质中迁移、转化、传播和扩散, 最终使环境稳定性遭到破坏, 而且会传播进入食物链, 进而危害人体健康.不仅如此, Huang等发现富含抗性基因的饲料和环境可能会有助于抗性基因在水产养殖环境中传播(Huang et al., 2015).目前已经有很多研究表明海产品可能作为抗生素耐药基因的储存库, 是耐药基因向人类传播的一个重要中介(Adesiji et al., 2014;Ahmed et al., 2015;Huang et al., 2015).Gao等检测了天津地区水生微生物耐药基因, 磺胺类耐药基因是被检率最高的抗性基因, 经进化树分析发现sulⅠ基因可以在肠道杆菌和土著杆菌之间迁移(Gao et al., 2012).ARGs可以在不同微生物之间转移, 加大了向人类传播的几率.研究发现许多引起人类患病的致病菌中均含有抗生素耐药基因, 如Bertsch等分析了不同来源李斯特菌(食源、临床及环境)的抗性基因及基因型, 发现几乎所有的新的抗性元件可以在微生物之间转移(Bertsch et al., 2014), Letchumanan等检测了副溶血性弧菌的耐药基因, 在TRH菌株中发现了氯霉素抗性基因(catA2) 及卡那霉素抗性基因(aphA-2), 预示着ARGs将进一步加大致病菌的危害(Letchumanan et al., 2015).ARGs对人类的健康存在严重的潜在威胁, 对微生物(尤其是水生微生物)的抗生素耐药基因的分子机制等相关内容的研究已成为与民生息息相关的内容.
磺胺类、四环素类、喹诺酮类、新一代氯霉素类(氟苯尼考等)抗生素是我国近年来水产养殖中最常用的4类抗生素(王硕等, 2013).本研究以这4类抗生素相应的耐药基因为研究对象, 采用PCR扩增法对上述4类抗生素进行检测, 并分析鱼塘生态系统中可培养微生物的耐药性.研究结果将为指导抗生素科学使用、预防抗生素滥用引起的生态污染及耐药基因分子机制等更深入的研究提供理论和数据支持.
2 实验方法(Materials and methods) 2.1 实验材料及仪器营养琼脂(NA)、营养肉汤(NB)、NaCl(分析纯)、甲醇(色谱纯)、乙腈(色谱纯)、乙酸乙酯、乙醚、甲酸、氨水、柠檬酸、磷酸二氢钠、磺胺二甲嘧啶标准溶液、恩诺沙星标准溶液、四环素标准溶液、氟苯尼考标准溶液、PCR引物(上海生工合成)、细菌基因组(北京庄盟生物)、质粒组DNA小量提取试剂盒(北京庄盟生物);电泳系统(Tanon EPS600)、凝胶呈像系统(Tanon 3500)、高速冷冻离心机(Eppendorf 5810R)、Oasis HLB柱、Agilent 1200高效液相色谱仪、Agilent 6410三重四极杆串联质谱仪、MN-913超微量分光光度计、高速PCR仪(德国耶拿)、快速混匀器、真空泵、过滤膜.
2.2 样品采集在杭州市某水产养殖区挑选养殖时间均在2年以上的6个鱼塘(土塘, 分别标记为:W1、W2、S3、S4、S5、S6) 采集底泥, 每个池塘随机选择4个位点采集底泥, 等份混合均匀.样品置于无菌玻璃瓶中, 低温(冰浴)运输至实验室, 置于4 ℃冰箱中保藏, 并在24 h内进行微生物分离.
2.3 菌株分离培养取20 g底泥样品溶解于含180 mL 0.85%的无菌生理盐水的三角瓶, 混匀后吸取1 mL样品悬液依次进行梯度稀释获得10-2、10-3、10-4、10-5、10-6、10-7 g·mL-1浓度样品液.
2.3.1 涂布分离培养取不同稀释度的样品100 μL涂布于NA琼脂平板进行筛选, 每个样品设置一个平行, 30 ℃培养48 h.
2.3.2 划线分离纯化从细菌总数在30~300 CFU·mL-1范围的平板上随机挑取20株菌株进行划线分离纯化, 30 ℃培养24 h.
2.3.3 二次划线分离纯化选取一次划线纯化得到的单菌落进行二次划线分离, 置于30 ℃培养24 h.
2.3.4 液体培养将二次分离纯化后的单菌落接种于含5 mL NB液体培养基的试管中, 置于30 ℃培养48 h, 获得新鲜菌液.
2.4 抗生素浓度测定采集的样品按四分法缩减, 于室内风干后粉碎过60目筛备测.准确称取2.0 g样品置于锥形瓶中, 用乙腈-磷酸缓冲液和EDTA超声提取4次, 经SAX-HLB柱浓缩纯化和6 mL甲醇洗脱后氮气吹至近干, 用1.0 mL甲醇复溶, 密封冷藏至4 ℃备用.其余步骤参照:磺胺二甲嘧啶的浓度测定参照农业部1025公告-23—2008, 恩诺沙星浓度测定参照GB/T 20366—2006, 四环素浓度测定参照GB/T 21317—2007, 氟苯尼考浓度测定参照GB/T 22338—2008.
2.5 DNA提取取新鲜菌液3 mL, 参照质粒和基因组DNA抽提试剂盒说明书进行基因提取.
2.6 耐药基因扩增根据文献(Guillard et al., 2011;Tamminen et al., 2011;Luo et al., 2010;郑朝朝等, 2010), 采用磺胺二甲嘧啶(磺胺类药物)、四环素(四环素类药物)、恩诺沙星(喹诺酮类药物)及新一代氯霉素类药物氟苯尼考4种抗生素相应耐药基因的引物和整合子的引物分别对质粒和细菌基因组DNA进行扩增, 所选基因及其引物见表 1.
| 表 1 4种抗生素耐药基因扩增引物及退火温度列表 Table 1 PCR Primers and annealing temperature for ARG amplification |
PCR扩增条件:95 ℃预变性4 min;95 ℃变性30 s, 退火条件见表 1, 72 ℃延伸30 s, 40个循环;72 ℃, 10 min.
PCR扩增体系:25 μL体系, 包括模板DNA 1 μL, 上下游引物各1 μL(10 μmmol·L-1), Taq酶0.2 μL(5 U·μL-1), dNTP Mix 2 μL, 10×PCR扩增缓冲液2.5 μL, 无菌水17.3 μL.
取PCR产物3 μL, 2%的琼脂糖凝胶电泳检20 min后于凝胶呈相系统下检测结果.
2.7 16S rDNA扩增27F 5′-TCCTACGGGAGGCAGCAGT-3′
1492R 5′-GGACTACCAGGGTATCTAATCCTGTT-3′
PCR扩增条件:95 ℃预变性4 min;95 ℃, 20 s, 58 ℃, 30 s, 72 ℃, 40 s, 40个循环;72 ℃, 10 min.
PCR扩增体系:25 μL体系, 包括模板DNA 1 μL、上下游引物各1 μL(10 μmmol·L-1)、Taq酶0.2 μL (5 U·μL-1)、dNTP Mix 2 μL、10×PCR扩增缓冲液2.5 μL、无菌水17.3 μL.
取PCR产物3 μL, 1%的琼脂糖凝胶电泳20 min后于凝胶成相系统下检测结果.
2.8 16S rDNA测序及NCBI数据库Blast比对分析16S rDNA PCR产物送至上海生物工程技术有限公司进行序列测定, 测序结果经NCBI上在线BLAST检索该细菌的种属, 并提交至NCBI GENEBANK.
3 结果与分析(Results and analysis) 3.1 鱼塘底泥微生物的分离本实验共从6个鱼塘采集获得6份底泥样品, 从每个样品随机挑选获得20株菌, 合计120株菌, 经纯化及二次纯化后存74株可培养.
3.2 鱼塘底泥抗生素浓度测定6份底泥样品的抗生素浓度测定后, 氟苯尼考在S5及S6鱼塘中检出, 浓度分别为19.8 ng·kg-1及12.1 ng·kg-1.磺胺二甲嘧啶和恩诺沙星在所有鱼塘中均检出, 见表 2.而四环素则在各鱼塘均未检出.由于没有做追踪调查研究, 4种抗生素的被检出浓度不同的主要原因可能是各鱼塘的发病情况不同, 投放的药物情况也不同.各个鱼塘中的抗生素浓度结果见表 2.
| 表 2 鱼塘中抗生素的检出浓度 Table 2 Concentrations of antibiotics in aquaculture ponds |
74株菌经耐药基因PCR检测后, 22株存在intⅠ基因特异性扩增片段, 12株存在floR基因特异性扩增片段, 13株存在sulⅡ基因特异性扩增片段, 37株存在qnrS基因特异性扩增片段.除floR基因外, 其余耐药基因均获得单一的目标条带, 片段大小与预期扩增片段大小相吻合, 部分结果见图 1.分离菌株耐药基因在6个塘中的分布情况见表 3.
 |
| 图 1 部分菌株耐药基因PCR片段电泳图(注: 1~4:分离菌株S6-10、S6-14、S6-15、S4-15基因组DNA intⅠ扩增片段;5~6:分离菌株W1-5和W1-19质粒DNA floR扩增片段; 7~8:分离菌株S5-8和S5-5质粒DNA sulⅡ扩增片段;9~15:分离菌株S5-7、S5-8、S5-9、S5-10、S5-14、S5-16和S5-18质粒DNA qnrS扩增片段.M代表DNA marker泳道) Fig. 1 Electrophoresis results of some isolates′ ARGs amplifications DNA electrophoresis of some strains |
| 表 3 鱼塘中抗生素耐药基因分布情况 Table 3 Distribution of antibiotic resistance genes in aquaculture ponds |
在底泥样品分离菌株耐药基因PCR扩增检测所得各抗生素ARGs检出率如图 2所示.喹诺酮类ARGs qnrS耐药基因的检出率较高, 为50.00%;磺胺类ARGs sulⅡ耐药基因和氯霉素类ARGs floR耐药基因的检出率均较低, 分别为17.56%和16.21%;而四环素类ARGs tetA耐药基因未检出.整合子intⅠ基因的检出率也相对较低, 为29.72%.与其他3种ARGs相比, qnrS的检出率最高.表明大部分菌株对喹诺酮类抗生素有耐药性, 而对磺胺类及氯霉素类抗生素的耐药性较则不明显, 对四环素无耐药性.其中含单种耐药基因(整合子及耐药基因中的一种)菌株有40株, 检出率为54.05%;含双重耐药基因(整合子及耐药基因中的两种)菌株有6株, 检出率为8.11%;含多重耐药基因(整合子及耐药基因中的3种)菌株有10株, 检出率为13.51%.结果见图 3.含耐药基因(整合子及耐药基因)的菌株总数有56株, 检出率为75.68%, 表明大部分菌株含有耐药基因或整合子.
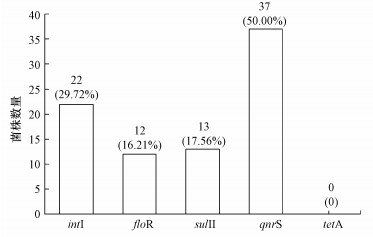 |
| 图 2 74株分离菌株中各类耐药基因分布(数字代表菌株数量, 百分数代表所占比率) Fig. 2 Distribution of different ARGs among the isolates(Number represents the number of isolates, percentage represents the proportion.) |
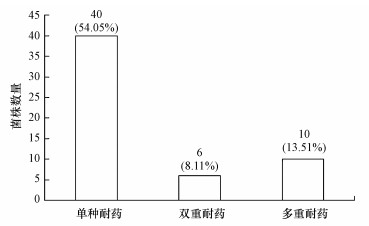 |
| 图 3 单种、双重及多重耐药菌检出结果(数字代表菌株数量, 百分数代表所占比率) Fig. 3 Single, double and triple ARGs detected among the isolates(Number represents the number of isolates, percentage represents the proportion.) |
74株菌的16S rDNA序列信息Blast比对分析结果表明, 本实验分离菌株的序列与genebank数据库中已有序列匹配率均达到98%及以上, 可分为12个属, 19个种:9株气单胞菌(Aeromonas spp.)、8株维氏气单胞菌(Aeromonas veronii)、6株嗜水气单胞菌(Aeromonas hydrophila)、3株斑点气单胞菌(Aeromonas punctata)、1株双壳气单胞菌(Aeromonas bivalvium)、1株达卡气单胞菌(Aeromonas dhakensis)及1株简氏气单胞菌(Aeromonas jandaei); 1株成晶节杆菌(Arthrobacter crystallopoietes)、1株硬结节杆菌(Arthrobacter scleromae);6株海水芽孢杆菌(Bacillus aquimaris)、2株黄海芽孢杆菌(Bacillus marisflavi)、2株芽孢杆菌属(Bacillus spp.)、1株解淀粉芽孢杆菌(Bacillus amyloliquefaciens)及1株弯曲芽孢杆菌(Bacillus flexus);2株粘着剑菌(Ensifer adhaerens)及1株剑菌属(Ensifer sp.);1株肠杆菌属(Enterobacter sp.);1株藤黄微球菌(Micrococcus luteus);7株假单胞菌属(Pseudomonas spp.)、3株类产碱假单胞菌(Pseudomonas pseudoalcaligenes)及2株铜绿色假单胞菌(Pseudomonas aeruginosa);1株霍乱弧菌(Vibrio cholerae);1株根瘤菌属(Rhizobium sp.);2株红球菌属(Rhodococcus spp.);1株脱色希瓦氏菌(Shewanella decolorationis);1株葡萄球菌(Staphylococcus condimenti);8株未培养菌株(Uncultured bacterium).各菌属中含有ARGs及整合子的菌株数量及比例见表 4.16S rDNA序列信息在GENBANK数据库中的登录号为:KY496244-KY496309(可培养菌株)和KY486248-KY486255(不可培养菌株).
| 表 4 不同种属分离菌株ARGs检出数量及检出率 Table 4 ARGs detection number and rate of the isolates in different genera |
本研究检测了采集的6个塘底泥中的抗生素浓度, 并用PCR的方法分析了各个塘中抗生素耐药基因的分布情况, 结果发现各塘中的检出抗生素浓度及耐药基因分布有所不同, 见表 2及表 3.74株分离菌株中, 37株菌检出qnrS耐药基因, 13株菌检出sulⅡ耐药基因, 12株菌检出floR耐药基因, 其检出率分别为50.00%、17.33%和16.00%.氟苯尼考是一种新型广谱抗生素, 对鱼虾的细菌性疾病治疗具有良好的效果, 被称作氯霉素的替代品, 在W1、W2、S4、S5及S6塘中均检测出floR, 其中塘W1中含floR菌株数量最多, 而氟苯尼考仅在塘S5及S6中被检出.磺胺类抗生素是一种广谱抗生素, 在人类疾病治疗及养殖动物疾病预防治疗中均有大量使用, 本研究中磺胺二甲嘧啶及耐药基因sulⅡ检出最多的塘均是W1, 其余塘中也均有磺胺二甲嘧啶及少数sulⅡ检出, 说明该养殖场可能在W1塘中投放较多的磺胺二甲嘧啶, 过量的药物残留增加了耐药基因的积累.喹诺酮类药物主要用于气单胞菌引起的病害, 本研究中所有塘中均有检出, 与之相关的qnrS不但在所有塘中均被检出, 而且是检出率最高的耐药基因, 说明该养殖场中可能比较多的使用喹诺酮类药物来控制疾病.四环素是一类传统抗生素, 现今使用量已经远远不及往年, 在所有塘中四环素及耐药基因tetA未被检出.从结果可以发现各个塘中的耐药基因分布与检出抗生素浓度有所不同, 造成此种原因的可能情况是不同塘的病发情况不同, 使用各种药物的种类和量均有差别, 加之抗生素耐药基因在某些微生物中天然存在, 尽管比例较低, 但在不同环境选择压力下, 耐药基因会呈现不同程度的富集.由于未做追踪调查, 对于未检出抗生素却存在相应耐药基因的情况(如W1、W2及S4中氟苯尼考), 推测可能与取样时间有关, 取样时间距离养殖中抗生素的投放时间较长, 加之养殖水体的更换均会导致抗生素检出浓度较低或不被检出.
3.6 菌株种属及耐药基因分析从表 4中可以看出:29株气单胞菌中普遍存在floR、sulⅡ及qnrS, 菌株数分别为10、11、28, 检出率分别为34.38%、37.93、96.55%, 但intⅠ在该菌中未被检出.而芽孢杆菌中的耐药基因分布情况与气单胞菌相反, intⅠ主要集中分布在芽孢杆菌中, 测得12株芽孢杆菌属菌中有11株菌存在整合子intⅠ耐药基因, 检出率为91.67%, 其余4种耐药基因均未检出.本研究共分离12株假单胞菌属菌, 检出3株含有intⅠ, 其余3种耐药基因均未检出.intⅠ能捕获和表达多种耐药基因, 是抗生素耐药基因在全球传播的一种重要途径, 通常嵌在质粒及转座子中, 利于转入范围更广的治病株中.在intⅠ存在时可能含有其他耐药基因, 由于本研究只检测4种耐药基因, 其他的耐药基因可能未被检测到.不同菌种携带耐药基因的偏好性不同, 比如研究发现气单胞菌属中有87.2%的A.dhakensis(34/39) 及90%的A.hydrophila(18/20) 中存在耐药基因cphA, 而分离的14株A.caviae均不含cphA(Sinclair et al., 2016).从本研究已有结果可以看出气单胞菌易于携带floR、sulⅡ及qnrS, 芽孢杆菌和假单胞菌易于携带intⅠ, 因此不同菌种在传播耐药基因时的作用可能不同, 在未来的研究种应该更加深入了解各种菌携带耐药基因的偏好性, 为控制耐药基因传播提供理论依据, 并指导养殖场合理用药.
本研究从养殖鱼塘底泥样品分离的微生物中, 气单胞菌属所占比例最高, 为39.19%, 其次为芽孢杆菌属和假单胞菌属, 各占16.22%.气单胞菌是一种条件致病菌, 广泛分布于水环境中, 对水产动物具有广泛的致病性, 可引起疖病, 是我国淡水养殖业发展的主要威胁.抗生素是治疗疖病的主要药物, 在本研究中28株检出qnrS耐药基因, 占气单胞菌总数的96.55%.随着全球含ARGs的气单胞菌数量的增加, 逐渐引起人们重视, 大量的研究表明, 自喹诺酮类药物用于防治气单胞菌病害以来, 嗜水气单胞菌对喹诺酮类药物已经产生了较大的耐药(薛慧娟等, 2012), 其耐药基因主要分布在质粒上, 并且环境微生物比如气单胞菌可能作为抗生素耐药基因的储存库(Hatosy et al., 2015).本研究进一步证实了该结论, 说明该鱼塘中使用较多喹诺酮类药物来控制气单胞菌引起的感染.本研究中所测12株假单胞菌属中有3株菌含有floR耐药基因, 不含其他3种耐药基因及整合子intⅠ, 说明假单胞菌属不易产生或整合耐药基因.芽孢杆菌在水产养殖中属有益菌, 常作为饲料添加剂使用, 其在生长繁殖过程中能产生多种维生素, 为养殖生物提供营养, 并降低水中化学需氧量COD, 改善养殖水环境.本研究中整合子ARGs intⅠ的检出率为29.72%, PCR结果显示节杆菌属、芽孢杆菌属、肠杆菌属、剑菌属、微球菌属、假单胞菌属、葡萄球菌属及不可培养细菌的菌株中均检测出整合子ARGs intⅠ耐药基因, 且大多来自于芽孢杆菌属.intⅠ能捕获和表达多种耐药基因, 是抗生素耐药基因在全球传播的一种重要途径.Luo等研究发现在海河沉积物中, intⅠ与sulⅠ的相对含量之间存在显著相关性, 并证实intⅠ介导了sulⅠ在环境中的水平传播(Luo et al., 2010).较高的intⅠ检出率说明本实验的芽孢杆菌中可能含有其他种类的抗生素耐药基因, 有待进一步研究, 但耐药基因在养殖环境中益生菌内检出也提示我们ARGs存在迁移、传播的风险.本次实验中仅分离培养出1株弧菌属细菌, 原因可能是采样的季节在5月份, 属于弧菌滋生的淡季, 经检测该菌株含有floR耐药基因.
3.7 鱼塘底泥微生物耐药性与水生环境的关系进入环境介质中的抗生素可以引起包括抑制有益微生物的活性, 对生态系统的物质循环和能量流动造成干扰, 影响植物、动物和微生物的生长和健康等相关的环境问题, 对生态系统稳定构成潜在性的风险(俞慎等, 2011).胡大胜研究表明, 水产养殖中各养殖区域、各养殖模式和各类菌株的耐药性各不相同, 细菌病必须根据药敏试验结果来用药防治才能确保疗效(胡大胜, 2013).滥用抗生素使环境中产生抗生素选择压力, 使耐药菌株及耐药基因富集, 耐药微生物不仅能够在环境中持续性残留, 在不同的环境介质中迁移、转化、传播和扩散(Perreten et al., 1997;Beaber et al., 2004)最终使环境稳定性遭到破坏, 而且会传播进入食物链, 人类健康构成巨大威胁.本实验中74株菌有56株菌被检测到含有整合子或者4种耐药基因, 被检率高达75.68%, 说明该养殖场中的含耐药基因菌株数量很多, ARGs在养殖场中广泛存在的问题应引起人们关注.水产养殖场作为一个重要的耐药基因储存库, 应加强对水产养殖场中抗生素耐药基因的监测及研究, 通过指导合理用药、研发抗生素替代品等方法控制ARGs基因在环境中的传播.
Adesiji Y O, Deekshit V K, Karunasagar I. 2014. Antimicrobial-resistant genes associated with Salmonella spp. isolated from human, poultry, and seafood sources[J]. Food Science & Nutrition, 2(4): 436–442.
|
Ahmed A M, Maruyama A, Khalifa H O, et al. 2015. Seafood as a reservoir of gram-negative bacteria carrying integrons and antimicrobial resistance genes in Japan[J]. Biomedical and Environmental Science, 28(12): 924–927.
|
Beaber J W, Hochhut B, Waldor M K. 2004. SOS response promotes horizontal dissemination of antibiotic resistance genes[J]. Nature, 427(6969): 72–74.
DOI:10.1038/nature02241
|
Bertsch D, Muelli M, Weller M, et al. 2014. Antimicrobial susceptibility and antibiotic resistance gene transfer analysis of foodborne, clinical, and environmental Listeria spp. isolates including Listeria monocytogenes[J]. Microbiologyopen, 3(1): 118–127.
DOI:10.1002/mbo3.2014.3.issue-1
|
Cabello F C. 2006. Heavy use of prophylactic antibiotics in aquaculture: a growing problem for human and animal health and for the environment[J]. Environmental Microbiology, 8(7): 1137–1144.
DOI:10.1111/emi.2006.8.issue-7
|
Gao P, Mao D, Luo Y, et al. 2012. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment[J]. Water Research, 46(7): 2355–2364.
DOI:10.1016/j.watres.2012.02.004
|
Guillard T, Moret H, Brasme L, et al. 2011. Rapid detection of qnr and qepA plasmid-mediated quinolone resistance genes using real-time PCR[J]. Diagnostic Microbiology & Infectious Disease, 70(2): 253–259.
|
Hatosy S M, Martiny A C. 2015. The ocean as a global reservoir of antibiotic resistance genes[J]. Applied & Environmental Microbiology, 81(21): 7593–7599.
|
Huang Y, Zhang L, Tiu L, et al. 2015. Characterization of antibiotic resistance in commensal bacteria from an aquaculture ecosystem[J]. Frontiers in Microbiology, 6: 914.
|
胡大胜. 2013. 2012年广西水产养殖致病菌耐药性调查[J]. 海洋与渔业·水产前沿, 2013(3): 75–77.
|
Letchumanan V, Yin W F, Lee L H, et al. 2015. Prevalence and antimicrobial susceptibility of Vibrio parahaemolyticus isolated from retail shrimps in Malaysia[J]. Frontiers in Microbiology, 6(33): 33.
|
梁惜梅, 施震, 黄小平. 2013. 珠江口典型水产养殖区抗生素的污染特征[J]. 生态环境学报, 2013, 22(2): 304–310.
|
Luo Y, Mao D, Rysz M, et al. 2010. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 44(19): 7220–7225.
|
罗义, 周启星. 2008. 抗生素抗性基因(ARGs)——一种新型环境污染物[J]. 环境科学学报, 2008, 28(8): 1499–1505.
|
Perreten V, Schwarz F, Cresta L, et al. 1997. Antibiotic resistance spread in food[J]. Nature, 389(6653): 801–802.
|
沈怡雯, 黄智婷, 谢冰. 2015. 抗生素及其抗性基因在环境中的污染、降解和去除研究进展[J]. 应用与环境生物学报, 2015, 21(2): 181–187.
|
Sinclair H A, Heney C, Sidjabat H E, et al. 2016. Genotypic and phenotypic identification of Aeromonas species and CphA-mediated carbapenem resistance in Queensland, Australia[J]. Diagnostic Microbiology & Infectious Disease, 85(1): 98–101.
|
Tamminen M, Karkman A, Lõhmus A, et al. 2011. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure[J]. Environmental Science & Technology, 45(2): 386–391.
|
王硕, 张晶, 邵兵. 2013. 超高效液相色谱-串联质谱测定污泥中氯霉素、磺胺类、喹诺酮类、四环素类与大环内酯类抗生素[J]. 分析测试学报, 2013, 32(2): 179–185.
|
薛慧娟, 邓玉婷, 姜兰, 等. 2012. 水产动物源嗜水气单胞菌药物敏感性及QRDR基因突变分析[J]. 广东农业科学, 2012, 39(23): 149–153.
DOI:10.3969/j.issn.1004-874X.2012.23.048 |
俞慎, 王敏, 洪有为. 2011. 环境介质中的抗生素及其微生物生态效应[J]. 生态学报, 2011, 31(15): 4437–4446.
|
郑朝朝, 刘聚祥, 李辉, 等. 2010. 猪源大肠杆菌耐氟苯尼考基因的检测及序列分析[J]. 畜牧与兽医, 2010, 42(3): 62–65.
|
 2017, Vol. 37
2017, Vol. 37


