多环芳烃(Polycyclic Aromatic Hydrocarbons, PAHs)是一类典型的、分布广泛的有机污染物, 由于其具有致癌、致畸、致突变的“三致”作用, 而备受人们关注(Cai et al., 2007; Oliveira et al., 2015).此类化合物均含有十分稳定的环结构, 且具有一定的疏水性、高度的非极性和亲脂性, 不易被降解并容易在环境中沉降积累, 从而给生态环境带来巨大的威胁.此外, 环境中积累的PAHs还可通过食物链进行迁移, 直接危害人类的健康(Van et al., 2003; Xia et al., 2010; Hamdan et al., 2017).菲(Phenanthrene)是一种3个苯环以角状方式排列的典型PAHs, 是美国环保署所列的16种优先控制的典型PAHs之一, 常被用作研究PAHs污染的模式化合物(Seo et al., 2009).
微生物在环境PAHs的去除中具有不可替代的作用(Lu et al., 2011b), 当前, 包括菲在内的一些PAHs的微生物有氧降解机制已经被阐明.如在好氧条件下, 恶臭假单胞菌能以菲为唯一碳源和能源生长, 并通过水杨酸途径对菲进行降解.其基本降解过程为:首先菲在C-3, 4位上被双加氧酶羟基化, 随后利用一系列相关酶转化为1-羟-2-萘甲酸, 之后在羟化酶的催化作用下生成1, 2-二羟萘, 再通过不同降解酶的作用逐步最终转化为水杨酸及邻苯二酚(张丹等, 2010).而邻苯二酚的降解又可分为两条途径, 一条是生成顺, 顺-己二烯二酸, 另一条则是生成2-羟基黏糠酸半醛后, 分别进入三羧酸循环, 完全降解成二氧化碳和水(Kiyohara et al., 1982; Yang et al., 1994).
然而, PAHs的厌氧降解也不可忽视(Haritash et al., 2009), 如在沉积物及土壤等存在大量微氧或厌氧的环境中, 硝酸盐、硫酸盐、铁还原及产甲烷等厌氧代谢过程可能与PAHs的厌氧分解过程偶联(侯晓鹏等, 2016).当前, 菲在反硝化条件下的降解也已经被人们所证实.Mcnally等(1998)研究了3株反硝化细菌在厌氧条件下对蒽、菲、芘的降解, 发现它们均能在一定程度上降解3~4环的PAHs, 且蒽的降解速率高于菲, 而芘的降解速率最慢.然而, 降解PAHs的反硝化微生物的纯培养并不多见, 且仅限于少数几种PAHs(Rockne et al., 2000).另一方面, 微生物的PAHs共代谢已经被人们所认识, 尤其在土壤复杂环境下, 反硝化条件下PAHs的降解也不可能由单一或少数几种微生物参与.因此, 近年来PAHs厌氧代谢菌群的富集研究也越来越为人们所关注(杨旭等, 2014; 张荣秋等, 2014).如Lu等(2011)从海洋沉积物富集获取了能够厌氧降解PAHs的混合菌群, 包括19株γ-变形菌纲(γ-Proteobacteria)细菌和4株放线杆菌纲(Actinobacteria)细菌.可见, 富集培养是获取PAHs厌氧代谢微生物的有效手段, 能够为深入探讨PAHs厌氧代谢的机理及环境PAHs污染的修复提供有力的研究对象及理论依据.
本研究以江汉油田区域的农田土壤为对象, 富集获取土壤中的菲反硝化降解菌群, 测定其对菲的降解效率.同时, 利用定量PCR测定其中反硝化相关功能基因的丰度, 并通过Illumina MiSeq测序对其中的细菌群落结构进行解析, 从而为土壤PAHs污染的生物修复提供一定的理论依据, 并为进一步深入探究菲的反硝化微生物降解机制奠定基础.
2 材料与方法(Materials and methods) 2.1 土壤样品的采集及富集用培养基土壤样品采集于具有50多年历史的江汉油田区域的农田表层(0~20 cm)土壤(112°43′E, 30°49′N).土壤类型为潮土, 主要理化性质如下: pH=8.2, 有机质24.95 g·kg-1, 硝态氮21.84 mg·kg-1, 铵态氮4.16 mg·kg-1, 有效磷15.94 mg·kg-1, PAHs 0.80 mg·kg-1.
富集菲反硝化降解菌群用的培养基(PDN)参照Lu等(2011)的无机盐液体培养基配方, 在此基础上分别配置以下各组分.A液: Na2CO3 3.46 g·L-1、NaNO3 0.85 g·L-1、KH2PO4 0.0342 g·L-1、K2HPO4 0.0858 g·L-1; B液: CaCl2 9 g·L-1; C液: MgSO4·7H2O 29.44 g·L-1; D液: NiSO4·7H2O 9.2 g·L-1、(NH4)2MoO4·4H2O 0.7 g·L-1、NaBO2·10H2O 0.3 g·L-1; E液: FeCl3·6H2O 7.1 g·L-1、MnCl2·4H2O 1.9 g·L-1、ZnCl2 1 g·L-1、CoCl2·2H2O 1 g·L-1、CuCl2·2H2O 0.5 g·L-1.A、B和C液分别于高压蒸汽灭菌锅灭菌(121 ℃, 30 min), 备用; D、E液经滤膜(0.25 μm)过滤除菌后, 备用.每1000 mL A液中依次加入5 mL B液、5 mL C液、2 mL D液、2 mL E液, 用于富集培养.
2.2 反硝化富集培养本研究中, 通过在PDN培养基中添加菲(单一碳源)并接种土壤来富集菲反硝化降解菌群, 初次富集共设置了10个加土的富集处理, 同时设置了不加土的对照(CK).具体的富集培养过程如下:首先, 在100 mL的血清瓶中加入1 mL 0.5 g·L-1的菲丙酮溶液, 室温下, 在通风橱抽气30 min后(使丙酮挥发), 加入5 g土壤; 随后, 加入灭菌的PDN培养基45 mL, 使菲的终浓度为10 mg·L-1, 用丁基胶塞及铝盖密封后, 向瓶内充入高纯氮气5 min, 以排除血清瓶中的空气, 再用加水的注射器平衡血清瓶内外压强, 并用凡士林涂布于铝盖上; 最后, 将密闭好的血清瓶放于25 ℃恒温摇床(160 r·min-1)进行避光培养, 培养过程中每隔6 d充入高纯氮气进行换气, 培养12 d后, 从各瓶中分别吸取5 mL培养物接种至45 mL新鲜的培养液中, 进行下一轮富集.在第4次富集培养结束后测定各血清瓶中的菲含量, 筛选获得3个能够明显降解菲的反硝化菌群(PDN-1、PDN-2、PDN-3), 它们在反硝化条件下对菲的降解率分别为40%、30%、20%.以上述3个具有不同菲降解率的富集培养物为研究对象, 再次转接培养, 各富集培养物均接种3瓶, 另外设置只接种5 mL无菌水的对照(CK)3个.按上述方法进行培养, 最终测定菲的含量并计算降解率.
2.3 菲的提取及含量测定菲的提取依据国家标准(HJ 478—2009), 采用整瓶萃取再浓缩法, 以正己烷(液相色谱纯)为萃取剂对富集培养液进行抽提和测定.具体步骤如下:将50 mL富集培养液倒入250 mL分液漏斗中, 加入1.5 g NaCl, 用35 mL正己烷分3次洗涤血清瓶, 并倒入分液漏斗中, 振摇5 min, 充分萃取, 静置分层, 收集有机相; 重复萃取3遍后合并有机相, 加适量无水Na2SO4, 轻摇静置; 于旋转蒸发仪上45 ℃蒸发有机相至5 mL左右; 加入25 mL甲醇(液相色谱纯), 53 ℃旋蒸浓缩至3~4 mL, 转移到比色管中并用甲醇定容至5 mL, 摇匀后过0.45 μm滤膜待测; 最后, 以菲的标准品(Dr. Ehrenstorfer, 德国)配制标准曲线, 用高效液相色谱仪HITACHI L-7100(Hitachi High-Technologies Corporation, 日本)来测定菲含量, 并通过公式(1)计算菲的降解率P.
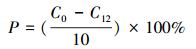
|
(1) |
式中, C0为培养结束后CK处理中菲的浓度(mg·L-1); C12为培养结束后各富集培养物处理中菲的浓度(mg·L-1).
2.4 培养物基因组DNA的提取及narG、nirS基因的定量将富集培养液(20 mL)于20 ℃、8000 r·min-1下离心10 min收集菌体, 用Fast DNA® SPIN Kit for Soil(MP Biomedicals, 美国)试剂盒, 参照其说明书提取富集菌群的总DNA, 并用1%的琼脂糖凝胶电泳进行检测.
反硝化相关的narG及nirS基因丰度的检测在实时荧光定量PCR仪(ABI 7500, Applied Biosystems, 美国)上, 用SYBR GREEN法进行.定量PCR过程参照Premix Ex TaqTM(TaKaRa Biotechnology, 日本)的说明书进行, 反应采用25 μL体系(12.5 μL SYBR EroTaq 2X, 正反向引物各1 μL, 0.5 μL Ro-DYe, 1 μL DNA模板, 9 μL ddH2O), 基因扩增所用的引物序列及扩增程序见表 1.实验中标准曲线的制备参照He等(2007)的方法进行.在各反应完成后, 设置了溶解曲线程序, 用来检测定量PCR产物的特异性, 最后通过ABI 7500 Software(version 2.0.6)来分析所得的数据, 计算各基因拷贝数.
| 表 1 定量PCR所用引物及扩增程序 Table 1 Primer sets and amplification program used for quantitative PCR |
为了对各富集培养物中的细菌群落结构进行解析, 将提取的各样品DNA送上海美吉生物信息公司进行Illumina MiSeq测序.简要流程如下:首先, 以各样品DNA为模板用引物对515F: GTGCCAGCMGCCGCGG和907R: CCGTCAATTCM TTTRAGTTT(Zhou et al., 2011)对细菌16S rDNA进行PCR扩增(ABI GeneAmp® 9700, 美国), PCR产物经切胶回收后, 用Tris-HCl洗脱, 并用QuantiFluorTM-ST蓝色荧光定量系统(Promega, 美国)进行定量检测和混合, 构建Illumina平台文库并测序.使用mothur软件(version v.1.30.1)对粗序列进行质控筛选和过滤, 按照97%相似性对非重复序列(不含单序列)进行OTU(Operational Taxonomic Unit)聚类, 在聚类过程中去除嵌合体, 得到各OTU的代表序列, 并计算Coverage、Chao1及Shannon指数.
2.6 数据处理实验数据的统计分析在SPSS 17.0软件中, 用单因素方差分析的S-N-K(Student-Newman-Keuls test)法检验不同样品之间的差异显著性(p < 0.05表示差异显著).用SPSS中的Pearson相关性检验进行菲降解率与反硝化功能基因丰度(narG、nirS及narG/nirS)之间的相关分析.在R3.3.2软件(http://cran.r-project.org)中利用Pheatmap package构建基于各富集样品优势细菌属的热图.
3 结果(Results) 3.1 富集培养菌群对菲的降解富集培养物PDN-1中菲的浓度最低, 降解率为45.18%, 显著高于PDN-3的降解率(p < 0.05), PDN-2菲降解率为34.04%, 而PDN-3的降解率只有25.92%, 二者之间无显著性差异(图 1).
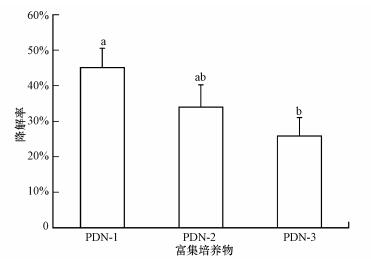 |
| 图 1 3种富集培养物对菲的降解率(误差线为标准偏差(n=3), 小写字母指示菲降解率在样品间的显著性差异(p < 0.05)) Fig. 1 Degradation rate of phenanthrene in the enriched consortiums |
由图 2可知, 富集菌群PDN-1中narG的丰度(3.29×105 copies·mL-1)显著低于PDN-2(2.43×106 copies·mL-1)和PDN-3 (2.62×106 copies·mL-1)(p < 0.05), 而PDN-2中nirS的丰度(9.44×105 copies·mL-1)显著高于PDN-1(2.34×105 copies·mL-1)及PDN-3(2.64×105 copies·mL-1)(p < 0.05).Pearson相关分析表明, 菲降解率与narG基因丰度呈显著负相关(r=-0.94, p < 0.01), 但与nirS的丰度无显著相关性(r=-0.13, p=0.81).此外, PDN-1和PDN-3分别具有最低和最高的narG与nirS的丰度比(narG/nirS), 相关分析的结果表明, 菲的降解率与narG/nirS有显著的负相关关系(r=-0.88, p < 0.05).各富集培养物中narG的丰度均显著高于nirS的丰度(p < 0.05), 这表明与亚硝酸盐还原过程相比硝酸盐还原过程可能在菲的反硝化降解过程中占主导地位.
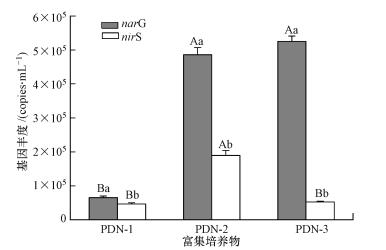 |
| 图 2 3种富集培养物中narG及nirS基因的丰度(误差线为标准偏差(n=3), 大小写字母分别指示narG及nirS基因在样品间和样本内的显著性差异(p < 0.05)) Fig. 2 Abundances of narG and nirS genes in the enriched consortiums |
由表 2可知, 从3个富集菌群PDN-1(35321)、PDN-2(34645)、PDN-3(38742)中获取了较为接近的总序列数.在97%的相似度水平下, 它们的Coverage指数均超过99.9%, 表明测序结果能够真实反映各样品中的细菌群落结构.具有最高菲降解率的PDN-1中的OTU最多, 且其Chao1和Shannon指数也均高于其它两个富集样品.样品PDN-2的OTU及Chao1指数均高于PDN-3, Shannon指数低于PDN-3.可见, 菲的降解能力可能与富集样品细菌的多样性存在正相关, 这也进一步说明PAHs等复杂化合物的降解可能需要多种微生物来协同完成.
| 表 2 3种富集培养物中细菌的多样性指数 Table 2 Bacterial diversity indices in the enriched consortiums |
各富集培养物中细菌门水平的群落结构如图 3所示.可见, 变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)和拟杆菌门(Bacteroidetes)是3个富集物中的优势细菌门.其中, Proteobacteria在PDN-1(97.78%)、PDN-2(96.57%)、PDN-3(93.90%)中的比例均超过其它两个门, Bacteroidetes在PDN-1和PDN-2中的比例低于PDN-3, 但PDN-1和PDN-2中Verrucomicrobia的比例高于PDN-3.
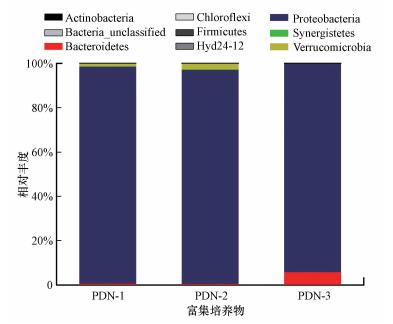 |
| 图 3 3种富集培养物中细菌门水平上的群落结构 Fig. 3 Bacterial community structures based on the phyla in the enriched consortiums |
基于各富集样品中优势菌属构建的热图(图 4)表明, 3个富集菌群中的优势属均为假单胞菌属(Pseudomonas)和嗜甲基菌属(Methylophilus), 但它们在不同富集物中的比例并不相同.其中, Pseudomonas在PDN-1中的比例最高(58.30%), 在PDN-2中最低(23.37%), 而Methylophilus在PDN-2中最高(65.92%), 在PDN-1中最低(20.05%).
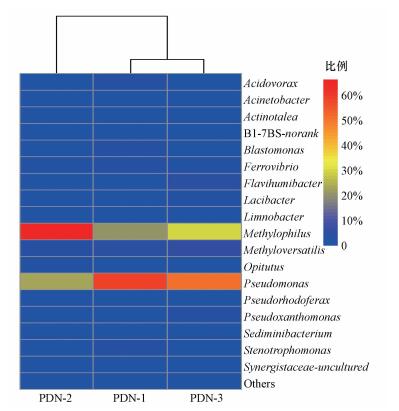 |
| 图 4 基于富集培养菌群细菌属组成构建的热图 Fig. 4 Heat map showing the bacterial genus compositions in the enriched consortiums |
本研究获取的3个菲的反硝化降解菌群12 d内对菲的降解率在25.92%~45.18%之间, 这与当前报道的反硝化条件下微生物对菲的降解率一致.如Lu等(2011a)报道, 在以硝酸盐为单一电子受体的条件下, 12 d内海洋沉积物中菲的降解率在50%左右.此外, 本研究中反硝化富集菌群对菲的降解率高于已有的一些纯培养单菌株及产甲烷富集菌群(Liang et al., 2014; Mcnally et al., 1998).如据Liang等(2014)报道, 厌氧条件下单菌株JP1(假单胞菌属)40 d内对菲的降解率仅为5%.有研究报道了一种产甲烷富集菌群27 d内对菲的降解率为20%~30%(张书颖等, 2011).可见本研究富集出较为高效的菲反硝化降解菌群, 且富集的反硝化降解菌群可能较单菌株及其它还原条件的富集菌群具有更强的菲代谢能力.
培养结束后, 各富集菌群中narG的丰度均显著高于nirS, 这可能表明在反硝化富集菌群代谢菲的过程中, 硝酸盐还原的强度高于随后的亚硝酸盐还原过程.当前, 关于反硝化过程中电子受体还原与PAHs降解偶联机理的研究还不多见, 但有研究表明, 硝酸盐还原过程可能在PAHs的反硝化降解中占主导地位.如Yang等(2013)发现, 其获取的反硝化菌群在以NO3-作为唯一电子受体代谢芘的过程中有明显的NO2-积累, 该结果从物质转化的角度证实了硝酸盐还原作用在PAHs反硝化降解过程中的主导作用.此外, 吴艳阳等(2012)的研究表明, 受到2~3环PAHs污染的河流沉积物中narG的丰度高于nirS, 这也可说明反硝化过程中的硝酸盐还原阶段可能在低分子量PAHs代谢中起主导作用.
细菌多样性及群落结构分析的结果表明, 具有最高菲降解率的富集菌群PDN-1, 同时也有最高的细菌多样性, 表明菲降解率与富集物细菌多样性具有一定的正相关.Sharma等(2016)报道, 3株纯培养的菌株好氧条件下各自对菲的降解率在14%~27%之间, 但含3种菌的复合菌群对菲的降解率高达67.8%.再如, Guo等(2010)从红树林沉积物中分离出21株PAHs降解菌, 且发现其中的鞘氨醇单胞菌属和分枝杆菌属的混合菌群具有比单一菌株更强的PAHs降解能力, 可见细菌多样性组成会影响其对PAHs的降解能力.变形菌门为本研究获取的富集物中最大的优势门, 该门涵盖了假单胞菌属(Pseudomonas)、产碱杆菌属(Alcaligenes)、根瘤菌属(Rhizobium)等常见的反硝化细菌(方晶晶等, 2010).该门中的Pseudomonas和Methylophilus则是本研究中各富集菌群中的最优势菌属, 其中, Pseudomonas是广泛存在于水、土壤等环境中的重要PAHs降解菌(Ahn et al., 1998).当前分离到的能够降解萘、菲及芘的好氧或厌氧的纯培养菌株也多属于Pseudomonas(Liang et al., 2014; Lin et al., 2014; 黄兴如等, 2016).Pseudomonas在菲降解率最高的PDN-1中的比例最高, 可见它在菲的反硝化代谢中起到了重要作用.另一个优势菌属为Methylophilus, 因其具备利用芳香烃降解过程中产生的甲基类物质为碳源进行生长的特点, 被认为具有作为油气勘探或油气污染指示菌的潜质(满鹏等, 2012).因此, 本研究中Methylophilus极有可能是因为菲降解过程中产生的中间代谢物而得以富集.疣微菌门(Verrucomicrobia)是各富集菌群中仅次于变形菌门的优势门, 该门的细菌可能是土壤和沉积物中CH4代谢的参与者(郑勇等, 2009).目前, 还鲜有关于疣微菌门的细菌降解PAHs的报道, 但有研究表明, PAHs污染能够影响该门细菌的多样性, 在芘污染的白三叶草根际也观察到疣微菌门数量的增加(Kawasaki et al., 2012; 2016).综上, 变形菌门的Pseudomonas在反硝化的菲降解中起主导作用, 而Methylophilus及疣微菌门中的细菌可能在菲的反硝化代谢中具有与前者相互协作的功能, 且它们在富集培养物中的不同比例可能导致不同的菲降解效率.
5 结论(Conclusions)1) 富集到的3个菲降解菌群在以菲为单一碳源的培养基中12 d内对菲的降解率为25.92%~45.18%.
2) 培养结束后, 各富集菌群中narG基因的丰度均显著高于nirS基因, 且基因的丰度与菲降解率无显著相关关系.
3) 菲降解率与富集物细菌多样性具有一定的正相关.变形菌门是所有富集物中最大的优势门, 且其中的Pseudomonas(γ-Proteobacteria)和Methylophilus(β-Proteobacteria)为最大的优势菌属.
Botsou F, Hatzianestis I. 2012. Polycyclic aromatic hydrocarbons (PAHs) in marine sediments of the Hellenic coastal zone, eastern Mediterranean: levels, sources and toxicological significance[J]. Journal of Soils and Sediments, 12(2): 265–277.
DOI:10.1007/s11368-011-0453-1
|
Cai Q Y, Mo C H, Li Y H, et al. 2007. Occurrence and assessment of polycyclic aromatic hydrocarbons in soils from vegetable fields of the Pearl River Delta, South China[J]. Chemosphere, 68(1): 159–168.
DOI:10.1016/j.chemosphere.2006.12.015
|
Crampon M, Bureau F, Akpa-Vinceslas M, et al. 2014. Correlations between PAH bioavailability, degrading bacteria, and soil characteristics during PAH biodegradation in five diffusely contaminated dissimilar soils[J]. Environmental Science and Pollution Research, 21(13): 8133–8145.
|
方晶晶, 马传明, 刘存富. 2010. 反硝化细菌研究进展[J]. 环境科学与技术, 2010, 33(6E): 206–210.
|
Guo C L, Dang Z, Wong Y, et al. 2010. Biodegradation ability and dioxgenase genes of PAH-degrading Sphingomonas and Mycobacterium strains isolated from mangrove sediments[J]. International Biodeterioration & Biodegradation, 64(6): 419–426.
|
Hamdan H Z, Salam D A, Hari A R, et al. 2017. Assessment of the performance of SMFCs in the bioremediation of PAHs in contaminated marine sediments under different redox conditions and analysis of the associated microbial communities[J]. Science of the Total Environment, 575: 1453–1461.
DOI:10.1016/j.scitotenv.2016.09.232
|
Haritash A K, Kaushik C P. 2009. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs):a review[J]. Journal of Hazardous Materials, 169(1/3): 1–15.
|
He J Z, Shen J P, Zhang L M, et al. 2007. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 9(9): 2364–2374.
DOI:10.1111/emi.2007.9.issue-9
|
侯晓鹏, 李春华, 叶春, 等. 2016. 不同电子受体作用下微生物降解多环芳烃研究进展[J]. 环境工程技术学报, 2016(1): 78–84.
|
黄兴如, 张彩文, 张瑞杰, 等. 2016. 多环芳烃降解菌的筛选、鉴定及降解特性[J]. 微生物学通报, 2016, 43(5): 965–973.
|
Kawasaki A, Warren C R, Kertesz M A. 2016. Specific influence of white clover on the rhizosphere microbial community in response to polycyclic aromatic hydrocarbon (PAH) contamination[J]. Plant and Soil, 401(1): 1–15.
|
Kawasaki A, Watson E R, Kertesz M A. 2012. Indirect effects of polycyclic aromatic hydrocarbon contamination on microbial communities in legume and grass rhizospheres[J]. Plant and Soil, 358(1): 169–182.
|
Kiyohara H, Nagao K, Kouno K, et al. 1982. Phenanthrene-degrading phenotype of Alcaligenes faecalis AFK2[J]. Applied and Environmental Microbiology, 43(2): 458–461.
|
Liang L, Song X, Kong J, et al. 2014. Anaerobic biodegradation of high-molecular-weight polycyclic aromatic hydrocarbons by a facultative anaerobe Pseudomonas sp.JP1.[J]. Biodegradation, 25(6): 825–833.
DOI:10.1007/s10532-014-9702-5
|
Lin M, Hu X, Chen W, et al. 2014. Biodegradation of phenanthrene by Pseudomonas, sp.BZ-3, isolated from crude oil contaminated soil[J]. International Biodeterioration & Biodegradation, 94: 176–181.
|
Lu X, Zhang T, Fang H P, et al. 2011. Biodegradation of naphthalene by enriched marine denitrifying bacteria[J]. International Biodeterioration & Biodegradation, 65(1): 204–211.
|
Lu X Y, Li B, Zhang T, et al. 2011a. Enhanced anoxic bioremediation of PAHs-contaminated sediment[J]. Bioresource Technology, 104(1): 51–58.
|
Lu X Y, Zhang T, Fang H H P. 2011b. Bacteria-mediated PAH degradation in soil and sediment[J]. Applied Microbiology and Biotechnology, 89(5): 1357–1371.
DOI:10.1007/s00253-010-3072-7
|
满鹏, 齐鸿雁, 呼庆, 等. 2012. 利用PCR-DGGE分析未开发油气田地表微生物群落结构[J]. 环境科学, 2012, 33(1): 305–313.
|
McNally D L, Mihelcic J R, Lueking D R. 1998. Biodegradation of three-and four-ring polycyclic aromatic hydrocarbons under aerobic and denitrifying conditions[J]. Environmental Science & Technology, 32(17): 2633–2639.
|
Michotey V, Méjean V, Bonin P. 2000. Comparison of methods for quantification of cytochrome, cd1-denitrifying bacteria in environmental marine samples[J]. Applied & Environmental Microbiology, 66(4): 1564–1571.
|
Oliveira M, Slezakova K, Fernandes A, et al. 2015. Biomonitoring of firefighters occupational exposure to polycyclic aromatic hydrocarbons during the 2014 hot season[J]. Toxicology Letters, 238(2): S358.
|
Philippot L, Piutti S, Martinlaurent F, et al. 2002. Molecular analysis of the nitrate-reducing community from unplanted and maize-planted soils[J]. Applied & Environmental Microbiology, 68(12): 6121–6128.
|
Pinyakong O, Habe H, Omori T. 2003. The unique aromatic catabolic genes in sphingomonads degrading polycyclic aromatic hydrocarbons (PAHs)[J]. Journal of General & Applied Microbiology, 49(1): 1–19.
|
Rockne K J, Cheesanford J C, Sanford R A, et al. 2000. Anaerobic naphthalene degradation by microbial pure cultures under nitrate-reducing conditions[J]. Applied & Environmental Microbiology, 66(4): 1595–1601.
|
Sangwan P, Chen X, Hugenholtz P, et al. 2004. Chthoniobacter flavus gen.nov.sp.nov.the first pure-culture representative of subdivision two, Spartobacteria classis nov.of the Phylum Verrucomicrobia[J]. Applied & Environmental Microbiology, 70(10): 5875–5881.
|
Seo J S, Keum Y S, Li Q X. 2009. Bacterial degradation of aromatic compounds[J]. International Journal of Environmental Research & Public Health, 6(1): 278–309.
|
Shao M, Zhang T, Fang H H P. 2009. Autotrophic denitrification and its effect on metal speciation during marine sediment remediation[J]. Water Research, 43(12): 2961–2968.
DOI:10.1016/j.watres.2009.04.016
|
Sharma A, Singh S B, Sharma R, et al. 2016. Enhanced biodegradation of PAHs by microbial consortium with different amendment and their fate in in-situ condition[J]. Journal of Environmental Management, 181: 728–736.
DOI:10.1016/j.jenvman.2016.08.024
|
Tian W, Bai J, Zhao Y, et al. 2014. The effect of irrigation with oil-polluted water on microbial communities in estuarine reed rhizosphere soils[J]. Ecological Engineering, 70(3): 275–281.
|
Uribe-Jongbloed A, Bishop P L. 2007. Comparative study of PAH removal efficiency under absence of molecular oxygen:effect of electron acceptor and hydrodynamic conditions[J]. Environmental Engineering and Science, 6(4): 367–376.
DOI:10.1139/s06-057
|
Van d O R, Beyer J, Vermeulen N P. 2003. Fish bioaccumulation and biomarkers in environmental risk assessment:a review[J]. Environmental Toxicology & Pharmacology, 13(2): 57–149.
|
Wang Q F, Zhang S Y, Li Z, et al. 2011. Impact of anthracene addition on microbial community structure in soil microcosms from contaminated and uncontaminated sites[J]. Biomedical and Environmental Sciences, 24(5): 543–549.
|
吴艳阳, 吴群河, 黄珊, 等. 2012. 沉积物中多环芳烃对反硝化功能基因垂直分布的影响[J]. 环境科学, 2012, 33(10): 3592–3597.
|
Xia Z, Duan X, Qiu W, et al. 2010. Health risk assessment on dietary exposure to polycyclic aromatic hydrocarbons (PAHs) in Taiyuan, China[J]. Science Total Environmental, 408(22): 5331–5337.
DOI:10.1016/j.scitotenv.2010.08.008
|
Yang X A, Ye J X, Lyu L M, et al. 2013. Anaerobic biodegradation of pyrene by Paracoccus denitrificans under various nitrate/nitrite-reducing conditions[J]. Water, Air, & Soil Pollution, 224(5): 1–10.
|
Yang Y, Chen R F, Shiaris M P. 1994. Metabolism of naphthalene, fluorene, and phenanthrene:preliminary characterization of a cloned gene cluster from Pseudomonas putida NCIB 9816[J]. Journal of Bacteriology, 176(8): 2158–2164.
DOI:10.1128/jb.176.8.2158-2164.1994
|
杨旭, 陈芳艳, 唐玉斌. 2014. 修复PAHs复合污染体系的高效菌群构建及降解特性[J]. 环境工程学报, 2014, 8(1): 360–365.
|
张丹, 李兆格, 包新光, 等. 2010. 细菌降解萘、菲的代谢途径及相关基因的研究进展[J]. 生物工程学报, 2010, 26(6): 726–734.
|
张荣秋, 董纯明, 盛华芳, 等. 2014. 白令海和楚科奇海表层沉积物中多环芳烃降解微生物多样性[J]. 海洋学报, 2014, 36(4): 52–61.
|
张书颖, 谢曙光. 2011. 菲厌氧降解对细菌和古细菌群落的影响[J]. 北京大学学报(自然科学版), 2011, 47(5): 923–928.
|
张银萍, 王芳, 杨兴伦, 等. 2010. 土壤中高环多环芳烃微生物降解的研究进展[J]. 微生物学通报, 2010, 37(2): 280–288.
|
郑勇, 郑袁明, 张丽梅, 等. 2009. 极端环境下嗜热酸甲烷营养细菌研究进展[J]. 生态学报, 2009, 29(7): 3864–3871.
|
中华人民共和国环境保护部发布. 2009. 中华人民共和国国家环境保护标准: HJ 478--2009. 水质多环芳烃的测定-液液萃取和固相萃取高效液相色谱法[S]. 北京: 中国标准出版社
|
Zhou J Z, Wu L Y, Deng Y, et al. 2011. Reproducibility and quantitation of amplicon sequencing-based detection[J]. Isme Journal, 5(8): 1303–1313.
DOI:10.1038/ismej.2011.11
|
Zhou Y, Wei J, Jiang T, et al. 2007. Characterisation of a thermostable catechol-2, 3-dioxygenase from phenanthrene-degrading Pseudomonas sp.strain ZJF08[J]. Annals of Microbiology, 57(4): 503–508.
DOI:10.1007/BF03175346
|
 2017, Vol. 37
2017, Vol. 37


