2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085
CH4是一种重要的温室气体, 其增温潜势在100年时间尺度是等物质的量CO2的25倍(IPCC, 2001).自工业时代以来, CH4对全球温室效应的作用迅速增加, 目前所占比重已高达20%(Knittel et al., 2009).大气CH4的产生由环境中的生物过程主导, 来源于湿地产甲烷古菌、稻田土壤、反刍动物及白蚁的消化系统和微生物氧化甲烷等(Conrad et al., 2009).
一直以来, 人们普遍认为甲烷只能在好氧条件下被消耗(Strous et al., 2004), 反硝化厌氧甲烷氧化反应(nitrite-dependent anaerobic methane-oxidizing, n-damo)的发现改变了这一认识.研究发现, 在厌氧条件下, 甲烷能够以亚硝酸盐作为电子受体而被氧化(Raghoebarsing et al., 2006; Ettwig et al., 2008; 2009;2010; Luesken et al., 2012; Haroon et al., 2013).反硝化厌氧甲烷氧化反应的发现彻底颠覆了人们对甲烷循环的传统理解.
n-damo菌隶属于NC10门, 其全基因序列在2010年首次被获得, 并暂时命名为“Candidatus Methylomirabilis oxyfera”(Ettwig et al., 2010).反硝化厌氧甲烷氧化反应不仅能够消耗CH4, 更连接着地球上碳和氮循环.在土壤氨氧化古菌(AOA)、氨氧化细菌(AOB)及反硝化细菌的作用下, 一部分铵盐和硝酸盐会被转化为亚硝酸盐, 为n-damo反应的发生提供了可能.通过将CH4转化为CO2, n-damo反应可在一定程度上缓解地球温室效应.
迄今为止, n-damo菌已被发现广泛分布于生物反应器、富集培养基及湿地生态系统中(Ettwig et al., 2009; Hu et al., 2009; 2011; Luesken et al., 2011a; 2011b; 2012; Wu et al., 2011a; 2011b).目前, 人们对其在陆地生态系统, 尤其是旱地农田土壤中的垂向分布规律、生物多样性及反应活性还知之甚少.研究n-damo菌在旱地土壤中的分布规律及生物特性, 对减缓地球温室效应及建立更加完善的碳和氮循环具有极其重要的作用.
本研究选取河北省北澧河附近一处长期施肥的小麦田, 采集夏季和冬季土壤柱状样品(0~1 m)为研究对象, 通过扩增、测序、构建克隆文库等分子生物学技术对n-damo菌的群落结构进行研究, 并利用13C和15N稳定同位素示踪技术, 深入探究n-damo菌、AOA、AOB及反硝化细菌间的相互作用关系.
2 材料与方法(Materials and methods) 2.1 样品采集与保存选取河北省北澧河附近旱地农田(27°28′18.49″ N, 114°58′19.45″ E)作为研究对象, 此处农田灌溉河水受到氨氮和硝氮的污染.本次分别采集了夏季和冬季(2013年6月和12月)的土壤柱状样品, 采样深度均为1 m (每20 cm一层).样品分别取3个平行(间距9 m), 将其混合均匀后密封贮存于聚乙烯无菌自封袋中, 4 ℃下运输至实验室, 到达后立即测定理化性质.然后, 一部分样品冷冻干燥后存放于-80 ℃超低温冰箱中, 用于DNA提取及相关分子生物学实验.剩余样品保存于黑暗的4 ℃冷库中, 留待日后测定硝化速率、反硝化速率及反硝化厌氧甲烷氧化反应速率.
2.2 样品理化指标的测定土壤NH4+、NO3-、NO2-含量的测定参考《土壤农化分析》(鲍士旦, 2000), 将样品用浓度为2 mol·L-1的KCl溶液浸提1 h, 经0.45 μm微孔滤膜抽滤后, 用连续流动分析仪(SAN plus, Skalar Analytical B.V., Breda, the Netherlands)测定.依据标准方法测定土壤中总碳(TC)、总氮(TN)和总硫(TS)(Breda, the Netherlands).以土壤550 ℃的烧失量(LOI550)代表总有机质量(张文河等, 2007).土壤pH值需配成土壤/水质量比为1:2.5的溶液后测定.样品指标均进行3次平行测定以减少数据误差.
2.3 样品DNA提取、聚合酶链式反应(PCR)及序列分析利用FastDNA Spin kit for soil提取试剂盒(MP Biomedicals, USA), 称取0.33 g左右冷冻干燥后的土壤样品, 提取总DNA后保存在-20 ℃冰箱中.利用针对n-damo菌pmoA功能基因的特异性引物进行巢式PCR扩增, 第1步引物选用A189F/cmo682, 第2步引物选用cmo182/cmo568(Luesken et al., 2011).PCR扩增产物的切胶纯化选用Promega Agarose Gel DNA (Promega, Madison, WI)纯化试剂盒进行, 将回收的PCR扩增产物与pGEM-Teasy载体(Promega, Madison, WI)连接后转入JM109感受态细胞(T AKARA, 大连), 然后筛选蓝白斑.随机从每个克隆文库中挑选合适数量的白色克隆子, 选用pGEM-T Easy Vector通用引物T7(5′-TAATACGACTCACTATAGGG-3′)和SP6(5′-ATTTAGGTGACACTATAGAA-3′)进行PCR扩增, 鉴定阳性克隆.将所得序列在Genbank基因数据库中进行BLAST比对并选取参比序列, 以邻接法(Neighbor-Joining)运用Mega 4.1软件构建系统发育树.
2.4 实时荧光定量PCR分析分别对n-damo菌、AOA、AOB、反硝化细菌和全细菌采用实时荧光定量PCR方法进行分析.利用引物p1F/R和p2F/R, 分别对n-damo菌的16S rRNA基因进行定量分析(Ettwig et al., 2009);利用引物archea-amoAF/R对AOA的amoA基因进行定量分析(Francis et al., 2005);利用引物amoA-1F和amoA-2R对AOB的amoA基因进行定量分析(Rotthauwe et al., 1997);利用引物cd3aF和R3cd对反硝化菌的nirS基因进行定量分析(Michotey et al., 2000; Throbäck et al., 2004);利用引物8F和Eub338-I R对全细菌16S rRNA基因进行定量分析(Juretschko et al., 1998; Amann et al., 1990).使用ABI 7300 Real-Time PCR System扩增仪(Applied Biosystems, CA, USA), 将引物与SYBR Green ExTaq酶试剂盒(T AKARA, 大连)按比例配制成20 μL反应体系, 为获得标准曲线, 将已知拷贝数的质粒DNA按10倍浓度梯度稀释, 所得的6个标准样品与土壤样品一起进行定量扩增.用7300 SDS System配套软件对结果进行分析运算, 扩增效率和可决系数R2分别大于95%和0.98, 溶解曲线为单一峰的结果方可采用.所有样品均做3次平行, 分别得出n-damo菌、AOA、AOB、反硝化细菌和全细菌的丰度值.
2.5 13C稳定同位素示踪法测定n-damo菌活性称取约120 g新鲜土壤样品和240 g原位水放入500 mL血清瓶中, 加入磁力搅拌器转子, 以200 r·min-1的转速在85-2A数显测速恒温磁力搅拌器(上海司乐仪器有限公司, 上海)上搅拌混匀, 同时曝氦气.然后, 转移5 mL混合物至12.0 mL玻璃瓶中(Labco, UK), 曝氦气30 min创造厌氧环境.预培养一段时间以消耗其中残留的NO-x和O2.每个Labco瓶中充入0.2 mL丰度为99%的13C-CH4气体(Sigma-Aldrich Co. LLC., USA), 向15个实验组Labco瓶中注入0.10 mL无O2和CO2的50 mg·L-1 NaNO2(以N计)溶液, 向15个硫酸盐对照组Labco瓶中注入0.10 mL无O2和CO2的50 mg·L-1 Na2SO4(以S计)溶液, 向15个阴性对照组Labco瓶中注入0.10 mL无O2和CO2的超纯水.混匀样品后, 将Labco瓶置于黑暗中的HZQ-7恒温振荡器(苏州威尔实验用品有限公司, 江苏)内培养, 转速为200 r·min-1, 并按一定时间点分别取样.用稳定同位素比质谱仪Delta V Advantage (Thermo Electron Corporation, Bremen, Germany)和带有ECD检测器的GC7890气相色谱仪(Agilent Technologies, USA)测得反应生成45CO2的量, 计算可得n-damo菌反应速率.
2.6 15N稳定同位素示踪法测定反硝化反应速率称取约3.3 g新鲜土壤样品放入12.0 mL玻璃瓶中(Labco, UK), 加入2颗玻璃珠以均匀混合, 向玻璃瓶中注满饱和氮气无氧原位水.将玻璃瓶在室温条件下置于QB-228旋转培养器(海门市其林贝尔仪器制造有限公司, 江苏)上培养.待去除其中的NO3-后, 分别注入一定量丰度为99.29%的15NaNO3, 进行3种不同处理:①仅加15NH4+;②同时加入15NH4+和14 NO3-;③同时加入14NH4+和15NO3-.按一定的时间点取样, 取样后注入0.2 mL浓度为7 mol·L-1的ZnCl2溶液以终止反应.在YQX-Ⅱ型厌氧操作箱(上海新苗医疗器械制造有限公司, 上海)中, 抽出2 mL完成反应的样品上清液, 导入已充满氦气的Labco玻璃瓶中, 剧烈振荡, 使玻璃瓶中水、气达到平衡.通过气相色谱-同位素比值质谱仪(Gasbench-MAT253, 外接GasBench Ⅱ多用途在线气体制备仪, Thermo Electron Corporation, Bremen, Germany), 测得29 N2和30N2的含量, 计算可得反硝化反应的速率.
2.7 硝化反应速率测定硝化速率的测定采用氯酸盐抑制法(Kurola et al., 2005).取3.0 g左右土壤样品放入50 mL离心管中, 加入20 mL磷酸盐缓冲溶液及1 mmol·L-1硫酸铵.另外, 加入终浓度为10 mg·L-1的氯酸钾, 以抑制亚硝酸盐氧化.在室温下黑暗培养24 h, 加入2 mol·L-1的氯化钾溶液5 mL, 以浸提亚硝酸盐.离心后取上清液, 用乙二胺分光光度法在540 nm处测定亚硝酸盐浓度, 计算得硝化速率.样品均设3个平行.
3 结果(Results) 3.1 北澧河小麦田土壤柱状样理化性质由于长期施肥, 土壤中含氮化合物含量很高.NH4+冬季含量为0.57~5.4 mg·kg-1, 最高值出现在20~40 cm土层;夏季含量为1.58~2.46 mg·kg-1, 最高值出现在40~60 cm土层.冬、夏季NO2-含量分别为0.02~0.07 mg·kg-1和0.02~0.09 mg·kg-1, 最高值分别出现在60~80 cm和0~20 cm土层.由于农田灌溉水中含有大量NO3-, 土壤样品中NO3-含量也很高, 冬季含量为7.11~14.47 mg·kg-1, 最高值出现在0~20 cm土层;夏季含量为3.54~15.77 mg·kg-1, 最高值出现在80~100 cm土层.土壤样品中CH4含量极低, 冬、夏季含量分别为47.32~47.49和44.87~46.30 mg·m-3(以土壤计).TC、TN和TS的最大值均出现在0~20 cm土层.土壤呈弱碱性, 总有机质含量最大值夏季出现在80~100 cm土层, 而冬季出现在0~20 cm土层.北澧河小麦田土壤柱状样(0~1 m)的主要理化性质如图 1所示.
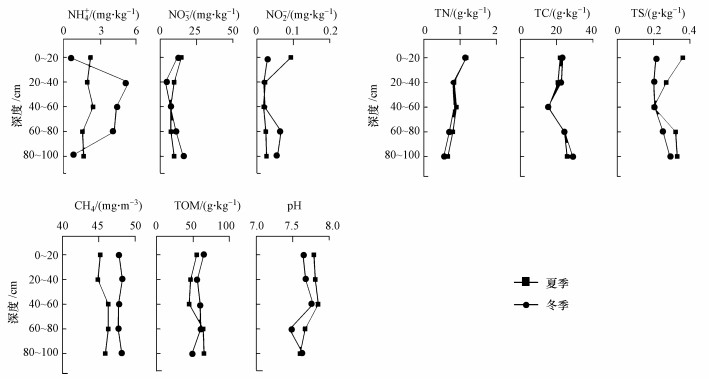 |
| 图 1 北澧河小麦田土壤柱状样品理化指标 Fig. 1 Chemical characteristics of the soil samples in Beili wheat field |
选取北澧河小麦田冬、夏土壤柱状样(0~1 m)的5个代表深度(0~20、20~40、40~60、60~80和80~100 cm), 对n-damo菌的pmoA基因通过巢式PCR进行扩增.夏季深层土壤(80~100 cm)和冬季深层土壤(60~80和80~100 cm)的DNA在第二步PCR扩增中未获得阳性结果(图 2).这一结果直接证明在旱地垂向分布中, n-damo菌更多存在于浅层土壤中.
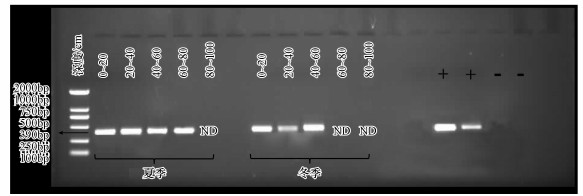 |
| 图 2 n-damo菌pmoA基因定性PCR电泳图(ND表示未检测到的样品, “+”表示阳性对照, “-”表示阴性对照) Fig. 2 Electrophoresis of the n-damo bacteria pmoA gene amplification |
为了研究旱地土壤n-damo菌的群落结构和空间变化特性, 选取0~20和40~60 cm土壤样品的PCR阳性产物构建克隆文库.冬、夏季土壤样品每层各5条序列进行分析, 通过构建系统发育树(图 3), 将所得序列与已知M. oxyfera-like菌对比分析, 结果显示, 扩增序列与已知的M. oxyfera-like菌(JQ429432)具有较近的亲缘关系.将所得序列与JQ429432进行BlAST比对, 其同源性高达90%~98%.同时, 冬、夏两季, 来自0~20 cm和40~60 cm土壤的序列完全分开,处于系统发育树不同分枝, 证明北澧河小麦田n-damo菌具有明显的空间异质性.
 |
| 图 3 n-damo菌pmoA基因的系统发育树(注:北澧河小麦田n-damo菌冬、夏季代表序列分别用“BLW”和“BLS”表示, 括号中为序列所在土壤深度;本文中的参照序列引自GenBank数据库, 括号中为参照序列在Genbank数据库中的获取号;用邻接法构建系统发育树, 分支节点上的数字表示每1000次的bootstrap分析所支持的次序, 图中标尺表示1%序列差异的分支长度.) Fig. 3 Phylogenetic tree of n-damo bacteria 16S rRNA sequences |
采用实时荧光定量PCR技术对n-damo菌的16S rRNA基因进行定量分析(图 4).夏季其丰度值为1.44×104~1.02×105 copies·g-1, 占全细菌的0.0005%~0.007%;冬季丰度值为3.66×104~2.67×105copies·g-1, 占全细菌的0.0004%~0.02%.迄今为止, 针对n-damo菌pmoA基因仍然没有更加合适的引物用以qPCR定量分析.针对其16S rRNA基因的定量引物是基于富集环境中的NC10门克隆序列设计的, 特异性还有待进一步优化.
 |
| 图 4 n-damo菌(a)、AOB (b)、反硝化细菌(c)、AOA (d)及全细菌(e)(比例小于0.01%时用“-”表示) Fig. 4 Abundance of n-damo bacteria (a), AOB (b), denitrification bacteria (c), AOA (d) and total bacteria (e) |
土壤中AOB、AOA和反硝化细菌均可为n-damo菌提供底物NO2-, 同时, 反硝化菌与n-damo菌都利用NO2-作为电子受体完成代谢过程, 互为有力竞争者.为了解其在N循环过程中的相互作用关系, 采用荧光定量PCR技术对AOA和AOB的amoA基因进行定量分析.其中, AOB冬季的丰度为1.57×102~5.84 ×106 copies·g-1(占全细菌的0.001%~0.07%);夏季为7.16×103~1.48×106 copies·g-1(占全细菌的0.0003%~0.0042%).另一方面, AOA的丰度值夏季为4.76×107~1.65×109 copies·g-1, 冬季为6.73×105~6.59×108 copies·g-1.以上结果显示, 土壤中AOA的数量显著高于AOB, AOA可能比AOB为n-damo菌提供更多的NO2-.对反硝化菌nirS基因的定量结果显示, 反硝化菌数量夏季明显高于冬季, 夏季其丰度为2.60×108~5.75×108 copies·g-1(占全细菌的1.62%~26.56%), 冬季降为2.93×106~4.75×107 copies·g-1(占全细菌的0.45%~24.42%).
3.5 北澧河小麦田n-damo菌及相关N循环微生物活性测定测定旱地土壤n-damo菌的活性, 能够量化其反应速率潜势, 并且更加准确评估地n-damo菌在土壤氮、碳循环及控制温室效应中的作用.经过26 d的培养实验, 仅在夏季表层土壤测得极低的n-damo菌反应速率(0.18 nmol ·g-1·d-1, 以CO2计), 其他土层均未检测到13CO2产生(图 5), 说明在这些土层中发生着极低的n-damo反应甚至没有发生.以上实验结果证明, 旱地土壤并不是n-damo菌适宜的生存环境.
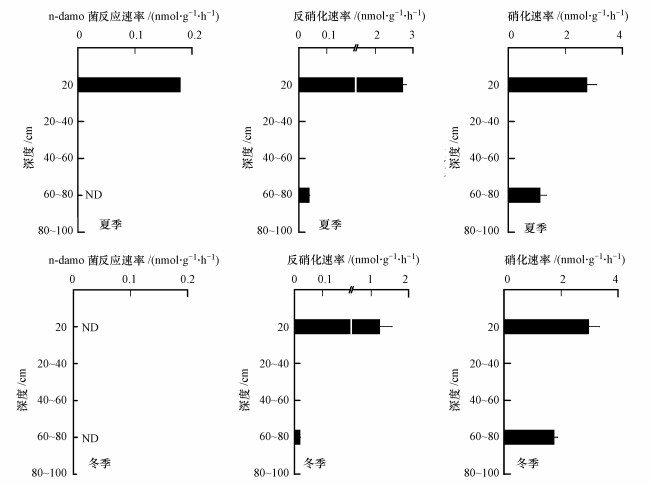 |
| 图 5 反硝化厌氧甲烷氧化、反硝化和硝化速率 Fig. 5 n-damo rate, denitrification rate and nitrification rate |
同时, 为了确定n-damo菌的NO2-底物来源, 探究不同的N循环过程对其贡献, 对硝化和反硝化反应速率进行测定.结果表明, 在所选取的0~20 cm和60~80 cm土壤中均有硝化和反硝化反应发生.其中, 冬季硝化速率为1.76~2.97 nmol·g-1·h-1(以N计, 下同), 夏季为1.11~2.76 nmol·g-1·h-1, 与此同时, 冬、夏季反硝化速率分别为0.02~1.23和0.04~2.73 nmol·g-1·h-1(以N计)(图 5).冬、夏两季, 硝化和反硝化速率均随土壤深度的增加而逐渐降低, 最高值出现0~20 cm土层.
4 讨论(Discussion)随着地球碳循环研究的不断深入, n-damo菌陆续被发现广泛分布在不同领域, 如生物反应器、富集培养基及湿地生态系统中(Ettwig et al., 2009; Hu et al., 2009; 2011; Luesken et al., 2011a; 2011b; 2012; Wu et al., 2011a, b).然而迄今为止, 针对其在旱地土壤的分布规律研究较少, 尤其是垂向分布尚未有明确的研究.本次实验研究了n-damo菌在旱地土壤的垂向分布特征, 对其底物来源进行了深入探索.结果表明, 旱地土壤n-damo菌更多地分布在浅层区域, 深层土壤未检测到其活性, 并且其群落结构具有明显的空间异质性.另外, 在浅层土壤中硝化和反硝化反应共同为n-damo菌提供底物来源, 而在深层土壤中, 硝化反应为其提供主要的底物来源.这些研究结果有利于建立更加完善的土壤碳、氮循环机制.
在本次研究的典型旱地土壤中, n-damo菌更多地存在于浅层土壤中.此前在西溪湿地的研究中也有类似的结论, 证明n-damo菌仅在表层能够检测到, 深层土壤中未发现其存在(Shen et al., 2014).然而, 该分布规律明显不同于湿地, n-damo菌仅分布于深层土壤中而在表层土壤中未检测到其存在(Wang et al., 2012).此次研究还发现, n-damo菌的丰度随着深度的增加而减少.早前在稻田土壤的研究中也发现类似规律, n-damo菌的丰度分布是随着土壤深度的增加而逐渐减低(Wang et al., 2012).与之相反的是, n-damo菌在湿地沉积物的分布却随着深度的增加而增多(Shen et al., 2014).
进一步分析发现, 多种土壤理化因素的综合作用导致了n-damo菌这一独特的分布特征.此处旱地土壤中CH4含量较低, 底物不足使得n-damo菌活性受到抑制.在检测不到n-damo菌存在的深层土壤中, AOA和AOB的数量明显减少.在冬季深层土壤中(60~80和80~100 cm), AOA的丰度仅为5.73×105~2.94×107 copies·g-1, AOB的丰度为1.57×102~2.16×105 copies·g-1, 在夏季80~100 cm土壤中, 其丰度同样较低, AOA的丰度为4.76×107 copies·g-1, AOB的丰度为7.16×103 copies·g-1.AOA和AOB无法提供充足的NO2-底物, 使n-damo菌的生长环境较为不利.不仅如此, 由于n-damo菌和反硝化细菌的代谢过程都是以NO2-作为电子受体, 两者间存在明显的竞争关系.在深层土壤中, 总碳含量(冬季24.32~25.76 mg·kg-1, 夏季28.79 mg·kg-1)和总有机质含量(冬季63.83~65.07 mg·kg-1, 夏季48.46 mg·kg-1)较高, 为反硝化细菌的生存提供了充足的能源和底物来源.反硝化菌数量在冬季深层(2.93×106~1.21×107 copies·g-1)和夏季深层(2.60×108 copies·g-1)土壤中明显偏高, 在竞争中占据优势地位, 更加不利于n-damo菌的生存.从以上结果可清晰看出, 多种土壤理化条件及微生物间的相互作用关系直接导致n-damo菌更多地分布在旱地浅层土壤中.
值得注意的是, 由于人类活动及农田施肥的影响, 表层土壤中硝酸盐含量极高(冬季为14.47 mg·kg-1, 夏季为11.70 mg·kg-1).由于硝酸盐带负电荷, 在同样带负电荷的土壤中不会被吸附, 从而不断向下迁移(Booltink, 1994), 为浅层土壤中n-damo菌的生长提供了更加有利的条件.总体看来, 在生存条件最有利的表层土壤中, n-damo菌的活性最大.随着土壤深度的增加, 反硝化菌的丰度不断增加, 在两者的竞争中逐渐占据有利地位, n-damo菌生长被抑制, 因而表层土壤以下均检测不到其活性.
比较土壤氮循环不同过程的速率发现, 在0~20 cm的土壤中, 硝化速率略高于反硝化速率, 其中, 冬季硝化速率(2.97 nmol·g-1·h-1)高于反硝化速率(1.23 nmol·g-1·h-1);夏季硝化速率为2.76 nmol·g-1·h-1, 反硝化速率与之持平(2.73 nmol·g-1·h-1), 两者共同为n-damo菌提供底物NO2-.而在60~80 cm的土壤中, 硝化速率(冬季为1.76 nmol·g-1·h-1, 夏季为1.11 nmol·g-1·h-1)显著高于反硝化速率(冬季为0.04 nmol·g-1·h-1, 夏季为0.02 nmol·g-1·h-1), n-damo菌的NO2-底物主要来源于硝化过程.
据系统发育分析显示, 来自土壤0~20 cm和40~60 cm的n-damo菌序列完全分开, 处于发育树不同分枝, 证明旱地土壤中n-damo菌的群落结构空间异质性较强, 随土壤深度的变化不断改变.类似结论在之前的研究中也有报道, 表层沉积物的M.oxyfera-like菌序列与其底层序列完全不同, 无法聚集在系统发育树同一分枝(Zhu et al., 2015).同样, 属于NC10门A组类的n-damo菌序列来自土壤深层, 而相似于B组类的序列来自土壤浅层(Shen et al., 2015).
5 结论(Conclusions)在北澧河典型旱地农田土壤中, n-damo菌仅分布于浅层, 其丰度随着土壤深度的增加而降低.并且, n-damo菌活性较低, 仅在夏季表层土壤中能检测到较低的活性, 表层以下均检测不到其活性.较低的丰度及活性显示旱地土壤并不是n-damo菌适宜的生存环境, 同时也说明n-damo菌对旱地农田土壤碳循环的贡献可能不大.为建立更加完善的土壤碳、氮循环机制, 关于旱地土壤中n-damo菌的特性还需要更加深入的研究.
| [${referVo.labelOrder}] | Amann R I, Binder B J, Olson R J, et al. 1990. Combination of 16S rRNA-targeted oligonucleotide probes with flow cytometry for analyzing mixed microbial populations[J]. Appl Environ Microbiol, 56(6) : 1919–1925. |
| [${referVo.labelOrder}] | 鲍士旦. 2000. 土壤农化分析[M]. 北京: 中国农业出版社. |
| [${referVo.labelOrder}] | Booltink H W G. 1994. Field monitoring of nitrate leaching and water flow in a structured clay soil[J]. Agriculture Ecosystems & Environment, 52(2/3) : 251–261. |
| [${referVo.labelOrder}] | Conrad R. 2009. The global methane cycle:recent advances in understanding the microbial processes involved[J]. Environmental Microbiology Reports, 1 : 285–292. DOI:10.1111/j.1758-2229.2009.00038.x |
| [${referVo.labelOrder}] | Ettwig K F, Shima S, van de Pas-Schoonen K T, et al. 2008. Denitrifying bacteria anaerobically oxidize methane in the absence of Archaea[J]. Environ Microbiol, 10(11) : 3164–3173. DOI:10.1111/emi.2008.10.issue-11 |
| [${referVo.labelOrder}] | Ettwig K F, Alen T V, Pas-Schoonen K T V D, et al. 2009. Enrichment and molecular detection of denitrifying methanotrophic bacteria of the NC10 phylum[J]. Applied & Environmental Microbiology, 75(11) : 3656–3562. |
| [${referVo.labelOrder}] | Ettwig K F, Butler M K, Le P D, et al. 2010. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria[J]. Nature, 464(7288) : 543–548. DOI:10.1038/nature08883 |
| [${referVo.labelOrder}] | Francis C, Roberts K, Beman J, et al. 2005. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences, 102(41) : 14683–14688. DOI:10.1073/pnas.0506625102 |
| [${referVo.labelOrder}] | Haroon M F, Hu S, Shi Y, et al. 2013. Anaerobic oxidation of methane coupled to nitrate reduction in a novel archaeal lineage[J]. Nature, 500(7464) : 567–570. DOI:10.1038/nature12375 |
| [${referVo.labelOrder}] | Hu S, Zeng R J, Burow L C, et al. 2009. Enrichment of denitrifying anaerobic methane oxidizing microorganisms[J]. Environmental Microbiology Reports, 1(5) : 377–384. DOI:10.1111/j.1758-2229.2009.00083.x |
| [${referVo.labelOrder}] | Hu B L, Rush D, van der B E, et al. 2011. New anaerobic, ammonium-oxidizing community enriched from peat soil[J]. Applied and Environmental Microbiology, 77(3) : 966–971. DOI:10.1128/AEM.02402-10 |
| [${referVo.labelOrder}] | Juretschko S, Timmermann G, Schmid M, et al. 1998. Combined molecular and conventional analyses of nitrifying bacterium diversity in activated sludge:Nitrosococcus mobilis and Nitrospira-like bacteria as dominant populations[J]. Appl Environ Microbiol, 64 : 3042–3051. |
| [${referVo.labelOrder}] | Knittel K, Boetius A. 2009. Anaerobic oxidation of methane:progress with an unknown process[J]. Annual Review of Microbiology, 63 : 311–334. DOI:10.1146/annurev.micro.61.080706.093130 |
| [${referVo.labelOrder}] | Kurola J, Salkinoja-Salonen M, Aarnio T, et al. 2005. Activity, diversity and population size of ammonia-oxidising bacteria in oil-contaminated landfarming soil[J]. FEMS Microbiology Letters, 250(1) : 33–38. DOI:10.1016/j.femsle.2005.06.057 |
| [${referVo.labelOrder}] | Luesken F A, Sánchez J, van A T A, et al. 2011a. Simultaneous nitrite-dependent anaerobic methane and ammonium oxidation[J]. Appl Environ Microbiol, 77(19) : 6802–6807. DOI:10.1128/AEM.05539-11 |
| [${referVo.labelOrder}] | Luesken F A, Alen T A V, Biezen E V D, et al. 2011b. Diversity and enrichment of nitrite-dependent anaerobic methane oxidiz-ing bacteria from wastewater sludge[J]. Appl Microbiol Biotechnol, 92(4) : 845–854. DOI:10.1007/s00253-011-3361-9 |
| [${referVo.labelOrder}] | Luesken F A, Zhu B, van A T A, et al. 2011c. pmoA primers for detection of anaerobic methanotrophs[J]. [J].Applied & Environmental Microbiology, 77(11) : 3877–80. |
| [${referVo.labelOrder}] | Luesken F A, Wu M, Op den C H J M, et al. 2012. Effect of oxygen on the anaerobic methanotroph 'Candidatus Methylomirabilis oxyfera':kinetic and transcriptional analysis[J]. Environmental Microbiology, 14 : 1024–1034. DOI:10.1111/emi.2012.14.issue-4 |
| [${referVo.labelOrder}] | McIlvin M R, Altabet M A. 2005. Chemical conversion of nitrate and nitrite to nitrous oxide for nitrogen and oxygen isotopic analysis in freshwater and seawater[J]. Anal Chem, 77(17) : 5589–5595. DOI:10.1021/ac050528s |
| [${referVo.labelOrder}] | Michotey V, Mejean V, Bonin P. 2000. Comparison of methods for quantification of cytochrome cd1-denitrifying bacteria in marine samples[J]. Appl Environ Microbiol, 66(4) : 1564–1571. DOI:10.1128/AEM.66.4.1564-1571.2000 |
| [${referVo.labelOrder}] | Raghoebarsing A A, Pol A, Kt P S, et al. 2006. A microbial consortium couples anaerobic methane oxidation to denitrification[J]. Nature, 440(7086) : 918–921. DOI:10.1038/nature04617 |
| [${referVo.labelOrder}] | Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63(12) : 4704–4712. |
| [${referVo.labelOrder}] | Shen L D, Wu H S, Gao Z Q. 2015. Distribution and environmental significance of nitrite-dependent anaerobic methane-oxidising bacteria in natural ecosystems[J]. Applied Microbiology and Biotechnology, 99(1) : 133–142. DOI:10.1007/s00253-014-6200-y |
| [${referVo.labelOrder}] | Shen L D, Liu S, Zhu Q, et al. 2013. Distribution and diversity of nitrite-dependent anaerobic methane-oxidising bacteria in the sediments of the Qiantang River[J]. Microbial Ecology, 67(2) : 341–349. |
| [${referVo.labelOrder}] | Solomon S D, Qin D, Manning M, et al. 2007. Climate Change 2007:The Physical Science Basis. Working Group Ⅰ Contribution to the Fourth Assessment Report of the IPCC[M]. New York: Cambridge University Press: 1–21. |
| [${referVo.labelOrder}] | Strous M, Jetten M S M. 2004. Anaerobic oxidation of methane and ammonium[J]. Annual Review of Microbiology, 58(1) : 99–117. DOI:10.1146/annurev.micro.58.030603.123605 |
| [${referVo.labelOrder}] | Throbäck I N, Enwall K, Åsa J, et al. 2004. Reassessing PCR primers targeting nirS, nirK, and nosZ, genes for community surveys of denitrifying bacteria with DGGE[J]. Fems Microbiology Ecology, 49(3) : 401–417. DOI:10.1016/j.femsec.2004.04.011 |
| [${referVo.labelOrder}] | Wang Y, Zhu G, Harhangi H R, et al. 2012. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil[J]. Fems Microbiology Letters, 336(2) : 79–88. DOI:10.1111/fml.2012.336.issue-2 |
| [${referVo.labelOrder}] | Wu M L, De V S, van A T A, et al. 2010. Physiological role of the respiratory quinol oxidase in the anaerobic nitrite-reducing methanotroph 'Candidatus Methylomirabilis oxyfera'[J]. Microbiology, 157(Pt 3) : 890–898. |
| [${referVo.labelOrder}] | Wu M L, Ettwig K F, Jetten M S, et al. 2011. A new intra-aerobic metabolism in the nitrite-dependent anaerobic methane-oxidizing bacterium Candidatus 'Methylomirabilis oxyfera'[J]. Biochemical Society Transactions, 39(1) : 243–248. DOI:10.1042/BST0390243 |
| [${referVo.labelOrder}] | 张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理, 2007, 30(3) : 455–459. |
| [${referVo.labelOrder}] | Zhu G, Zhou L, Wang Y, et al. 2015. Biogeographical distribution of denitrifying anaerobic methane oxidizing bacteria in Chinese wetland ecosystems[J]. Environmental Microbiology Reports, 7(1) : 128–138. DOI:10.1111/emi4.2015.7.issue-1 |
 2017, Vol. 37
2017, Vol. 37


