2. 成都市固体废弃物卫生处置场, 成都 610041
2. Chengdu Municipal Solid Waste Disposal Site, Chengdu 610041
就地回灌是目前国内外垃圾填埋场渗滤液的主要处理手段之一 (蒋宝军等,2006;王晓东,2013),因此,填埋场的上层区域内既有来自于下层厌氧环境中逸出的甲烷,又有来自于回灌渗滤液中的硝酸盐/亚硝酸盐.同时,填埋场上层处于干湿交替的微氧环境,这个区域就有可能发生微氧条件下的甲烷氧化反硝化过程.对此过程的研究不仅对完善全球碳/氮循环具有重要理论意义,同时对于原位去除垃圾填埋场中的温室气体甲烷和渗滤液中的硝氮/亚硝氮具有重要现实意义.
甲烷好氧氧化偶联反硝化 (Aerobic Methane Oxidation Coupled to Denitrification, AME-D) 过程于20世纪70年代最初由Harremoes和Christensen在一硝化后的城市污水中发现,在甲烷存在的条件下存在硝酸盐减少的现象 (Harremoes et al., 1971),随后Davies分离出在反硝化过程中能利用甲烷及其它有机物作为电子供体的微生物 (Davies, 1973).Rhee和Fuhs于1978年发现好氧条件下甲烷氧化伴随着反硝化现象,并首次证实该过程由甲烷氧化菌和反硝化菌联合作用来完成,即反硝化菌利用甲烷氧化过程中产生的有机物来完成反硝化 (Rhee et al., 1978).
相比AME-D,甲烷厌氧氧化偶联反硝化 (Anaerobic Methane Oxidation Coupled to Denitrification, ANME-D) 直到2006年才被人们所发现.Raghoebarsing等 (2006)以淡水沟渠为接种物,在序批式反应器中经过16个月的富集培养,获得了ANME-D的培养物,该富集物由80%的NC10门细菌和20%的甲烷厌氧氧化古菌组成,推测该过程是由ANME古菌通过逆产甲烷途径将电子转移给反硝化细菌.但随着更长时间的富集培养,发现古菌数量逐渐减少直至消失,因此,推测该过程是由一种NC10门细菌M.oxyfera通过独特代谢途径独立完成 (Ettwig et al., 2008),随后研究人员从基因组、转录组、蛋白组均证实出该细菌独特的内产氧代谢途径 (Hanson et al., 2015),即亚硝酸盐在亚硝酸盐还原酶作用下生成一氧化氮,一氧化氮在一氧化氮歧化酶催化下生成氮气和氧气,利用生成的氧气将甲烷依次氧化为甲醇、甲醛和甲酸,最后转化为二氧化碳.而对于硝酸盐的还原则是由甲烷厌氧氧化古菌通过逆产甲烷途径将其还原为亚硝酸盐 (Haroon et al., 2013).虽然不同学者对好氧条件下的AME-D (Modin et al., 2007; Zhu et al., 2016) 和厌氧条件下的ANME-D都有较多的研究 (Shen et al., 2015; 吴伟祥等,2014),但关于在微氧条件下二者的存在情况还很少有报道.本研究尝试在微氧条件下以硝酸盐氮/亚硝酸盐氮为电子受体,甲烷为唯一有机碳源,对反硝化型甲烷氧化微生物进行富集培养,借助现代分子生物学技术分析富集培养物中AME-D和ANME-D型功能微生物,最后通过二代测序技术分析主要功能微生物菌群,以期探清微氧条件下以甲烷为碳源的反硝化是AME-D过程还是ANME-D过程.
2 材料与方法 (Materials and methods) 2.1 接种物接种物取自成都市第四污水处理厂厌氧活性污泥,VSS为16 g·L-1,pH为5.93.
2.2 培养基参考Ettwig等 (2008)的无机培养基,成分为 (g·L-1):KHCO3 0.5~1.0,KH2PO4 0.05,CaCl2 0.23,MgSO4·7H2O 0.2,NaNO30.1~0.2,NaNO2 0.2~0.4,酸性微量元素溶液0.5 mL,碱性微量元素溶液0.2 mL.每升酸性微量元素溶液含FeSO4·7H2O 2.085 g、ZnSO4·7H2O 0.068 g、CoCl2·6H2O 0.12 g、MnCl2·4H2O 0.5 g、CuSO4 0.32 g、NiCl2·6H2O 0.095 g、H3BO3 0.014 g;每升碱性微量元素溶液含SeO2 0.067 g、Na2WO4·2H2O 0.05 g、Na2MoO4 0.242 g.
2.3 富集培养装置采用自行设计的序批式反应器 (Sequencing Batch Reactor, SBR) 进行实验.如图 1所示,反应器总体积为7 L,添加培养基至5 L,添加空心球填料以利于微生物的附着生长.气袋体积为40 L,集气瓶和收集瓶均为1 L,总气相体积为44 L,气相为混合气体 (CH4 30%~35%,O2 1%~5%,N2 60%~69%),富集培养启动时,采用上述混合气体吹脱置换系统中的空气.通过增压泵的作用不断进行循环曝气,每8 h曝气2 h,每周取样2~3次对理化指标进行测定,当培养基内硝酸盐/亚硝酸盐和气体甲烷消耗完毕时及时补充,每月置换1 L新鲜培养基.富集培养温度为25~30 ℃.
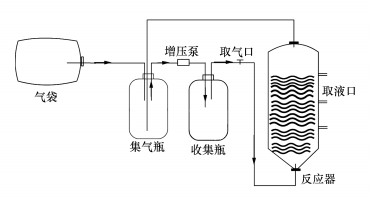 |
| 图 1 序批式反应器装置示意图 Fig. 1 Schematic diagram of SBR system |
控制反应器内的NO3--N和NO2--N浓度分别为15~25 mg·L-1和35~45 mg·L-1,当低于此值时补加NaNO2和NaNO3溶液,气袋里添加混合气体 (CH4 30%~35%,O2 1%~5%,N2 60%~69%),整个测试阶段未进行补充,每天早上取样进行分析.
2.5 理化指标分析方法亚硝酸盐分析采用N-(1-萘基)-乙二胺光度法 (GB7493-87),硝酸盐分析采用紫外可见分光光度法;气体CH4、CO2、O2和N2浓度由Agilen 7890A型气相色谱仪分析,配TCD检测器,PORAPAK Q 80/100 SS和MOLSIEVE 13×80/100 SS填充柱,载气为Ar,流速为30 mL·min-1,进样口、色谱柱和检测器温度分别为200、80和200 ℃.
2.6 分子生物学分析DNA提取与PCR扩增:定期取培养物于-20 ℃冻存,采用Power Soil DNA Kit (Mo Bio Laboratories, Carlsbad, CA, USA) 进行DNA抽提,按照试剂盒说明书进行操作,抽提结果用1%的琼脂糖凝胶电泳检测.
采用巢式PCR分别对富集培养物中的AME-D和ANME-D功能菌群的pmoA基因进行扩增.本实验所使用的引物 (表 1) 及PCR反应条件分别参照已报道的文献 (Luesken et al., 2011a; 2011b;刘晶静, 2012).PCR反应Premix Ex TaqTM(包括Mg2+、dNTPs、缓冲液) 购买自TIANGEN生物股份有限公司.用1%的琼糖凝胶电泳对PCR产物进行检测.
| 表 1 本试验所用的引物 Table 1 Primer used in this study |
克隆与测序:将含有目的条带的PCR产物连接到pEASY-T1载体上,按照试剂盒上的说明书进行操作.用菌液PCR验证阳性克隆, 将阳性克隆送至苏州金唯智生物科技有限公司测序.
16S rRNA和pmoA基因系统发育分析:获得的序列用MEGA 6.0软件进行编辑,用BLAST在GenBank中搜索相似序列,对这些序列用Clustal X2软件进行完全比对,最后采用MEGA 6.0的邻接 (Neighbor-Joining) 法构建系统发育树.16S rRNA以3%的序列差异作为一个OUT,pmoA以5%的序列差异作为一个OTU (朱群等, 2013)
高通量测序:将富集培养物的DNA送至中科院成都生物研究所高通量测序平台,采用Illumina Miseq测序仪进行高通量测序.
3 结果 (Results) 3.1 SBR富集培养图 2a为SBR运行中硝氮和亚硝氮还原速率的变化,硝氮和亚硝氮还原速率波动较大,这可能是由于气液传质不均衡造成的.总体而言,亚硝氮的还原速率高于硝氮的还原速率.图 2b为SBR运行中气体CH4和CO2含量变化,可以看出,每一阶段内CH4含量都呈现明显的下降趋势,而CO2始终在1%~8%范围内波动变化.
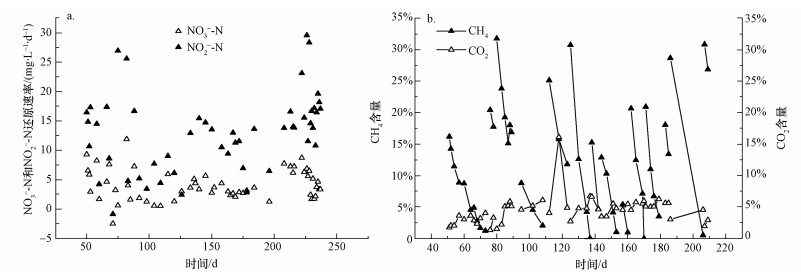 |
| 图 2 SBR运行结果 (a.硝氮、亚硝氮还原速率;b.甲烷、二氧化碳气体含量变化) Fig. 2 The running resutl of SBR (a. the reduction rates of nitrate and nitrite nitrogen, b. the mass profiles of methane and carbon dioxide) |
图 3为反硝化活性测试结果.从图中可以看出,亚硝酸氮的消耗速率明显高于硝酸氮,硝态氮平均消耗速率为3.69 mg·L-1·d-1 (以N计),亚硝态氮平均消耗速率为18.04 mg·L-1·d-1 (以N计) (图 3a).而从图 3b可以看出,甲烷含量表现出明显的下降趋势,由初始的29.44%下降为3.01%,而相应的氮气含量则表现出明显的上升趋势,由初始的65.79%上升为92.41%,而O2含量始终维持在1.00%~3.00%这样一个微氧环境内,CO2含量也维持在2.00%左右的较低水平.从反硝化活性测试结果可以看出,在微氧条件下该富集培养物具有明显的甲烷氧化偶联反硝化活性.
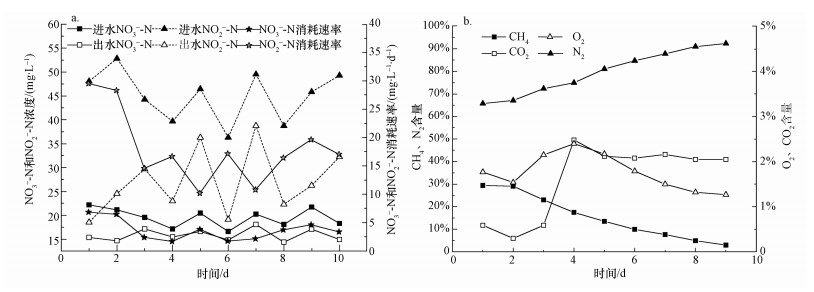 |
| 图 3 活性测试结果 (a.硝氮、亚硝氮浓度变化;b.气体含量变化) Fig. 3 Biological activity test results (a. nitrogen mass profiles in nitrate and nitrite, b. mass profiles of gas) |
甲烷单加氧酶pmoA是好氧甲烷氧化菌氧化甲烷过程的关键酶,已成为甲烷氧化菌的标志性基因,通过PCR成功扩增出该基因特异性序列,14条有郊序列彼此之间的相似度为92.18%~100.00%, 归为3个OTUs (图 4).分别与水稻田土 (Qiu et al., 2008)、水稻田根际土 (Ferrando et al., 2009)、一株甲烷氧化菌 (Hoefman et al., 2014) 的pmoA序列相似性最高,相似度分别为99%、96%、96%.
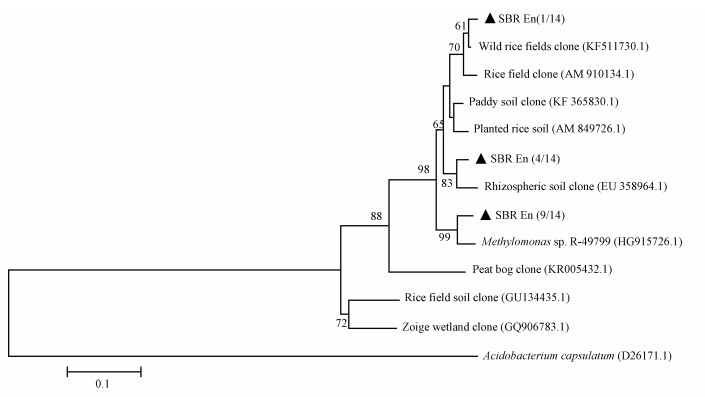 |
| 图 4 SBR富集培养物AME-D菌的pmoA基因系统发育树 Fig. 4 Phylogenitic tree of AME-D bacteria-relatedpmoA gene fragments in the clone libraries of samples from SBR |
在ANME-D过程中起主要作用的功能微生物为隶属于NC10门细菌的M.oxyfera,虽然其甲烷氧化途径也是通过独特的内产氧好氧甲烷氧化途径,但其pmoA基因序列不同于传统的好氧甲烷氧化菌的pmoA序列.通过巢式PCR成功地在富集培养物中扩增出了ANME-D菌的pmoA基因的特异性条带,并对PCR产物进行克隆测序,对20条有效序列处理后与NCBI中已知部分ANME-D菌的pmoA序列共同构建系统发育树 (图 5).比对后发现所获得的pmoA序列彼此之间的相似度较高,达到96%以上,归为1个OTU,与已知M.oxyfera的pmoA序列相似度较高,为96%左右.
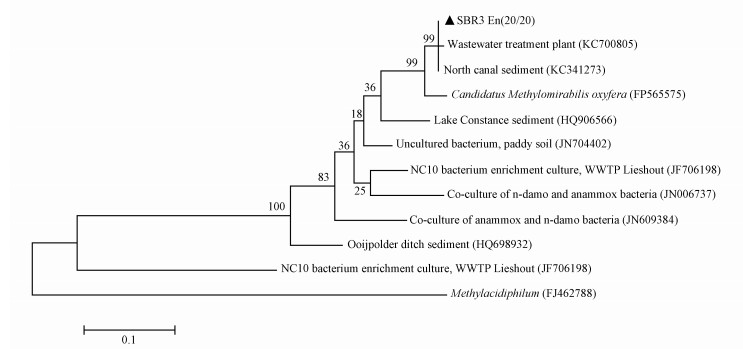 |
| 图 5 SBR富集培养物ANME-D菌的pmoA系统发育树 Fig. 5 Phylogenitic tree of ANME-D bacteria-related pmoA gene fragments in the clone libraries of samples from SBR |
对富集培养后微生物 (细菌) 进行高通量测序,结果表明,在门水平上 (图 6a),占优势的菌群是变形杆菌门 (Proteobacteria)、拟杆菌门 (Bacteroidetes)、绿菌门 (Chlorobi) 和绿弯菌门 (Chloroflexi),分别占50.10%、19.92%、9.75%和8.43%;而在ANME-D过程中起主要作用的NC10门细菌相对含量确极为低下 (小于0.1%,归为others).在科水平上 (图 6b),以β-变形菌纲 (Betaproteobacteria) 的嗜甲基菌科 (Methylophilaceae) 和γ-变形菌纲 (Gammaproteobacteria) 的甲基球菌科 (Methylococcaceae) 占有优势,相对含量分别为16.21%和23.20%, 其余还有厌氧绳菌科 (Anaerolinacea,2.84%)、嗜氢菌科 (Hydrogenophilaceae,1.09%)、腐螺旋菌科 (Saprospiraceae,1.29%) 等.在属水平上 (图 6c),甲基球菌科 (Methylococcaceae) 中的甲基单胞菌属 (Methylomonas) 占绝对优势,相对含量为21.86%.
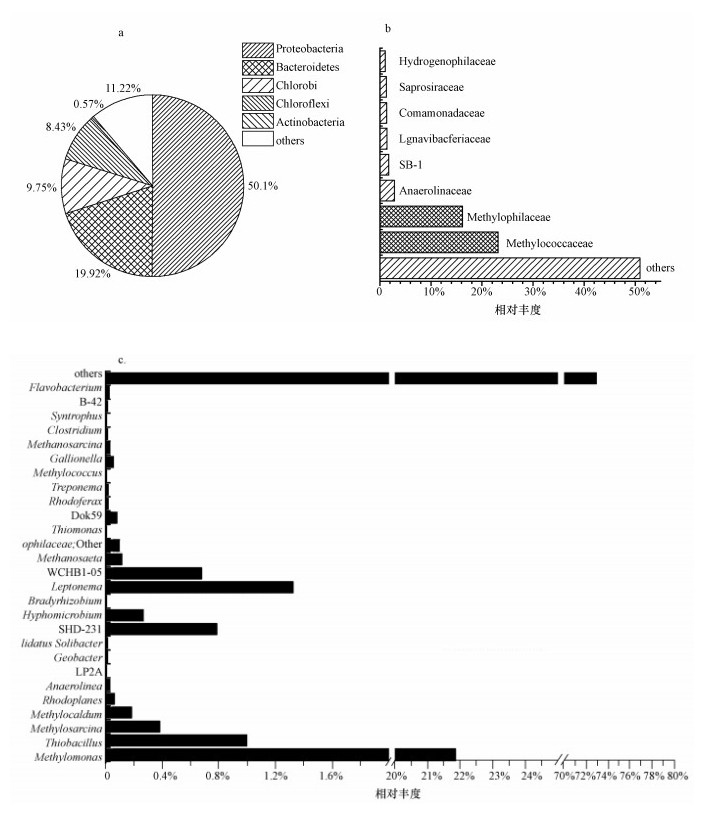 |
| 图 6 SBR中细菌群落结构 (a.门水平,b.科水平,c.属水平) Fig. 6 Bacterial distribution of the SBR cultures (a. at the phylum level, b. at the family level, c. at the genus level) |
污水处理厂厌氧消化池在有机物的分解过程中会产生大量甲烷气体,同时污水中也含有丰富的硝酸盐和亚硝酸盐,因此,很适合甲烷氧化偶联反硝化型功能微生物的生长,Hu和Kampman等都曾以活性污泥来作为ANME-D的接种物进行富集培养 (Hu et al., 2009; Kampman et al., 2014),和前人研究结果一致,本研究在厌氧活性污泥中不仅检测到ANME-D功能微生物的存在,同时也检测到AME-D功能微生物的存在,进一步证实厌氧活性污泥适合作为甲烷氧化偶联反硝化型功能微生物富集培养的接种物.虽然AME-D和ANME-D过程都需要氧气的参与,但在微氧条件下经过200多天的富集培养,ANME-D功能微生物NC10门细菌的含量仍旧极其低下,表明外界氧气的存在会对其产生抑制作用,这与Luesken等 (2012)的研究结果一致,当环境中O2含量为2%时即可对M.oxyfera的多数功能基因产生抑制作用.在科水平上,优势菌群为β-变形菌纲 (Betaproteobacteria) 的嗜甲基菌科 (Methylophilaceae) 和γ-变形菌纲 (Gammaproteobacteria) 的甲基球菌科 (Methylococcaceae),同刘晶静 (2012)的研究结果一致,2种微生物分别在AME-D过程中的好氧反硝化和甲烷好氧氧化过程中起主要作用,表明微氧条件下反应器内硝酸盐/亚硝酸盐的还原主要是AME-D过程在起主要作用.
整个富集培养过程中亚硝氮的去除速率始终要高于硝氮,从热动力学角度来看 (式 (1)、(2)),亚硝酸氮的还原更容易进行 (Raghoebarsing et al., 2006).
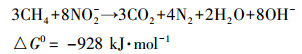
|
(1) |
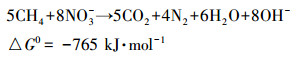
|
(2) |
CO2一直维持在较低水平,主要是因为CO2在水中的溶解度较高,且能够通过式 (3)~(5) 与反硝化过程中产生的碱度发生中和反应,导致气体中CO2含量进一步下降.
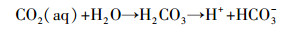
|
(3) |
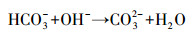
|
(4) |
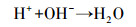
|
(5) |
本研究虽然获得了具有甲烷氧化偶联反硝化功能的富集培养物,但其反硝化速率始终较低,未来应继续优化温度、pH、溶解氧等条件以找出最佳的甲烷氧化偶联反硝化条件,从而将甲烷氧化偶联反硝化技术用于垃圾填埋场的渗滤液回灌处理.为深入了解微氧条件下甲烷氧化偶联反硝化的过程机理,明确甲烷氧化偶联反硝化过程中的中间体有机物,后期实验还应对溶液里的有机物质进行测定.
5 结论 (Conclusions)通过分子生物学手段成功在污水处理厂厌氧活性污泥中检测到AME-D菌的pmoA序列和ANME-D菌的pmoA序列,证明了厌氧活性污泥中有AME-D和ANME-D现象的发生.微氧条件更有利于AME-D过程的发生, 且亚硝氮的去除速率高于硝氮的去除速率,但微氧条件会对ANME-D过程产生抑制.
| [${referVo.labelOrder}] | Costa C, Stams A J M, Dijkema C, et al. 2000. Denitrification with methane as electron donor in oxygen-limited bioreactor[J]. Appl Microbiol Biotechnol, 53(6) : 754–762. DOI:10.1007/s002530000337 |
| [${referVo.labelOrder}] | Costello A M, Lidstrom M E. 1999. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments[J]. Applied and Environmental Microbiology, 65(11) : 5066–5074. |
| [${referVo.labelOrder}] | Davies T R. 1973. Isolation of bacteria capable of utilizing methane as a hydrogen donor in process of denitrification[J]. Water Research, 7(4) : 575–579. DOI:10.1016/0043-1354(73)90056-0 |
| [${referVo.labelOrder}] | Eisentraeger A, Klag P, Vansbotter B, et al. 2001. Denitrification of groundwater with methane as sole hydrogen donor[J]. Water Research, 35(9) : 2261–2267. DOI:10.1016/S0043-1354(00)00516-9 |
| [${referVo.labelOrder}] | Ettwig K F, Shima S, van de Pas-Schoonen K T, et al. 2008. Denitrifying bacteria anaerobically oxidize methane in the absence of Archaea[J]. Environmental Microbiology, 10(11) : 3164–3173. DOI:10.1111/emi.2008.10.issue-11 |
| [${referVo.labelOrder}] | Ferrando L, Tarlera S. 2009. Activity and diversity of methanotrophs in the soil-water interface and rhizospheric soil from a flooded temperate rice field[J]. Journal of Applied Microbiology, 106(1) : 306–316. DOI:10.1111/jam.2008.106.issue-1 |
| [${referVo.labelOrder}] | Hanson B T, Madsen E L. 2015. In situ expression of nitrite-dependent anaerobic methane oxidation proteins by Candidatus Methylomirabilis oxyfera co-occurring with expressed anammox proteins in a contaminated aquifer[J]. Environmental Microbiology Reports, 7(2) : 252–264. DOI:10.1111/1758-2229.12239 |
| [${referVo.labelOrder}] | Harremoes P H C M. 1971. Denitrification with methane (Denitrifik ation med methan)[J]. Vand(1) : 7–11. |
| [${referVo.labelOrder}] | Haroon M F, Hu S, Shi Y, et al. 2013. Anaerobic oxidation of methane coupled to nitrate reduction in a novel archaeal lineage[J]. Nature, 500(7464) : 567–570. DOI:10.1038/nature12375 |
| [${referVo.labelOrder}] | Hoefman S, van der Ha D, Boon N, et al. 2014. Niche differentiation in nitrogen metabolism among methanotrophs within an operational taxonomic unit[J]. Bmc Microbiology, 14(1) : 1. DOI:10.1186/1471-2180-14-1 |
| [${referVo.labelOrder}] | Hu S, Zeng R J, Burow L C, et al. 2009. Enrichment of denitrifying anaerobic methane oxidizing microorganisms[J]. Environmental Microbiology Reports, 1(5) : 377–384. DOI:10.1111/j.1758-2229.2009.00083.x |
| [${referVo.labelOrder}] | 蒋宝军, 李俊生, 杨威, 等. 2006. 垃圾渗滤液反渗透浓缩液回灌处理中试研究[J]. 哈尔滨商业大学学报 (自然科学版), 2006, 22(6) : 36–40. |
| [${referVo.labelOrder}] | Kampman C, Temmink H, Hendrickx T L G, et al. 2014. Enrichment of denitrifying methanotrophic bacteria from municipal wastewater sludge in a membrane bioreactor at 20 degrees C[J]. Journal of Hazardous Materials, 274 : 428–435. DOI:10.1016/j.jhazmat.2014.04.031 |
| [${referVo.labelOrder}] | Luesken F A, van Alen T A, van der Biezen E, et al. 2011. Diversity and enrichment of nitrite-dependent anaerobic methane oxidizing bacteria from wastewater sludge[J]. Applied Microbiology and Biotechnology, 92(4) : 845–854. DOI:10.1007/s00253-011-3361-9 |
| [${referVo.labelOrder}] | 刘晶静. 2012. 基于渗滤液沉积物的甲烷氧化反硝化耦合微生物学机理研究[D]. 杭州: 浙江大学 |
| [${referVo.labelOrder}] | Luesken F A, Wu M L, den Camp H J M O, et al. 2012. Effect of oxygen on the anaerobic methanotroph 'Candidatus Methylomirabilis oxyfera':kinetic and transcriptional analysis[J]. Environmental Microbiology, 14(4) : 1024–1034. DOI:10.1111/emi.2012.14.issue-4 |
| [${referVo.labelOrder}] | Luesken F A, Zhu B, van Alen T A, et al. 2011. pmoA primers for detection of anaerobic methanotrophs[J]. Applied and Environmental Microbiology, 77(11) : 3877–3880. DOI:10.1128/AEM.02960-10 |
| [${referVo.labelOrder}] | Modin O, Fukushi K, Yamamoto K. 2007. Denitrification with methane as external carbon source[J]. Water Research, 41(12) : 2726–2738. DOI:10.1016/j.watres.2007.02.053 |
| [${referVo.labelOrder}] | Nesterov A I, Ivanova T I, Il'chenko V Y, et al. 1998. Protein and polysaccharide composition during growth of Methylomonas methanica under chemostat condition[J]. Sov Biotechnol, 5 : 18–22. |
| [${referVo.labelOrder}] | Qiu Q, Noll M, Abraham W R, et al. 2008. Applying stable isotope probing of phospholipid fatty acids and rRNA in a Chinese rice field to study activity and composition of the methanotrophic bacterial communities in situ[J]. Isme Journal, 2(6) : 602–614. DOI:10.1038/ismej.2008.34 |
| [${referVo.labelOrder}] | Raghoebarsing A A, Pol A, van de Pas Schoonen K T, et al. 2006. A microbial consortium couples anaerobic methane oxidation to denitrification[J]. Nature, 440(7086) : 918–921. DOI:10.1038/nature04617 |
| [${referVo.labelOrder}] | Rhee G Y, Fuhs G W. 1978. Wastewater denitrification with one-carbon compounds as energy-source[J]. Journal Water Pollution Control Federation, 50(9) : 2111–2119. |
| [${referVo.labelOrder}] | 王晓东. 2013. 城市生活垃圾渗滤液-浓缩液厌氧/好氧回灌处理技术效能研究[D]. 长春: 吉林建筑大学 |
| [${referVo.labelOrder}] | 袁梦冬, 朱静, 吴伟祥. 2014. 微生物厌氧甲烷氧化反硝化研究进展[J]. 微生物学报, 2014, 54(2) : 129–138. |
| [${referVo.labelOrder}] | Shen L D, He Z F, Wu H S, et al. 2015. Nitrite-dependent anaerobic methane-oxidising bacteria:Unique microorganisms with special properties[J]. Current Microbiology, 70(4) : 562–570. DOI:10.1007/s00284-014-0762-x |
| [${referVo.labelOrder}] | Zhu J, Wang Q, Yuan M, et al. 2016. Microbiology and potential applications of aerobic methane oxidation coupled to denitrification (AME-D) process:A review[J]. Water Research, 90 : 203–215. DOI:10.1016/j.watres.2015.12.020 |
| [${referVo.labelOrder}] | 朱群, 沈李东, 胡宝兰, 等. 2013. 西湖底泥中的反硝化型甲烷厌氧氧化菌的分子生物学检测[J]. 环境科学学报, 2013, 33(5) : 1321–1325. |
 2017, Vol. 37
2017, Vol. 37


