大量氨氮排入水体会引起水体富营养化,造成水体发黑变臭.目前,氨氮已经成为污染物总量控制的约束性指标,因此, 污水处理过程中氮素的去除越来越受到人们的重视.然而传统脱氮技术存在能耗物耗高、工艺流程较长,外加大量有机碳源等缺点,所以开发低能耗物耗的脱氮技术成为水污染治理领域研究的热点.
厌氧氨氧化 (ANAMMOX) 菌的发现使污水自养脱氮成为可能,1977年,Broda根据热力学反应自由能计算 (Broda,1997),推测出自然界中可能存在某种自养微生物将NH4+氧化成N2.1994年,Mulder等在荷兰Delft大学运行污水脱氮流化床工艺时发现ANAMMOX现象 (Van de Graaf et al., 1995).1997年,他又通过15N标记实验发现,ANAMMOX菌在厌氧或缺氧条件下,发生ANAMMOX反应的电子供体和受体分别是NH4+和NO2-(Van de Graaf et al., 1997),随后,Strous等通过大量实验认为ANAMMOX菌在厌氧条件下将NH4+和NO2-按式 (1) 进行脱氮反应,同时有少许NO3-的生成 (Strous et al., 1998).
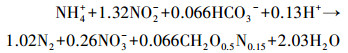
|
(1) |
目前从遍布污/废水处理厂到北极冰盖的许多生态系统中都发现了ANAMMOX菌,如中国的华北平原白洋淀沉积物 (王衫允等,2012)、南方稻田 (王雨等,2013) 及极寒和贫营养的天山天池 (姜博等,2015) 等自然生境中,以及人工废水处理系统和海洋生态系统中等 (Third et al.,2005;Rysgaard et al., 2004).这些例子不仅表明了ANAMMOX过程在海洋生态系统中的重要作用 (对于海洋生态系统的N2产生量具有50%~70%的贡献) (Kuypers et al.,2003),而且清楚地暗示出ANAMMOX菌是到处都存在的.目前基于16S rRNA基因的分析认为, ANAMMOX菌是浮霉菌门分支进化而来的一类自养化能群体.已报道的ANAMMOX菌分属于5个属 (Schmid et al.,2005,2007;Kartal et al., 2007),分别是 Candidutus Brocadia、Candidatus Kuenenia、Candidatus Scalindua、Candidatus Anammoxoglobus和Candidatus Jettenia.厌氧氨氧化工艺具有不需外加有机碳源、曝气能耗低,污泥产量低等优点.目前人们已成功开发、启动并运行了基于ANAMMOX自养脱氮工艺.然而成功启动ANAMMOX反应器的关键因素是ANAMMOX菌的富集和持留,在现有的报道中,大多是以富含ANAMMOX菌污泥、厌氧污泥、好氧污泥或者几种污泥的混合物作为底泥在SBR或UASB等持留污泥能力强的反应器中来启动ANAMMOX反应,而以自然生境中沉积物为底泥启动ANAMMOX的技术报道较少.
利用分子生物学方法对不同生态环境中ANAMMOX菌进行观察和鉴定,发现同一生态系统ANAMMOX菌的微生物多样性较低 (Penton et al.,2006;Schmid et al.,2007;Kartal et al., 2007).白洋淀是我国华北平原最大的淡水湖泊湿地,但是近年来白洋淀水体富营养化现象较为严重,部分水质逐年恶化,引起了专家学者对该地区氮素循环的关注.祝贵兵等利用分子生物学方法证实了白洋淀ANAMMOX菌的存在, 并利用15N同位素示踪技术测得白洋淀湖泊水陆交错岸边带由ANAMMOX菌贡献的N损失量约为103 g·Nm-2·a-1(Zhu et al., 2013).基于此,本研究通过接种以自然生境中的白洋淀岸边带沉积物为底泥进行ANAMMOX的启动,经过9个月的培养富集,对不同时期反应器中菌群结构进行解析,揭示ANAMMOX菌在SBR反应器富集过程中的演变规律,以便采取适当措施针对性的促进ANAMMOX菌的生长,实现厌氧氨氧化反应器的快速启动.
2 材料与方法 (Materials and methods) 2.1 样品来源选取安新县宋庄白洋淀水域的3个岛作为采样点,采集距离水陆交界带的水相1 m处的底泥;样品部分保存在-80 ℃超低温冰箱中直至DNA提取,一部分置于SBR反应器中培养.
2.2 反应器装置和运行条件本实验将白洋淀岸边带沉积物接种在8 L的SBR反应器中进行ANAMMOX系统的启动.反应器有效体积为6 L,装置如图 1所示.设置反应器每天一个循环,每个循环包括5个阶段:曝氮气 (25 min)、进水 (15 min)、厌氧反应 (20 h)、沉淀 (3 h)、排水 (15 min).
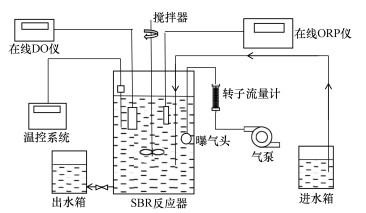 |
| 图 1 试验系统装置示意图 Fig. 1 Diagram of experimental system |
在实验过程中,采用机械搅拌方式,维持反应器温度为35 ℃,搅拌速度在80 r·min-1左右,pH在7.5~8.2之间.整个反应装置均用锡纸包裹以达到避光效果.反应器初始污泥浓度为5500 mg·L-1.实验进水为人工配水,以NH4Cl和NaNO2作为进水中氨氮和亚硝态氮的主要来源,进水NH4Cl和NaNO2的浓度在ANAMMOX系统启动过程中逐渐递增,其中NH4Cl由15 mg·L-1逐渐增加至60 mg·L-1,NaNO2的浓度由15 mg·L-1逐渐增加至78 mg·L-1.此外,进水中还添加基质NaHCO3 1.85 g、KH2PO4 0.00625 g、CaCl2·2H2O 0.3 g、MgSO4·7H2O 0.2 g、FeSO4·7H2O 0.018 g、EDTA·2H2O 0.0125 g和微量元素1 mL.微量元素根据Van de Graaf等 (1996)报道所配.
2.3 样品理化指标分析pH采用Mettler Toledo Delta 320 pH计测定;初始样品NH4+-N、NO2--N、NO3--N依照《土壤农化分析》(第5版) 方法测定 (鲍士旦,2000),采用2 mol·L-1氯化钾溶液浸提2 h,抽滤后用紫外分光光度仪测定.过程水质监测中,NH4+-N采用纳氏试剂分光光度法测定,NO2--N采用N-(1-萘基)-乙二胺分光光度法测定,NO3--N采用氨基磺酸分光光度法测定.
2.4 反应器中细菌群落结构分析 2.4.1 DNA的提取与PCR扩增称取约0.5 g的污泥样品分别利用FAST土壤DNA提取试剂盒提取总DNA (Qbiogene,Carlsbad,CA).总细菌的扩增分别采用通用引物27F (AGAGTTTGATCMTGGCTCAG) 和518R (ATTACCGCGGCTGCTGG) 先进行第一轮PCR扩增,再用带40 bp的GC夹的357F (CCTACGGGAGGCAGCAG) 和518R进行巢式PCR (陈燕等,2015).ANAMMOX菌的扩增采用巢式PCR,第1步采用浮霉菌门通用引物PLA46f/630r (Neef et al., 1998;Purkhold et al., 2000;),再对第1步扩增产物进行第2轮扩增,采用ANAMMOX特异引物Amx368f/Amx820r (Amano et al., 2007) 扩增得到最终产物.
2.4.2 PCR-变性梯度凝胶电泳 (DGGE)PCR-DGGE技术用来解析污泥样品的总细菌群落结构.将1.5 μL的Loading buffer和4 μL PCR扩增后的DNA样品混合后,加入D-code系统 (Bio-Rad,美国),选取8%的聚丙烯酰胺凝胶,变性剂浓度为35%~65%.设置电泳电压110 V, 电泳温度60 ℃, 电泳时间400 min.用SYBR GREEN I染料对凝胶进行染色,最后在凝胶成像系统 (Bio-Rad,Gel-DocTM XR+,美国) 进行拍照,得到DGGE图谱.试验所得图像用BIO-RAD的Quantity One软件分析.通过NTSTS软件利用UPGMA算法对样品间相似性进行分析.微生物多样性指数采用Shannon-wiener指数 (H) 表示 (魏健等,2014),计算公式如下:
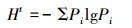
|
(2) |
式中,Pi=ni/N,ni为第i个条带的吸收峰的面积;N为所有条带吸收峰的总面积.
2.4.3 PCR产物的克隆、酶切、测序与系统发育树构建使用Promega Agarose Gel DNA (Solarbio,北京) 纯化试剂盒对PCR扩增产物进行切胶纯化,将纯化后的回收产物与pGEM-Teasy载体 (Promega,Madison,WI) 进行连接,转入Trans1-T1感受态细胞,经涂板37 ℃过夜培养后,进行蓝白斑筛选.随机选取96个白色克隆,制成菌液进行阳性鉴定.用限制性内切酶HhaⅠ和MboⅠ对阳性克隆子进行分型后,挑取菌株进行测序 (金唯智生物科技有限公司,北京).使用DNA star软件对返回的序列进行去载体处理,然后,运用Mothur软件按3%的差异度分析,得出各样品的独立操作单元 (OTU) 及生物多样性指数.每个操作单元中选取代表序列通过NCBI数据库进行在线BLAST比对分析,并与已发表的ANAMMOX细菌16SrRNA序列进行多重序列比对,采用MEGA5.0软件的邻近相接法 (Neighbor-Joining) 构建系统发育树 (Garbeva et al., 2001).
2.4.4 基因的实时荧光定量PCR (qPCR)脱氮微生物氨氧化细菌 (AOB)、亚硝酸盐氧化菌 (NOB) 和ANAMMOX菌的丰度测定采用qPCR技术来完成,其中NOB只检测了最常见的硝化杆菌属和硝化螺菌属的丰度.基因的qPCR采用SYBY Green法,基本过程如下:①标准曲线的制作:将提取的DNA样品送往生物公司 (金唯智生物科技有限公司,北京),由生物公司来完成3种细菌的特异性扩增、克隆、克隆子选取、质粒提取、纯化和定量工作.获得标准质粒后,用TE缓冲液进行系列稀释 (7个浓度梯度),用SYBY Green法进行定量测定.反应体系总容积为20 μL,成份如下:SYBY Green mix 10 μL,正反向引物 (浓度为5 μmol·L-1) 各0.4 μL,DNA模板1 μL,ddH2O补足至20 μL.引物信息和反应条件详见表 1(范强等,2016).另外,每次样品的测定都要进行标准曲线的制作.以基线荧光信号的标准偏差的10倍作为阈值,得到标准曲线与CT值,要求扩增斜率在-3.5~-4.2之间,扩增效率大于95%,溶解曲线为单一峰值.②样品的qPCR测定:采用SYBY Green法进行,引物信息、反应体系和条件均与标准曲线制定过程中的一样.
| 表 1 实验所用引物及反应程序 Table 1 PCR primers in the amplification |
白洋淀采样点水层和沉积物层样品的理化指标如表 2所示.
| 表 2 白洋淀采样点沉积物的相关理化指标 Table 2 Characteristics of the sediments samples in Baiyangdian Lake |
反应器进水为人工配水,采用一般的两段式ANAMMOX启动方法对所取污泥样品 (白洋淀岸边带沉积物) 进行低基质启动.启动过程可分为3个阶段 (图 2),第1~52 d主要为底泥的吸附解吸阶段,HRT为4 d,NH4+和NO2-按物质的量比 (下同) 接近1:1的比例由15 mg·L-1逐渐增加至30 mg·L-1.由于土壤里富含能吸附氨氮和硝酸盐氮的无机酸,使得该时期吸附阶段TN去除率最高可达55%,随着反应器的运行,吸附物质逐渐排出系统,未排除系统的吸附物质开始解吸,导致解吸阶段的TN去除率为负.第53~187 d为ANAMMOX菌的活性迟滞期,HRT由4 d逐渐降至2 d,NH4+和NO2-的投加浓度比例为1:1.32,进水NH4+-N和NO2--N分别增加至50 mg·L-1和66 mg·L-1.该阶段前期异养菌利用细菌死亡裂解释放出的有机物发生反硝化作用,后期异养菌由于缺乏碳源逐渐排出系统,然而由于进水中有溶解氧和反应器搅拌复氧,使得某些氨氧化细菌进行硝化作用,为防止系统崩溃,对装置进行进一步密封,并提高N2的吹脱时间.此阶段的TN去除率维持在20%左右.第188~265 d为ANAMMOX菌的活性提高期 (富集培养阶段),HRT为2 d,NH4+和NO2-的浓度分别增加至60 mg·L-1和78 mg·L-1,NH4+和NO2-的最高去除率达95%以上,该阶段的TN去除最高达82%.此时,NH4+和NO2-的去除比值为1.3左右,ANAMMOX现象明显,污泥由最初的灰色变为土黄色最后变为棕黄色.
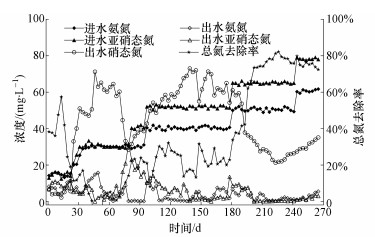 |
| 图 2 反应器氮素转化关系 Fig. 2 The nitrogen transformation in the reactor |
反应器运行不同时期总细菌的DGGE图谱和其泳道对比示意图如图 3所示.图中1~5依次分别为第1、30、90、165、250 d样品.
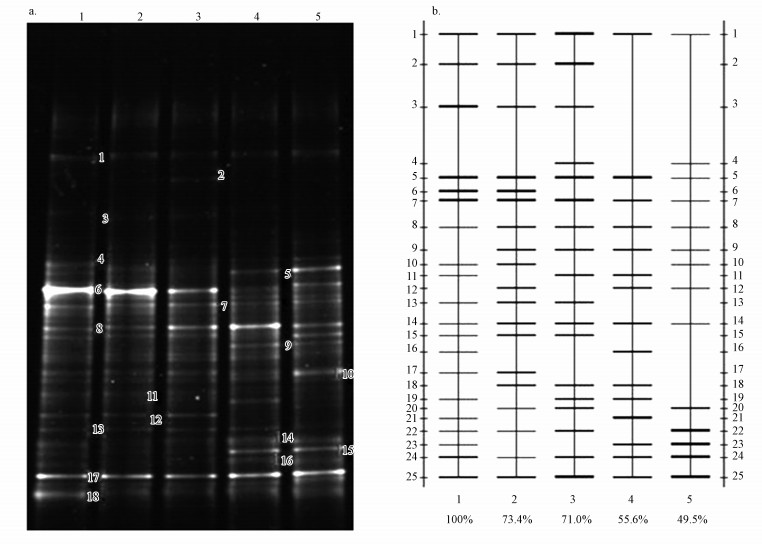 |
| 图 3 不同时期总细菌的DGGE图谱 (a) 及其泳道对比示意图 (b) Fig. 3 DGGE profile of total bacteria under different period (a) and the bands and strength schematic of DGGE profile (b) |
从图 3可以看出,微生物群落结构随反应器运行时间的延长而发生了较大的演变.在启动阶段的第1~30 d里,微生物结构发生较大变化,第30 d的反应器内菌群与第1 d的相似度为73.4%,说明该时期部分不能适应环境的微生物开始被逐渐淘汰,而适应系统环境的微生物开始出现或得到富集.启动阶段的第31~250 d,随反应器运行时间的推移,菌种演变更加明显,部分条带逐渐减弱或消失,如图 3a中的条带2、3、6、11、12、18,说明这部分条带所代表的细菌对系统环境的适应能力降低,并逐渐转变为非优势种群甚至淘汰;一部分条带随反应器运行而逐渐增强,如条带6、8、9、10、14、15和16,说明这些条带所代表的细菌适合在该系统环境下生存,逐渐变为优势菌群,而这部分菌群在该系统中发挥着关键作用;同时也发现部分条带随反应器运行时间的推移并无明显变化,如条带1、4、7和17,这说明此类条带所代表的细菌对环境适应能力强,其中条带17在系统中占有绝对优势地位.通过图 3b中的泳道对比示意图可知,系统运行过程中反应器内微生物与初始微生物的相似度逐渐降低,第250 d的微生物与第1 d的微生物相似度仅为49.5%,这表明经过250 d的运行,系统中群落结构已发生了的动态变化为26.6%~50.5%,属于中高程度的变化 (Osborn et al.,2000), 而且可能会引起系统出水水质的变化,造成系统运行不稳定.
采用NTSYS软件对DGGE图谱进行UPGMA聚类分析,结果如图 4所示.图中1~5依次分别为第1、30、90、165和250 d样品,从该图可以较为直观地看出,微生物种群相似性较高的聚为一类.结果显示,1、2、3聚于一个大的分支,其中2和3又聚于一个小的分支,即第1、30和90 d聚为一个大分支,而第30和85 d的微生物种群相似性最高,然而4和5聚为另一分支,即第165和250 d聚为另一大分支,与初始污泥差异性较大,这说明随反应器运行时间的延长,系统内微生物菌群发生了较大演变.
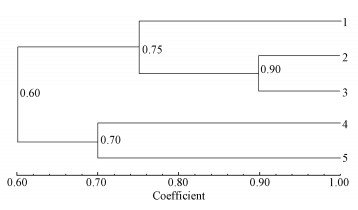 |
| 图 4 不同样品间 UPGMA 聚类分析 Fig. 4 Cluster analysis of DGGE profile by UPGMA of different samples |
根据总细菌的DGGE图谱 (图 3a),利用公式 (2) 计算出系统内不同运行时间微生物的Shannon-Weiner指数,如表 3所示.系统初始运行第1 d时的微生物多样性指数H为2.45,条带数也最多,这说明自然生境中的微生物种类十分丰富,根据DGGE的图谱可知,初始污泥的微生物分布的均匀性较高,各种细菌可以在生境中占有稳定的生态位,构成了生态系统的稳定性,这可能是造成初始污泥样品生物多样性最高的原因.随着反应器运行时间的推移,样品微生物的Shannon-Weiner指数H呈现出先变小后增大的趋势,同时样品的条带数逐渐减少,第165 d时样品微生物的Shannon-Weiner指数最低,H值下降到2.12,条带数减少至15条,第250 d时H值升高至2.27,同时从DGGE图谱可以看出这一时期的优势菌种占据的生态位明显,分布的均匀性有所提高,这说明一部分不适应系统环境的微生物被逐渐淘汰,适应系统环境的微生物逐渐生成或者富集为系统的优势菌种.而随着反应器的长时间运行,优势菌种的分布又趋向于均匀,系统内微生物多样性有所增加,系统最终趋于稳定.
| 表 3 不同时期总细菌的Shannon-Weiner指数 (H) Table 3 Shannon-Weiner index of total bacteria under different time |
对样品阳性克隆子进行测序,并通过Mothur软件将相似度大于97%的序列划分为一个OTU后,得到不同时期基于16SrRNA的ANAMMOX菌的生物多样性指数,具体见表 4.A1~A5分别表示第1、30、90、165和250 d样品的克隆文库名称.结合图 5,稀缺性曲线显示5个基因文库用于测序的克隆数量达到或接近饱和,表明本研究所构建的文库基本涵盖样品中的ANAMMOX菌的多样性.另外,由表 4可知,接种的原始污泥中ANAMMOX菌的生物多样性很低,在系统运行前30 d的适应期里ANAMMOX菌的生物多样性变化不大,Shannon指数和OTU数都非常接近,说明ANAMMOX菌存在于系统中,但是由于其生长缓慢,对环境条件要求高,因此短期内ANAMMOX菌不会发生明显的变化,这也直接导致了该时期的脱氮效率很低.在反应器运行到第90 d和第165 d时,OTU数、Chao1指数和Shannon指数都较高,其中第90 d的最高,这是因为ANAMMOX菌适应了系统环境,同时,氨氮浓度的升高有利于其生长与富集,从而使系统内ANAMMOX菌的生物多样性有所提高,有利于系统的稳定.当反应器运行到第250 d时,已经出现较明显的ANAMMOX现象,此时ANAMMOX菌的Shannon指数仅为0.53,其生物多样性在启动过程中最低,说明在一种特定生境中ANAMMOX菌的群落结构趋向于简单.
| 表 4 不同时期ANAMMOX细菌的α多样性指数 Table 4 Alpha diversity results of ANAMMOX bacteria under different time |
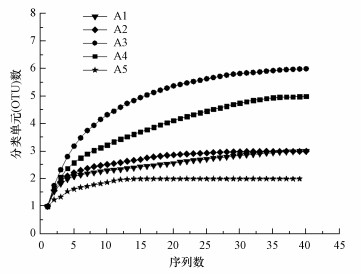 |
| 图 5 ANAMMOX菌16S rRNA基因稀缺性曲线 Fig. 5 Rarefaction curves of ANAMMOX bacteria 16S rRNA libraries |
对样品阳性克隆子进行测序并划分OTU后,选取ANAMMOX菌16S rRNA基因的OTU代表序列和GenBank数据库中的相关序列进行比对,选取同源性较高的序列构建ANAMMOX细菌的系统发育树,结果见图 6.
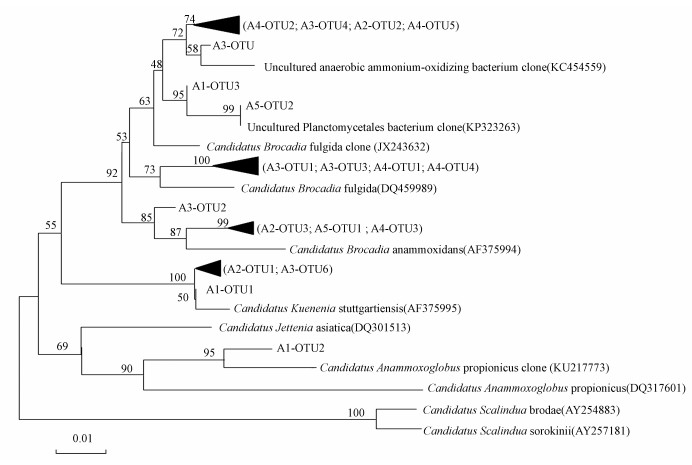 |
| 图 6 不同时期ANAMMOX菌的系统发育树 Fig. 6 Phylogenetic trees of ANAMMOX bacteria under different time |
依据图 6可知,在整个ANAMMOX反应器启动过程中,ANAMMOX菌在逐渐发生着演变.其中初始污泥样品A1文库中的ANAMMOX菌与Candidatus Brocadia fulgida clone、Candidatus Kuenenia stuttgartiensis和Candidatus Anammoxoglobus propionicus clone相似度达到98%以上,其中62.5%的ANAMMOX菌 (A1-OTU3,25条序列) 与Candidatus Brocadia fulgida clone相似度达到99%,说明初始污泥样品中ANAMMOX菌的Brocadia菌属为优势菌属,同时也有Kuenenia菌属和少量的Anammoxoglobus菌属.这是因为在白洋淀岸边带存在厌氧/缺氧界面,季节性覆水也会造成岸边带表露与淹没的交替,表现出强烈的边缘效应 (Wang et al.,2001),加之该地区氮素含量丰富,能为ANAMMOX细菌的生长提供必要的基质,使得该地区的ANAMMOX细菌得以生存.而从A2文库中的ANAMMOX菌的发育树图可以看出,随着反应器运行30 d后,不适应系统环境的Anammoxoglobus菌属被淘汰,Kartal等研究表明, 这种细菌能在颗粒团聚体中检测到,不适宜在絮体污泥中生长 (Kartal et al., 2007),本实验中由于搅拌作用,使原本白洋淀岸边带团聚的小颗粒污泥变为絮体污泥,从而将Anammoxoglobus菌属淘汰.另外,该文库中,72.5%的ANAMMOX菌 (A2-OTU2、A2-OTU3, 共29条序列) 与Candidatus Brocadia属同源,其余ANAMMOX菌与Candidatus Kuenenia 属同源,这说明厌ANAMMOX菌的Brocadia菌属开始得到富集.A3文库中85%的ANAMMOX菌 (A3-OTU1~A3-OTU5,共34条序列) 与Candidatus Brocadia属同源,剩余ANAMMOX菌与Candidatus Kuenenia同源.而由图 4可知,随着反应器的进一步运行,A4和A5文库中的ANAMMOX菌全部隶属于Brocadia属分支,这说明最终反应器内的ANAMMOX菌的优势菌种逐渐演变为单一种属,而以往研究结果表明,在一种特定生境中,ANAMMOX菌多样性较弱,一般占据优势的菌种为单一种属 (Schmid et al.,2007;Kartal et al., 2008;Hu et al., 2010).
综上所述,反应器运行过程中ANAMMOX菌优势菌种隶属于Brocadia属,其在反应器的启动过程中发挥着重要作用,同时Kuenenia菌属的优势地位逐渐减弱直到消失.当反应器运行至30 d时Anammoxoglobus属已被淘汰,而随着反应器运行时间的推移,ANAMMOX菌种趋向于单一,最终为Brocadia属.
3.5 反应器中主要功能菌的丰度采用实时荧光定量PCR技术检测反应器中主要功能菌的丰度,根据反应器运行过程中细菌丰度的动态变化,探究对系统脱氮效果的影响,为后续优化实验条件提供数据支持,结果如图 7所示.通过实验测得初始污泥样品中ANAMMOX菌、AOB和NOB的丰度分别为1.98×107、2.11×106和1.07×106 copies·g-1干污泥.依图 7可知在反应器启动的前30 d,3种细菌的丰度均有所提高,但是各种细菌的丰度依旧很低,TN的去除主要是由于吸附作用.而在随后的运行中由于溶解氧浓度的降低,NOB数量开始下降,而适应系统环境的AOB比NOB对氧的亲和能力高 (Laanbroek et al., 1993),因此AOB会继续生长,当反应器运行到第90 d后,AOB的丰度开始降低,因为由于AOB的生长造成亚硝酸盐的积累,反过来又影响AOB的生长和氧化活性 (祖波等,2009),此时耐受低氧的NOB有增长的趋势.第1~180 d ANAMMOX菌的丰度变化不大,活性较低,而当反应器运行180 d后AOB和NOB的丰度减少较快,ANAMMOX现象逐渐明显,脱氮效率增大,此时ANAMMOX菌的丰度逐渐升高至1.73×109 copies·g-1干污泥.图 7表明,除吸附作用外,总氮的去除率与厌氧氨氧化菌的丰度成正相关.
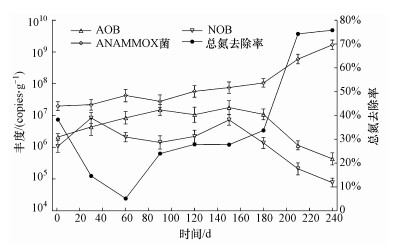 |
| 图 7 反应器运行过程中功能细菌的丰度变化趋势 Fig. 7 The number variation tendency of the function bacterial in the reactor during start-up process |
1) 以自然生境中白洋淀岸边带沉积物为底泥成功进行了低基质厌氧氨氧化SBR反应器的启动,富集培养阶段TN的去除率最高为82%.
2) PCR-DGGE的解析和UPGMA聚类分析结果表明,随着反应器运行时间的推移,系统内微生物群落结构发生了较大演变,运行过程中的微生物菌群动态变化水平为26.6%~50.5%.对DGGE图谱的Shannon指数分析表明,微生物多样性先变小后增大,这种变化会使优势菌种得到重新分布,系统最终趋于稳定状态.
3) ANAMMOX细菌的系统发育树分析结果表明,反应器内运行过程中厌氧氨氧化菌优势菌种隶属于Brocadia属,同时,原有污泥中的Anammoxoglobus属和Kuenenia属ANAMMOX菌不适应系统环境被逐渐淘汰.随着反应器运行时间的推移,厌氧氨氧化菌种趋向于单一种属,Brocadia属最终成为优势ANAMMOX菌属,在反应器的启动过程中发挥着重要的脱氮作用.
4) 定量PCR结果表明,在ANAMMOX细菌的活性提高后,ANAMMOX细菌得到了富集,富集培养阶段SBR系统中ANAMMOX细菌的最大生物量达到了1.73 × 109 copies·g-1干污泥,通过与反应器中总氮的去除率比较可知,除吸附作用外,TN的去除率与ANAMMOX菌的丰度成正相关.
| [${referVo.labelOrder}] | Amano T, Yoshinaga I, Okada K, et al. 2007. Detection of anammox activity and diversity of anammox bacteria-related 16S rRNA genes in coastal marine sediment in Japan[J]. Microbes and Environment, 22(3) : 232–242. DOI:10.1264/jsme2.22.232 |
| [${referVo.labelOrder}] | Broda E. 1977. Two kinds of lithotrophs missing in nature[J]. Zeitschrift Für Allgemeine Mikrobiologie, 17(6) : 491–493. DOI:10.1002/(ISSN)1521-4028 |
| [${referVo.labelOrder}] | 鲍士旦. 2000. 土壤农化分析[M]. 北京:: 中国农业出版社. |
| [${referVo.labelOrder}] | 陈燕, 刘国华, 范强, 等. 2015. 不同溶解氧条件下A/O系统的除碳脱氮效果和细菌群落结构变化[J]. 环境科学, 2015, 36(7) : 2610–2616. |
| [${referVo.labelOrder}] | 范强, 刘国华, 徐相龙, 等. 2016. 同步半硝化-厌氧氨氧化-反硝化一体式反应器的运行条件和微生物丰度研究[J]. 环境工程, 2016, 34(5) : 6–10. |
| [${referVo.labelOrder}] | Garbeva P, Van Overbeek L S, Van Vuurde J W L, et al. 2001. Analysis of endophytic bacterial communities of potato by plating and denaturing gradient gel electrophoresis (DGGE) of 16S rDNA based PCR fragments[J]. Microbial Ecology, 41(4) : 369–383. DOI:10.1007/s002480000096 |
| [${referVo.labelOrder}] | Hu B L, Zheng P, Tang C J, et al. 2010. Identification and quantification of anammox bacteria in eight nitrogen removal reactors[J]. Water Research, 44(17) : 5014–5020. DOI:10.1016/j.watres.2010.07.021 |
| [${referVo.labelOrder}] | 姜博, 祝贵兵, 周磊榴, 等. 2015. 低温高海拔湖泊岸边带厌氧氨氧化菌的存在/生物多样性及活性——以天山天池为例[J]. 环境科学学报, 2015, 35(7) : 2045–2051. |
| [${referVo.labelOrder}] | Kuypers M M, Sliekers A O, Lavik G, et al. 2003. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J]. Nature, 422(6932) : 608–611. DOI:10.1038/nature01472 |
| [${referVo.labelOrder}] | Kartal B, Rattray J, van Niftrik L A, et al. 2007. Candidatus 'Anammoxoglobus propionicus' a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 30(1) : 39–49. DOI:10.1016/j.syapm.2006.03.004 |
| [${referVo.labelOrder}] | Kartal B, Van Niftrik L, Rattray J, et al. 2008. Candidatus 'brocadia fulgida':an autofluorescent anaerobic ammonium oxidizing bacterium[J]. Fems Microbiology Ecology, 63(1) : 46–55. DOI:10.1111/fem.2008.63.issue-1 |
| [${referVo.labelOrder}] | Laanbroek H J, Gerards S. 1993. Competition for limiting amounts of oxygen between nitrosomonas-europaea and nitrobacterwinogradskyi grow in mixed continuous cultures[J]. Archives of Microbiology, 159(5) : 453–459. DOI:10.1007/BF00288593 |
| [${referVo.labelOrder}] | Neef A, Amann R, Schlesner H, et al. 1998. Monitoring a widespread bacterial group:in situ detection of planctomycetes with 16S rRNA targeted probes[J]. Microbiology, 144(12) : 3257–3266. DOI:10.1099/00221287-144-12-3257 |
| [${referVo.labelOrder}] | Osborn A M, Moore E R B, Timmis K N. 2000. An evaluation of terminal-restriction fragment length polymorphism (T-RFLP) analysis for the study of microbial community structure and dynamics[J]. Environmental Microbiology, 2(1) : 39–50. DOI:10.1046/j.1462-2920.2000.00081.x |
| [${referVo.labelOrder}] | Penton C R, Devol A H, Tiedje J M. 2006. Molecular evidence for the broad distribution of anaerobic ammonium-oxidizing bacteria in freshwater and marine sediments[J]. Applied and Environmental Microbiology, 72(10) : 6829–6832. DOI:10.1128/AEM.01254-06 |
| [${referVo.labelOrder}] | Purkhold U, Pommerening-Röser A, Juretschko S, et al. 2000. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis:implications for molecular diversity surveys[J]. Applied and Environmental Microbiology, 66(12) : 5368–5382. DOI:10.1128/AEM.66.12.5368-5382.2000 |
| [${referVo.labelOrder}] | Rysgaard S, Glud R N. 2004. Anaerobic N2 production in arctic sea ice[J]. Limnology and Oceanography, 49(1) : 86–94. DOI:10.4319/lo.2004.49.1.0086 |
| [${referVo.labelOrder}] | Strous M, Heijnen J J, Kuenen J G, et al. 1998. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammonium-oxidizing microorganisms[J]. Applied Microbiology and Biotechnology, 50(5) : 589–596. DOI:10.1007/s002530051340 |
| [${referVo.labelOrder}] | Schmid M C, Risgaard Petersen N, van de Vossenberg J, et al. 2007. Anaerobic ammonium-oxidizing bacteria in marine environments:widespread occurrence but low diversity[J]. Environmental Microbiology, 9(6) : 1476–1484. DOI:10.1111/emi.2007.9.issue-6 |
| [${referVo.labelOrder}] | Schmid M C, Maas B, Dapena A, et al. 2005. Biomarkers for the in situ detection of anaerobic ammonium oxidizing (anammox) bacteria[J]. Applied and Environmental Microbiology, 71(4) : 1677–1684. DOI:10.1128/AEM.71.4.1677-1684.2005 |
| [${referVo.labelOrder}] | Schmid M C, Risgaard-Petersen N, Van de Vossenberg J, et al. 2007. Anaerobic ammonium-oxidizing bacteria in marine environments:widespread occurrence but low diversity[J]. Environmental Microbiology, 9(6) : 1476–1484. DOI:10.1111/emi.2007.9.issue-6 |
| [${referVo.labelOrder}] | Third K A, Paxman J, Schmid M, et al. 2005. Enrichment of anammox from activated sludge and its application in the CANON process[J]. Microbial Ecology, 49(2) : 236–244. DOI:10.1007/s00248-004-0186-4 |
| [${referVo.labelOrder}] | Van de Graaf A A, Astrid A, Arnold Mulder. 1995. Anaerobic oxidation of ammonium is a biologically mediated process[J]. Applied Environmental Microbiology, 61(4) : 1246–1251. |
| [${referVo.labelOrder}] | Van de Graaf A A, De Bruijn P, Robertson L A, et al. 1997. Metabolic pathway of anaerobic ammonium oxidation on the basis of 15N studies in a fluidized bed reactor[J]. Microbiology, 143(7) : 2415–2421. DOI:10.1099/00221287-143-7-2415 |
| [${referVo.labelOrder}] | Van de Graaf A A, De Bruijn P, Robertson L A, et al. 1996. Autotrophic growth of anaerobic ammoniumoxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology (UK), 142(8) : 2187–2196. DOI:10.1099/13500872-142-8-2187 |
| [${referVo.labelOrder}] | 王衫允, 祝贵兵, 曲冬梅, 等. 2012. 白洋淀富营养化湖泊湿地厌氧氨氧化菌的分布及对氮循环的影响[J]. 生态学报, 2012, 32(21) : 6591–6598. |
| [${referVo.labelOrder}] | 王雨, 祝贵兵, 王朝旭, 等. 2013. 高含氮稻田深层土壤的氨氧化古菌和厌氧氨氧化菌共存及对氮循环的影响[J]. 生态学报, 2013, 33(6) : 1487–1493. |
| [${referVo.labelOrder}] | 魏健, 宋永会, 赵乐. 2014. MBR处理腈纶废水的效能及微生物群落结构分析[J]. 环境科学, 2014, 35(12) : 4610–4617. |
| [${referVo.labelOrder}] | Wang W D, Wang D L, Yin C Q, et al. 2001. A study on the groundwater quality of the Baiyangdian wetland ecosystem[J]. Acta Ecologica Sinica, 21(6) : 919–925. |
| [${referVo.labelOrder}] | Zhu G B, Wang S Y, Wang W D, et al. 2013. Hotspots of anaerobic ammonia oxidation at land-freshwater interfaces[J]. Nature Geoscience, 6 : 103–107. DOI:10.1038/ngeo1683 |
| [${referVo.labelOrder}] | 祖波, 张代钧, 祖建, 等. 2009. 硝化过程中影响亚硝酸盐积累的因素[J]. 环境科学技术, 2009, 32(4) : 1–5. |
 2017, Vol. 37
2017, Vol. 37


