2. 中国科学院青藏高原研究所, 高寒生态学与生物多样性实验室, 北京 100101
2. Key Laboratory of Alpine Ecology and Biodiversity, Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101
隐孢子虫引起的隐孢子虫病害可通过饮用水传播,对人类健康具有重大威胁,尤其对免疫力低下者可引发致死性腹泻.直径在4~6 μm隐孢子虫厚壁卵囊能抵抗常规水处理中的次氯酸消毒并在水中存活数月,而且在4 ℃水中保存13个月仍然具有感染活性(陈甫,2008), 饮用水中隐孢子虫的检测可以预防病害流行与发生,保证饮用水水质安全.目前国内及国际上生活饮用水中隐孢子虫的检测主要依据免疫磁分离方法来分离卵囊和荧光显微镜镜检卵囊(崔艳梅等,2016;余素华等,2009;King et al., 2016),这也是我国和美国饮用水中隐孢子虫检测的标准方法.该方法采用免疫磁分离试剂分离卵囊, 并使用免疫荧光技术进行染色镜检,其中显微镜观察比较依赖人的主观判断,某些荧光抗体的背景信号高、特异性低,容易引起结果不准确(Lemos et al., 2005;Quintero-Betancourt, 2003),而且免疫磁分离试剂价格昂贵,限制了该方法的广泛应用.近年来正在发展分子方法检测隐孢子虫,分子方法检测速度快,灵敏性高,选用不同的目标基因进行扩增,不仅可以测定样品的阳性率还可以进行qPCR基因定量(顾有方等,2015;Chalmers et al., 2013).目前饮用水或水源水中隐孢子虫分子检测仍处于探索阶段,未发展出成熟实用的方法,尤其在样品前处理等方面存在一定的问题.当采集样品量较低时(低于2.5 L),通常使用47 mm左右滤膜过滤,通过剪碎滤膜来直接提取样品DNA,进而对隐孢子虫基因进行定量分析(Guy et al., 2003;王明星等,2016),由于采集的样品体积量较少,因此也相应的减少了DNA含量,导致检测的范围缩小,不能正确指示受隐孢子虫污染情况.当采集样品量较多(高于5 L时),目前大多报道仍是依赖于免疫磁分离的方法分离卵囊(Almeida et al., 2010;Helmi et al., 2011;Xiao et al., 2006),实验成本较高而不利于广泛应用.本研究使用盘式过滤器过滤10 L水样品,滤膜直径142 mm,丙酮溶解滤膜,进而沉淀、浓缩,直接提取浓缩物DNA进行隐孢子虫qPCR定量检测,本研究对整个体系进行了测试和优化,建立了一种qPCR定量检测饮用水中隐孢子虫的有效方法.
2 材料与方法(Materials and methods) 2.1 隐孢子虫标准品及DNA提取 2.1.1 隐孢子虫小标样本研究中使用的隐孢子虫标准样品均购自美国Waterborne公司.共购有两个不同规格的标准品,一个被称为是隐孢子虫小标样,该标准品中包含(100±1)个完整的隐孢子虫卵囊(oocysts of Cryptosporidium parvum, Iowa strain),卵囊悬浮于0.75 mL磷酸盐缓冲液内,产品编号是PACIR3,产品批号是lot 94,用于体系测试和优化.
2.1.2 隐孢子虫大标样另一个标准品型号被称为隐孢子虫大标样,该标准品包含大约5×106个卵囊(oocysts of Cryptosporidium parvum, Iowa strain),卵囊悬浮于8 mL磷酸盐缓冲液内.产品编号是P104@5×10/6,批号为6-15,该标样70 ℃水浴10 min灭活后用于隐孢子虫DNA提取实验和水样加标测试.
2.1.3 隐孢子虫标准品DNA 4种提取方法比较将盛有隐孢子虫标准品的样品管(lot 94)最大速度涡旋5~10 min,取37.5 μL标准品悬浮溶液提取DNA.共采用4种方法提取DNA,每个方法4个平行.方法1~3均使用QIAamp DNA mini kit(QIAGEN公司)试剂盒,均采用冻融法进行前处理,首先加入360 μL ATL裂解液到盛有37.5 μL标准品悬浮溶液的EP管中,进行冻融前处理,用来裂解卵囊囊壁.方法1~4具体区别见表 1.
| 表 1 4种DNA提取方法列表 Table 1 Four DNA extraction method |
将2.1.2节中大标样最大速度涡旋5~10 min,将沉入底部和粘附在管壁的卵囊均匀悬浮起来, 取高浓度卵囊悬液100 μL,进行系列稀释,共稀释1000倍,取4 μL卵囊悬液(根据10次镜检结果确认(4±1)个卵囊)转入离心管中按照2.1.3节中方法3提取DNA,设置3个平行.
2.2 水样处理及DNA提取 2.2.1 水样加标参考2.1.4节中方法,对高浓度卵囊悬液进行系列稀释,取稀释后的80 μL卵囊悬液(经10次平行镜检确定为(100±10)个卵囊),分别加到10 L去离子水(本研究中使用的去离子水均已通过0.45 μm滤膜过滤)、10 L地下水源中水测试加标回收率.同样体积的去离子水和地下水源水分别设置不加标阴性对照,除了加标步骤去除,后续实验步骤与相应的阳性处理相同.
2.2.2 水样膜过滤及丙酮溶解浓缩水样前处理过程如下,使用盘式过滤器(KS-Ⅱ不锈钢过滤器,142 mm,日本ADVANTEC公司)和蠕动泵(BT300 1F, 保定兰格)过滤加标水样, 滤膜为142 mm直径、1 μm孔径的混合醋酸纤维素滤膜, 水样完全过滤后再依次用PBST(磷酸盐缓冲液+0.01%Tween80)、70%~100%乙醇、MiliQ超纯水清洗容器;收集滤膜放于50 mL离心管中,添加40 mL丙酮并晃动直至滤膜完全溶解(Hashimoto et al., 2002;张冬青,2009);1550 g离心力离心10 min,弃上清液,重复离心3~4次,直至滤膜完全溶解;向管中加入磷酸盐缓冲液,直至40 mL,2000 g离心力离心15 min,弃上清液,再加入磷酸盐缓冲液继续离心,洗去残留的丙酮.将沉淀物全部转移到5 mL离心管,14000 g离心,去掉部分上清夜,留取不超过400 μL沉淀物.
2.2.3 水样DNA提取加入一定体积的ATL裂解液到盛有水样浓缩液的EP管中(裂解液体积与悬浮液体积比为2.25:1),采用2.1.3节中方法3进行冻融前处理,加入蛋白酶K水浴3 h,取出EP管并放于90 ℃水浴中水浴20 min,取出EP管冰浴1 min,14000 g离心5 min,去除杂质.将上清液转到另一个EP管中,重新按照试剂盒操作进行,不再加入蛋白酶K, 其他步骤参照QIAamp DNA mini kit操作说明.收集洗脱DNA进行后续qPCR检测.
2.2.4 加标回收率的计算加标回收率可以根据加标的卵囊数目计算,也可以根据加标卵囊的基因拷贝数计算.本研究按照后者进行计算.回收率=(加标后卵囊基因的拷贝数-阴性对照卵囊基因的拷贝数)/加入的标准品卵囊的基因拷贝数×100%.
2.3 普通PCR引物测试选用了4套隐孢子虫引物分别进行特异性测试.这4套引物分别是:①扩增18S rRNA基因的JVAF/ R引物(Jothikumar et al., 2008),片段大小是159 bp,前引物JVAF序列是5′-ATGACGGGTAACGGGGAAT-3′,后引物JVAR序列是5′-CCAATTACAAAACCAAAAAGTCC-3′;②扩增18S rRNA基因的CRU18SF/R引物(Hadfield et al., 2011),片段大小是299 bp,CRU18SF前引物序列是5′-GAGGTAGTGACAAGAAATAACAATACAGG-3′,CRU18SR后引物序列是5′-CTGCTTTAAGCACT CTAATTTTCTCAAAG-3′;③扩增18S rRNA基因的18SF/R引物(Morgan et al., 1997),片段大小是283 bp,18SF前引物序列是5′-AGTGACAAGA AATAACAATACAGG-3′,18SR后引物序列是5′-CCTGCTTTAAGCACTCTAATTTTC-3′;④扩增Actin基因的Actin F/R引物(Yang et al., 2014),片段大小是163 bp,Actin F前引物序列是5′-ATCGTGAAAGAATGACWCAAATTATGTT-3′,Actin R后引物序列是5′-ACCTTCATAAATTGGAACG GTGTG-3′.分别用隐孢子虫标准品DNA、5 L去离子水DNA和5 L管网水DNA(管网水显微镜镜检隐孢子虫为阴性)对4套引物进行测试,去离子水和管网水水样浓缩和DNA提取参照2.2.2节和2.2.3节.
普通PCR反应体系:2XGoldSar Best Master Mix (康为世纪) 25 μL;10 μmol·L-1前引物及后引物各0.5 μL;模板DNA 2 μL; 加水补足50 μL.普通PCR扩增程序:第一步预变性,95 ℃,10 min;第二步,39个循环,包括94 ℃,30 s;57 ℃,45 s;72 ℃,19 s;第三步最后72 ℃延伸7 min.
引物测试模板DNA分别来自标准品隐孢子虫、经显微镜镜检检测确认没有隐孢子虫的5 L去离子水和5 L管网水.去离子水和管网水DNA提取及前处理浓缩均参照方法2.2.2节和2.2.3节.
普通PCR扩增产物在1.8%的琼脂糖凝胶电泳上检测,将假阳性扩增产物通过MinElute PCR Purification Kit(小片段PCR产物纯化试剂盒,QIAGEN公司)纯化后交给华大基因公司进行克隆测序.
2.4 qPCR测试 2.4.1 质粒制备引物对JVAF/R扩增标准品隐孢子虫DNA,PCR产物均经过PCR产物纯化试剂盒纯化后进行连接克隆,提取含有正确插入片段的克隆子的质粒,并使用Nanodrop 1000 (ThermoScientific)测定质粒DNA浓度,最后10倍逐级稀释为6个系列浓度用作qPCR标准曲线,分别从每微升包含106基因拷贝到每微升包含101基因拷贝.
2.4.2 SYBR green染料法qPCR测试SYBR green染料法qPCR反应体系:2X power SYBR green Master mix (ABI)10 μL; 10 μmol·L-1前引物及后引物各0.25 μL; 模板DNA 1.5 μL; 加水补足20 μL.扩增程序:第一步预变性,95 ℃,10 min;第二步,40个循环,包括95 ℃,15 s;60 ℃,1 min;在60 ℃处采集数据.染料法qPCR用于测试质粒标准曲线和隐孢子虫DNA 4种提取方法的检测.
2.4.3 Taqman探针qPCR测试Taqman探针qPCR反应体系:2XTaqMan Universal Master Mix‖10 μL(ABI); 10 μmol·L-1引物JVAF、JVAR各0.5 μL;10 μmol·L-1 Cryptosporidium探针(5′VIC, 3′BHQ1)0.3 μL; 模板DNA 1.5 μL,加水补足20 μL.qPCR扩增程序:第一步预变性,95 ℃,10 min;第二步,40个循环,包括95 ℃,15 s;57 ℃,45 s;72 ℃,45 s;在72 ℃,45 s处采集数据.探针法qPCR除用于质粒标准曲线测试,也用于隐孢子虫DNA 4种提取方法的检测及加标回收率检测.
3 结果(Results) 3.1 体系测试及优化 3.1.1 隐孢子虫引物测试及筛选使用4对引物进行普通PCR测试,模板分别是隐孢子虫标准品DNA、5L管网水DNA、5 L去离子水DNA,扩增产物经琼脂糖电泳检测扩增产物,结果见表 2.
| 表 2 隐孢子虫引物测试 Table 2 Primers test of Cryptosporidium |
前3对引物均检测隐孢子虫Cryptosporidium spp.的18S rRNA基因区域,第4对引物检测Actin基因区域.这4套引物扩增隐孢子虫标准品DNA的结果均显示正确片段的阳性扩增,均未见非特异条带产物.5 L去离子水DNA的扩增结果也均显示阴性.然而,引物对CRU18SF/R、18SF/R及Actin F/R扩增5 L管网水DNA结果显示有阳性产物.管网水和去离子水均经过显微镜镜检测试,未检出隐孢子虫,因此认为出现的扩增产物是假阳性产物.将阳性扩增条带回收进行克隆测序,序列结果在GenBank中比对,最相似微生物不是隐孢子虫,而是藻类微生物.表 1中除了JVAF/R之外的3对引物均出现了假阳性扩增,因此,在后续实验中,均采用JVAF/R这对引物,以防出现假阳性扩增.
3.1.2 隐孢子虫SYBR green染料法和Taqman探针法比较SYBR green染料法和Taqman探针法qPCR分别使用相同系列稀释的质粒标准品,检测标准曲线, 结果见图 1.
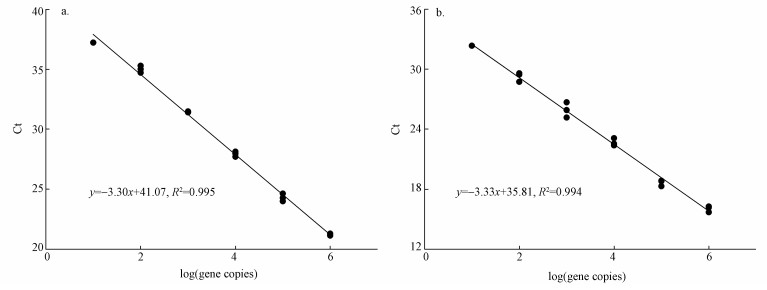 |
| 图 1 隐孢子虫qPCR标准曲线 (a.Taqman探针法, b. SYBR green染料法) Fig. 1 qPCR standard curve of Cryptosporidium (a.Taqman, b. SYBR green) |
从图中可知,相同浓度的标准品,染料法循环阈值(Cycle threshold, Ct)响应值低于探针法.基因拷贝数为10的质粒标准品,染料法检测的Ct值在30~35之间,探针法检测的Ct值在35~40之间.根据文献报道(Jothikumar et al., 2008),一个卵囊中有20个18S rRNA基因的拷贝,从图 1中可以看出,两种方法均可以检测到10个18S rRNA基因拷贝,即0.5个卵囊, 这个检测灵敏性与文献报道中相似(Fontaine et al., 2002).由于18S rRNA比较保守,因此,为了在检测中提高特异性,大多数实验采用的是Taqman探针法检测隐孢子虫(Jothikumar et al., 2008;Fontaine et al., 2002).
3.1.3 隐孢子虫标准品DNA提取比较4种方法分别比较了液氮/65 ℃冻融和液氮/沸水水浴冻融前处理,QIAamp DNA mini kit和MP FastDNA Spin kit试剂盒对隐孢子虫卵囊DNA提取的影响,qPCR的基因定量结果见图 2.Ct值越高,基因拷贝数越低,即DNA产量越低.SYBR Green染料法和Taqman探针法qPCR的检测结果相同:方法3的基因拷贝数高于其他方法, 方法1与方法2之间没有明显差异,方法1~3的基因拷贝数远高于方法4.方法3,液氮/沸水水浴,冻融5次之后使用QIAamp DNA mini kit的方法提取隐孢子虫DNA量较高.MP FastDNA Spin试剂盒提取隐孢子虫DNA产量最低.因此,方法3是较理想的隐孢子虫DNA的提取方法.
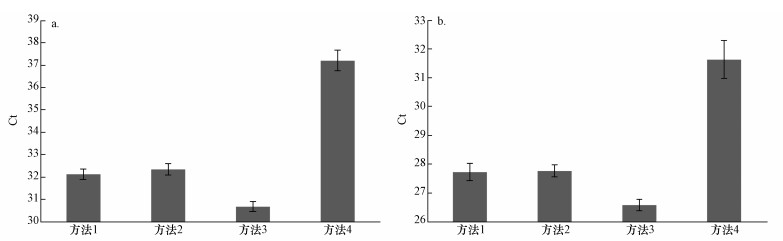 |
| 图 2 隐孢子虫DNA4种提取方法的qPCR检测 (a.Taqman探针法, b. SYBR green染料法) Fig. 2 qPCR comparison of Cryptosporidium spp. four DNA extraction methods (a.Taqman, b. SYBR green) |
依据2.1.3节中方法3,提取(4±1)个隐孢子虫卵囊DNA,采用探针法qPCR进行18S rRNA基因定量检测,测定的Ct平均值是37.29,标准偏差是0.43.因此,本研究中的方法3可以提取出(4±1)个隐孢子虫卵囊DNA.
3.2 加标回收率10 L去离子水、10 L地下水源水分别加标(100±10)个隐孢子虫卵囊,按照水样品前处理方法(2.2.2节)进行盘式过滤器过滤、丙酮溶解浓缩.沉淀浓缩液按照2.2.3节中的方法直接提取DNA.探针法qPCR检测结果见表 3和表 4.去离子水的加标回收率在50%以上,地下水源水样回收率达到46%.
| 表 3 去离子水中隐孢子虫加标回收 Table 3 Recovery result of Cryptosporidium in purified water |
| 表 4 地下水源水中隐孢子虫加标回收 Table 4 Recovery result of Cryptosporidium in underground water |
MP FastDNA kit是提取隐孢子虫DNA的一个常用选择(Li et al., 2016), 本研究发现该试剂盒提取隐孢子虫DNA量远低于QIAamp DNA mini kit,而且冻融前处理配合MP FastDNA kit效果也较差(数据未列出).根据文献报道(Carry et al., 2004;Harris et al., 1999),隐孢子虫卵囊外部结构中含有大量丝状蛋白组织,蛋白酶K对这种结构组织有较强的裂解效果,超声波几乎对这种结构没有任何破坏,胃蛋白酶(pepsin)的裂解效果也不如蛋白酶K明显,因此,QIAamp DNA mini kit里的蛋白酶K可能对隐孢子虫卵囊壁起了主要裂解作用.有文献报道液氮/沸水处理对DNA构像破环较大,容易使DNA二级结构混乱,进而影响以后的扩增效果(Guy et al., 2004),本研究比较了液氮/沸水冻融与液氮/65 ℃水浴冻融前处理,结果显示, 液氮/沸水5次冻融DNA扩增产量稍高于液氮/65 ℃冻融15次.另外,液氮/沸水冻融次数达到10次时DNA扩增效率明显下降(数据未列出),因此液氮/沸水冻融次数多会破坏DNA结构,影响后续的扩增效率.因此,5次液氮/沸水冻融既有助于破坏卵囊壁结构也没有影响扩增效率.
4.2 浓缩液直接提取隐孢子虫DNA方法的应用由于水源水中杂质较多,影响PCR扩增因素较多,本研究中加标的地下水源水样品采用2.1.3节中方法3提取DNA,qPCR测试没有检测到扩增产物.本研究参考相关文献对水源水DNA提取方法做了进一步优化(Nichols et al., 2003),采用方法为2.2.3节中的优化提取方法,去除了杂质等抑制剂后,回收率达到40%以上.优化后方法去除了泥土杂质等影响扩增的抑制物,同时整个实验过程没有外加试剂干扰,均采用QIAamp DNA mini kit试剂盒配备试剂,避免增加额外试剂到裂解液中,防止外加试剂会破坏缓冲液pH等微环境影响最后DNA的洗脱回收.因此,最大量回收DNA.
直接提取浓缩液DNA的方法,突破了常规方法中使用免疫磁珠分离卵囊的限制,极大的节省了实验成本.该直接提取DNA方法不仅与滤膜溶解浓缩配合使用,也可以与国标法中的滤囊/滤芯过滤、淘洗浓缩的方法配合使用.优化后的水样品DNA提取方法可直接提取大量水样品的回收浓缩液,在隐孢子虫分子检测中具有重要意义.
另外,水样品的来源和成份对隐孢子虫回收率影响较大,不同水源水隐孢子虫的加标回收率会有差异.
4.3 滤膜溶解浓缩饮用水的方法本方法使用盘式过滤器过滤,142 mm直径滤膜过滤样品量大,可以截留较多水样量中微生物,降低检出限,提高检测灵敏性.而且在3次丙酮溶解滤膜和磷酸盐沉淀中,可溶性杂质溶解在上清夜中,在前处理中被去除,有助于提高下游基因定量实验的扩增效率.滤膜的完全溶解是提高效果的一个关键步骤,如果滤膜不能充分溶解,隐孢子虫卵囊会被包裹在半溶解的胶粘状的滤膜碎片中不能被释放出来,影响DNA的提取.因此,要保证丙酮完全溶解滤膜.
4.4 SYBR Green染料法检测隐孢子虫的应用前景SYBR Green染料法是以SYBR GreenⅠ荧光染料为基础的一种qPCR检测方法,SYBR GreenⅠ是一种非特异的荧光染料,当与双链DNA(dsDNA)结合后发出强烈荧光,可以根据其荧光信号检测出结合的dsDNA数量;Taqman探针是一种寡核苷酸探针,在5′端连接的是荧光集团,而猝灭剂连接在3′端,寡核苷酸与靶序列特异性互补,每扩增一条DNA链,就有1个探针被切断并释放1个荧光信号,随着扩增循环数的增加,释放出来的荧光集团不断增多,荧光信号不断增强.两种方法都已经被广泛用于基因定量研究,但鲜有报道使用染料法定量检测隐孢子虫,本研究中SYBR Green染料法qPCR使用了JVAF/R引物对检测隐孢子虫,扩增产生的溶解曲线具有单一峰形,无非特异性扩增,该体系Ct响应值远低于探针法,而且检测结果与探针法相同,结果准确可靠.JVAF/R引物对扩增隐孢子虫的特异性需要进一步测试,从而将染料法qPCR发展成为可应用的隐孢子虫分子定量检测方法.
5 结论(Conclusions)1) 筛选出了隐孢子虫特异性的引物JVAF/JVAR.
2) 可以提取到(4±1)个隐孢子虫卵囊DNA.
3) 建立了用于隐孢子虫检测的直接提取浓缩液DNA的方法,极大的节省了实验成本.
4) 建立了可用于大量饮用水检测的隐孢子虫分子检测方法.
Almeida A, Moreira M, Soares S, et al. 2010. Biological and genetic characterization of Cryptosporidium spp.and giardia duodenalis isolates from five hydrographical basins in Northern Portugal[J]. The Korean Journal of Parasitology, 48(2): 105–111.
|
Cary C M, Lee H, Trevors J T. 2004. Biology, persistence and detection of Cryptosporidium parvum and Cryptosporidium hominis oocyst[J]. Water Research, 38: 818–862.
DOI:10.1016/j.watres.2003.10.012
|
Chalmers R M, Katzer F. 2013. Looking for Cryptosporidium:the application of advances in detection and diagnosis[J]. Trends in Parasitology, 29(5): 237–251.
DOI:10.1016/j.pt.2013.03.001
|
陈甫. 2008.动物隐孢子虫的分子流行病学、致病机理与抗感染药物的研究[D].南京: 南京农业大学
|
崔艳梅, 王云霞, 段效辉, 等. 2016. 水环境中贾第鞭毛虫和隐孢子虫检测方法的研究进展[J]. 食品安全质量检测学报, 2016, 7(4): 1369–1374.
|
Fontaine M, Guillot E. 2002. Development of a TaqMan quantitative PCR assay specific for Cryptosporidium parvum[J]. FEMS Microbiolology Letters, 214: 13–17.
DOI:10.1111/fml.2002.214.issue-1
|
顾有方, 汪凯, 刘德义, 等. 2015. 宠物犬蓝氏贾第鞭毛虫和隐孢子虫感染的分子检测刊名[J]. 中国寄生虫学与寄生虫病杂志, 2015, 5(33): 362–367.
|
Guy R A, Payment P, Krull U J, et al. 2003. Real-time PCR for quantitation of Giardia and Cryptosporidium in environmental water samples and sewage[J]. Applied and Environmental Microbiolology, 69: 5178–5185.
DOI:10.1128/AEM.69.9.5178-5185.2003
|
Guy R A, Xiao C, Horgen P A. 2004. Real-Time PCR assay for detection and genotype differentiation of Giardia lamblia in stool specimens[J]. Journal of Clinical Microbiology, 42(7): 3317–3320.
DOI:10.1128/JCM.42.7.3317-3320.2004
|
Hadfield S J, Guy R, Kristin E, et al. 2011. Detection and differentiation of Cryptosporidium spp.in human clinical samples by use of Real-Time PCR[J]. Journal of Clinical Microbiology, 49(3): 918–924.
DOI:10.1128/JCM.01733-10
|
Harris J R, Petry F. 1999. Cryptosporidium parvum:structural components of the oocyst wall[J]. Journal of Parasitology, 85(5): 839–849.
DOI:10.2307/3285819
|
Hashimoto A, Kunikane S, Hirata T. 2002. Prevalence of Cryptosporidium oocysts and giardia cysts in the drinking water supply in Japan[J]. Water Research, 36(3): 519–526.
|
Helmi K, Skraber S, Burnet J, et al. 2011. Two-year monitoring of Cryptosporidium parvum and Giardia lamblia occurrence in a recreational and drinking water reservoir using standard microscopic and molecular biology techniques[J]. Environ Monit Assess, 179: 163–175.
DOI:10.1007/s10661-010-1726-7
|
Jothikumar N, Silva A J D, Moura I, et al. 2008. Detection and differentiation of Cryptosporidium hominis and Cryptosporidium parvum by dual Taqman assays[J]. Journal of Medical Microbiology, 57: 1099–1105.
DOI:10.1099/jmm.0.2008/001461-0
|
King D N, Donohue M J, Vesper S J, et al. 2016. Microbial pathogens in source and treated waters from drinking water treatment plants in the United States and implications for human health[J]. Science of the Total Environment, 562: 987–995.
DOI:10.1016/j.scitotenv.2016.03.214
|
Lemos V, Graczy T, Alves M, et al. 2005. Identification and determination of the viability of Giardia lambila cysts and Cryptosporidium parvum and Cryptosporidium hominis oocysts in human fecal and water supply samples by fluorescent in situ hybridization (FISH) and monoclonal antibodies[J]. Parasitology Research, 98: 48–53.
DOI:10.1007/s00436-005-0018-6
|
Li P, Cai J, Cai M, et al. 2016. Distribution of Cryptosporidium species in Tibetan sheep and yaks in Qinghai, China[J]. Veterinary Parasitology, 215: 58–62.
DOI:10.1016/j.vetpar.2015.11.009
|
Morgan U M, Constantine C C, Forbes D A, et al. 1997. Differentiation between human and animal isolates of Cryptosporidium parvum using rDNA sequencing and direct PCR analysis[J]. Journal of Parasitology, 83: 825–830.
DOI:10.2307/3284275
|
Nichols R A B, Campbell B M, Smith H V. 2003. Identification of Cryptosporidium spp.oocysts in United Kingdom Noncarbonated natural mineral waters and drinking waters by using a modified nested PCR-restriction fragment length polymorphism assay[J]. Applied and Environmental Microbiology, 69(7): 4183–4189.
DOI:10.1128/AEM.69.7.4183-4189.2003
|
Quintero-Betancourt W. 2003 Assessment of current and new methods for investigating the occurrence of enteric pathogens in ambient waters and water sources for rehydration of a Florida wetland[D]. Florida: University of South Florida
|
王明星, 柏耀辉, 梁金松, 等. 2016. 应用FCM-qPCR方法定量检测水中常见病原体[J]. 环境科学, 2016, 37(1): 384–390.
|
Xiao L, Alderisio K, Jiang J. 2006. Detection of Cryptosporidiumo oocysts in water:effect of the number of samples and analytic replicates on test results[J]. Applied and Environmental Microbiology, 72(9): 5942–5947.
DOI:10.1128/AEM.00927-06
|
Yang R, Jacobson C, Gardner G, et al. 2014. Longitudinal prevalence, oocyst shedding and molecular characterisation of Cryptosporidium species in sheep across four states in Australia[J]. Veterinary Parasitology, 200(1/2): 50–58.
|
余素华, 陈贻球, 严锦玲. 2009. 免疫荧光法测定水中贾第鞭毛虫孢囊和隐孢子虫卵囊[J]. 环境监测管理与技术, 2009, 21(1): 33–35.
DOI:10.3969/j.issn.1006-2009.2009.01.010 |
张冬青, 李红岩, 李栋, 等. 2009. 密度梯度分离纯化/免疫荧光技术检测饮用水中"两虫"[J]. 中国给水排水, 2009, 25(2): 78–83.
DOI:10.3321/j.issn:1000-4602.2009.02.022 |
 2018, Vol. 38
2018, Vol. 38


