养殖的规模化发展实现了高效养殖,是畜牧业发展的趋势.同时,规模化养殖也导致大量的养殖粪污集中产生,造成严重环境压力(苏杨,2006;Leconte et al., 2011).采用微生物发酵的方式处理禽畜粪便,以农业副产品为垫料层,禽畜养殖于其上,垫料中的微生物对粪污进行发酵,实现了养殖过程的零排放,缓解规模化养殖造成的环境问题(蓝江林等,2010;蒋建明等,2013).
养猪微生物发酵床垫料具有丰富的营养构成,是一种复杂的微生态环境,表现出很高的微生物多样性.其中的细菌多样性不仅受垫料成分的影响,而且猪肠道细菌也是其细菌的来源之一.刘波等(2008)采用微生物群落脂肪酸生物标记分析了零排放猪场基质垫层多样性,共检测出37个垫料微生物生物标记.宦海琳等(2014)采用PCR-DGGE技术对发酵床垫料基质中的细菌群落多样性进行研究,结果表明垫料中的主要菌群是节杆菌属、放线菌属、芽胞杆菌属和梭菌属等,垫料的组成影响其中的微生物群落结构.采用构建16S rDNA文库的方法,毕泗伟等(2013)发现养猪发酵床中存在大量不可培养微生物.笔者前期对垫料微生物多样性的研究发现,棒状杆菌属、芽孢杆菌属、枝芽孢杆菌属、假单胞菌属、放线菌属、乳杆菌属是垫料中的主要细菌属(Chen et al., 2017).发酵床垫料中的微生物能够有氧或厌氧发酵降解粪污中的有机物,细菌在降解过程中起重要作用(陈杰等,2014).如芽孢杆菌能够降解垫料有机质,参与猪粪降解与发酵床病原生防过程(赵国华等,2015;王震等,2015);乳酸菌能够促进纤维素的降解,抑制病原菌(陈世琼等,2002).
垫料中的微生物是发酵床降污的核心,其种类和数量对发酵床的运行至关重要,发酵床中的微生物随着垫料使用时间而发生变化.赵国华等(2015)研究发现随着发酵床寿命的延长,其中的微生物群落多样性逐渐降低.郑雪芳等(2016)微生物发酵床中细菌数量最大,数量随着垫料使用时间的增加呈先上升后下降的趋势.基于宏基因分析养猪发酵床微生物组随垫料使用时间的变化研究未见报道.因此, 本文采用Illumina二代测序技术,研究不同发酵等级垫料中细菌微生物组多样性.本研究可为发酵床垫料的维护及发酵床猪粪的生物降解提供依据.
2 材料与方法(Materials and methods) 2.1 材料采样地点的微生物发酵床位于福建省福清渔溪现代设施农业样本工程示范基地.采集时间为10、11和12月份.每月采用五点取样法采集微生物发酵床3个发酵等级的垫料样本,混合后进行后续分析.垫料的发酵等级是基于垫料的表观颜色、干湿度、气味和腐烂度进行区分,并经色差法标记(潘志针等,2016).轻度发酵垫料呈黄色,干燥,无臭味;中度发酵垫料呈棕黄色,略湿,轻微氨臭味;深度发酵垫料;呈现深棕色,湿度大,具有氨臭味.垫料由70%椰糠和30%谷壳粉构成,轻度发酵垫料使用时间短,呈黄色,干燥(水分含量约50%),10、11和12月份的样本分别命名为ⅰ1、ⅰ2和ⅰ3;中度发酵垫料呈棕黄色,略湿(水分含量约58%),样本名称分别为ⅱ1、ⅱ2和ⅱ3;深度发酵垫料使用时间长,呈深棕色,湿度大(水分含量约65%),样本名称分别为ⅲ1、ⅲ2和ⅲ3(图 1).
 |
| 图 1 轻度(a)、中度(b)和深度(c)发酵垫料示意图 Fig. 1 Mildly(a), moderately(b) and deeply(c) fermented litters |
每个样本称取500 mg垫料,按土壤DNA提取试剂盒FastDNA SPIN Kit for Soil的操作指南,分别进行总DNA的提取.调整终浓度至1 ng·μL-1开展后续试验.
2.2.2 微生物组16S rDNA V3~V4区测序采用引物338F (5′-ACT CCT ACG GGA GGC AGC AG-3′)和806R (5- GGA CTA CHV GGG TWT CTA AT-3′扩增细菌16S rDNA基因V3~V4区,PCR反应重复3次,取相同体积混合后进行目的片段回收.所用胶回收试剂盒为AxyPrepDNA凝胶回收试剂盒(Axygen公司).采用QuantiFluorTM -ST蓝色荧光定量系统(Promega公司)对回收PCR产物进行定量检测.然后构建插入片段为350 bp的paired-end(PE)文库(TruSeqTM DNA Sample Prep Kit建库试剂盒,Illumina公司),经过Qubit定量和文库检测,HiSeq上机测序(上海美吉).
2.2.3 微生物组测序数据质控与分析对测序得到的原始数据进行拼接、过滤,得到有效数据.采用Mothur软件(version 1.36.1)基于有效数据进行OTUs(Operational Taxonomic Units)聚类和物种分类分析(Schloss et al., 2009).采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析(Wang et al., 2007).从各个OTU中挑选出一条序列作为该OTU的代表序列,将该代表序列与已知物种的16S数据库(Silva,http://www.arb-silva.de)进行物种注释分析;根据每个OTU中序列的条数,得到各个OTU的丰度值(Quast et al., 2013;Oberauner et al., 2013).反映组间各样品之间的共有及特有OTU数目的Venn图,采用VennDiagram软件生成(Derrick et al., 2012).
采用QIIME软件的迭代算法(Wang et al., 2012)进行主成分分析(Principal Component Analysis,PCA).物种热图利用颜色梯度可以很好反映出样本在不同物种下的丰度大小以及物种聚类、样本聚类信息,可利用R语言的gplots包的heatmap.2函数实现(Jami et al., 2013).16S功能预测是采用PICRUSt(Phylogenetic investigation of communities by reconstruction of unobserved states)分析(Langille et al., 2013).
相应的数据释放与SRA (Sequence Read Archive)数据库, 序列号分别为SRR5611219(ⅰ1),SRR5611218(ⅰ2),SRR5611221(ⅰ3),SRR5611220(ⅱ1),SRR5611215(ⅱ2),SRR5611214(ⅱ3),SRR5611217(ⅲ1),SRR5611216(ⅲ2)和SRR5611223(ⅲ3).
3 结果(Results) 3.1 不同发酵等级垫料细菌多样性评估3个发酵等级9个样本共获得1198467有效序列,有效碱基数525997860 bp,序列平均长度438.8 bp.序列数和碱基数在不同发酵等级样本间差异显著,短序列数量最大值为中度发酵垫料ⅱ3(149292),最小值为深度发酵垫料ⅲ3(111463).比较3个发酵等级的样本,中度发酵垫料的序列数最多,分别是轻度发酵和深度发酵垫料样本的1.12和1.22倍,说明发酵等级影响垫料细菌多样性.如表 1所示,样本OTUs范围在1336(ⅰ3)~974(ⅱ2),深度发酵垫料OTU数目较多;反映群落丰度的Ace和Chao指数,范围分别在1502(ⅰ3)~1192(ⅱ2)和1567(ⅱ1)~1213(ⅱ2),比较得出轻度发酵垫料的Ace和Chao指数最高,说明微生物发酵床轻度发酵垫料细菌丰度最高.香农指数、辛普森指数反映了群落多样性,Shannon香农指数范围在5.5(ⅲ3)~3.9(ⅱ2),深度发酵垫料的该指数最高,中度发酵垫料最低.结果说明微生物发酵床深度细菌种类最多,中度发酵垫料细菌种类最少.
| 表 1 发酵床垫料微生物细菌群落宏基因组测序结果统计分析 Table 1 Similarity-based OTUs and species richness estimates of the bacterial phylotypes of the litters |
共检测到1839种OTU类型,包含33个门,73纲,149目,272科,600属和976种细菌.轻度发酵垫料包含29个门,64纲,137目,256科,567属和906种细菌,中度发酵垫料包含32个门,69纲,142目,259科,563属和898种细菌,深度发酵垫料包含33个门,70纲,141目,256科,559属和903种细菌.对比垫料样本中微生物群落的分析获得不同发酵等级在各分类水平上微生物种类以及各微生物的相对丰度.
3.2.1 微生物发酵床细菌门群落结构发酵等级差异比较不同等级垫料样本在细菌门水平上,共检测到33个细菌门.在3种发酵程度的垫料中,主要的优势门为Bacteroidetes(拟杆菌门,252169)、Proteobacteria(变形菌门,166869)、Actinobacteria(放线菌门,117541)、Firmicutes(厚壁菌门,113404)、Saccharibacteria(糖杆菌门88161)、Deinococcus-Thermus(异常球菌-栖热菌门,24214)、Spirochaetae(螺旋菌门,13685)、Chloroflexi(绿弯菌门,12618)和Tenericutes(柔膜菌门,11261).
在门水平上,有一未明确门分类的细菌差异显著,说明垫料中含有未被发掘的微生物资源.其次为放线菌门、变形菌门和柔膜菌门.放线菌门(26.7%)和绿弯菌门(2.4%)在轻度发酵垫料中含量最高,随着发酵的加深,在垫料中的含量逐级降低,在中度发酵垫料中的含量分别为9.6%、1.3%,在深度发酵垫料中的含量发别为6.8%、0.9%.变形菌门是中度发酵垫料的优势菌,在轻度发酵垫料中的含量为20.1%,发酵至中等程度,含量升至27.1%,随着发酵进行至深度发酵程度,含量降至15.8%.Saccharibacteria(糖杆菌门)在3种发酵程度样本中含量差别不大(图 2).
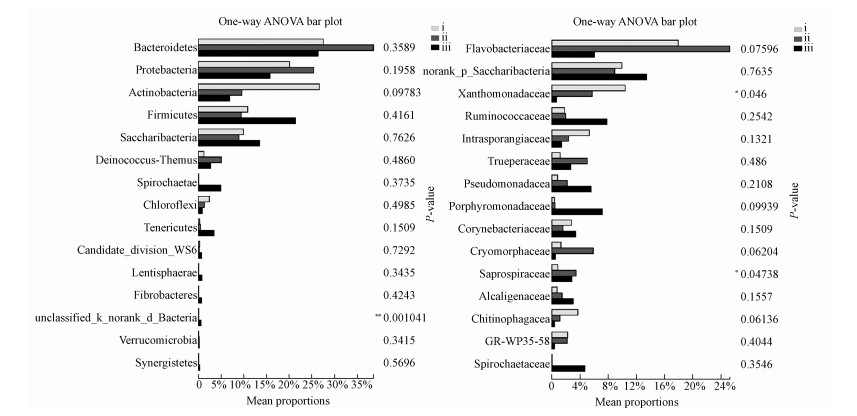 |
| 图 2 不同发酵等级垫料细菌在门(a)、科(b)水平上差异性分析 Fig. 2 Significance difference of bacteria in litters of different fermentation grade on the phylunm(a) and family(b) level |
微生物发酵床不同等级垫料样本共检测到272个科.主要为Flavobacteriaceae(黄杆菌科,133836)、未明确糖杆菌门一科(88066)、Xanthomonadaceae(黄单胞菌科,45703)、Ruminococcaceae(瘤胃菌科,31572)、Intrasporangiaceae(间孢囊菌科,24705)、Trueperaceae(特吕珀菌科,24214)、Pseudomonadaceae(假单胞菌科,23209)、Porphyromonadaceae(紫单胞菌科,21766)、Corynebacteriaceae(棒状杆菌科,21803)和Cryomorphaceae(20911).不同发酵等级的垫料在科水平上种类差异显著,并且细菌群落组成与垫料发酵程度相关.
黄单胞菌科和腐螺旋菌科在不同发酵等级垫料具有显著差异性(图 3).黄单胞菌科在轻度发酵垫料中含量最高(10.4%),随着发酵进行而逐级降低.间孢囊菌科在轻度发酵垫料中含量为5.3%,随着发酵进行降至2.4%(中度发酵垫料)和1.4%(深度发酵垫料).Chitinophagaceae在轻度、中度和深度垫料中的含量分别为3.7%、1.1%和0.4%.腐螺旋菌科在中度发酵垫料中含量最高(3.4%),在轻度(0.9%)和深度发酵垫料含量低(2.8%).Cryomorphaceae在轻度发酵垫料中的含量为1.3%,到中度发酵垫料升至5.9%,后又降至0.5%.黄杆菌科和特吕珀菌科具有同样的分布状态,在中度发酵垫料中含量高(25.2%、5.0%),在轻度(17.9%、1.2%)和深度发酵垫料(6.0%、2.7%)中含量低.瘤胃菌科(7.8%)、紫单胞杆菌科(7.1%)、假单胞菌科(5.5%)、Alcaligenaceae (产碱杆菌科,3.0%)和Spirochaetaceae(螺旋体科,4.7%)在深度发酵垫料中含量最高.尤其是瘤胃菌科、紫单胞杆菌科和螺旋体科.瘤胃菌科在深度发酵垫料中的含量是轻度发酵垫料的4.3倍.相比轻度发酵垫料,深发酵垫料中的紫单胞菌科和螺旋体科分别升高了17.8倍、78.3倍.Saccharibacteria门中的一科在3种垫料中差异不明显(图 3~图8).
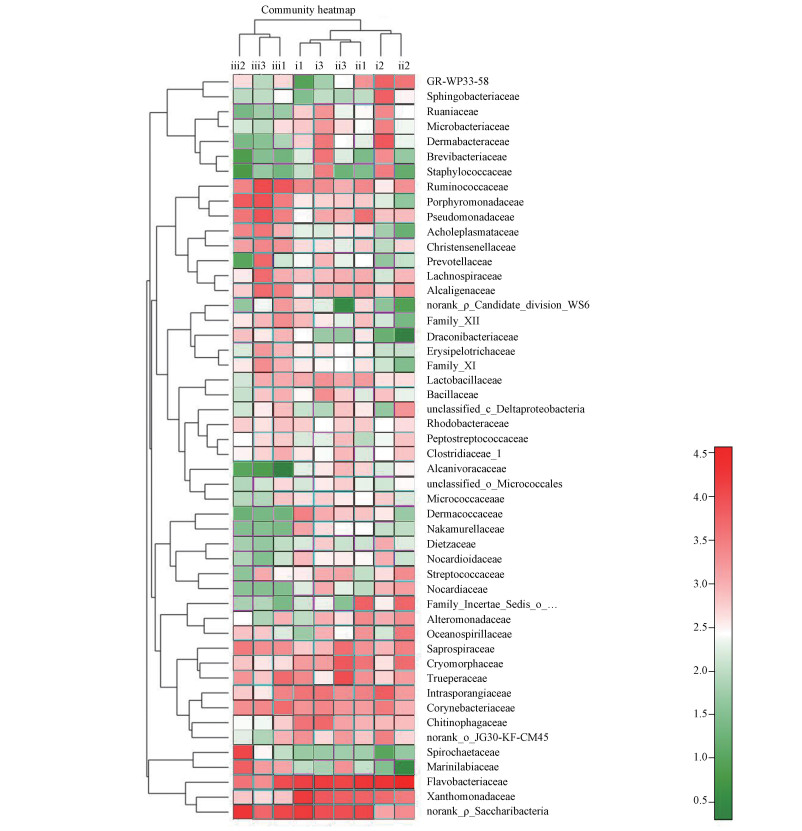 |
| 图 3 细菌在各样本间的分布热图(科水平) Fig. 3 Hierarchically clustered heatmap of bacterial distribution of different communities from the samples on the family level |
根据对不同分类水平的微生物多样性分析,根据发酵床不同发酵等级样本丰度前35的细菌科丰度比例结构,建立热图(图 3).根据细菌丰度差异,可聚成5类.第1类在深发酵等级样本ⅲ2和ⅲ3中含量高于其它样本,细菌为Spirochaetaceae(螺旋体科)和Marinilabiaceae(海滑菌科).第2类在所有样本中含量都很高,包括黄杆菌科、黄单胞杆菌科和未明确的糖杆菌门一科.第3类为浅发酵垫料ⅰ2中高含量菌,包括GR-WP33-58、Sphingobacteriaceae(鞘脂杆菌科)、Dermabacteraceae(皮杆菌科)、瘤胃菌科、Microbacteriaceae(微杆菌科)、Brevibacteriaceae(短杆菌科)和Staphylococcaceae(葡萄球菌科),其中Dermabacteraceae(皮杆菌科)、Ruaniaceae(阮氏菌科)、Microbacteriaceae(微杆菌科)、Brevibacteriaceae(短杆菌科)和Staphylococcaceae(葡萄球菌科)在浅发酵垫料样本ⅰ3中含量很高,轻度发酵垫料样本中,这5个科的细菌相对含量均高于中度和深度发酵垫料.第4类细菌在浅发酵垫料ⅰ2中含量低于其它样本,包括瘤胃菌科、Porphyromonadaceae(紫单胞杆菌科)和假单胞菌科等8科细菌.这些菌在轻度发酵样本ⅰ2、ⅰ3和中度发酵样本ⅱ2、ⅱ3中的相对含量也很低.第5类在不同等级样本间分布差异低于前4类样本.
不同发酵等级的垫料样本可聚成2大类,第1类为深度发酵垫料(ⅲ1、ⅲ2和ⅲ3),第2类为轻度和中度发酵垫料(ⅰ1、ⅰ2和ⅰ3;ⅱ1、ⅱ2和ⅱ3).第2类又可细分为3个亚类:轻度发酵样本ⅰ1和ⅰ3;中度发酵垫料ⅱ1和ⅱ3;垫料ⅰ2和ⅱ2.说明垫料的发酵等级与微生物群落结构相关,相同发酵等级的垫料在一定程度上有相似的细菌群落结构.但由于发酵是连续的过程,人为判别的发酵等级,在不同发酵程度间有样本的重叠,导致相同发酵等级垫料具有不同的细菌群落结构,相邻发酵等级的垫料具有相似的细菌群落结构.
3.4 微生物发酵床细菌群落RDA关联分析RDA结果显示,厚壁菌门、Saccharibacteria、螺旋菌门、异常球菌-栖热菌门与发酵等级呈锐角,与垫料发酵程度呈正相关(图 4).这些细菌在在深度发酵垫料中含量高于低发酵程度的垫料.变形菌门、拟杆菌门和放线菌门与发酵程度呈现钝角,与发酵程度负相关.放线菌门在轻度发酵垫料中含量最高.变形菌门和拟杆菌门在中度发酵垫料中含量较高.
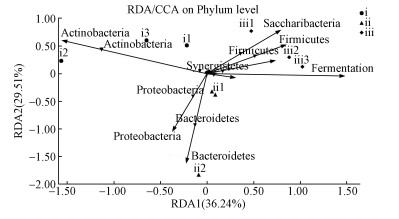 |
| 图 4 垫料微生物与发酵等级的RDA分析 Fig. 4 Redundancy analysis (RDA) of the relationship betweenbacteria and fermentation level |
PICRUSt预测3个发酵等级垫料中细菌的主要有机物降解途径,结果表明随着垫料等级的递增,细菌群落对氨基酸、碳水化合物和脂类代谢相关基因的拷贝数呈现递减趋势.轻度发酵垫料(发酵程度ⅰ级)中,细菌碳水化合物、脂类和氨基酸的代谢基因的拷贝数分别为6145878、2128199和6400389,而中度发酵垫料中的相应值为4808014、1786265和5379729,相对于轻度发酵垫料,深度发酵垫料的相应基因拷贝数降低了31.8%、37.8%和31.7%.根据PICRUSt的预测信息,发酵床轻度发酵垫料中细菌组的有机物代谢基因数量最高,随着垫料等级的增加,其代谢能力降低.这与赵国华等(2015)研究结果一致.
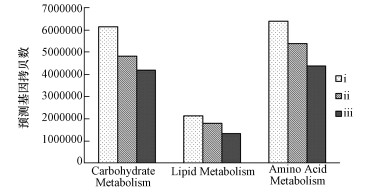 |
| 图 5 发酵床不同发酵程度垫料中细菌群落氨基酸、碳水化合物和脂类代谢相关基因的分布 Fig. 5 Functional prediction of bacterial community in amino acid, carbohydrate and lipid metabolism in litters of different fermentation grade |
养殖业的迅猛发展导致大量养殖废弃物产生,微生物发酵床技术分解消纳粪便,是一种环保型的养殖方式.本研究采用高通量测序技术研究了微生物发酵床3个发酵等级垫料的细菌多样性,发现垫料的发酵等级与微生物群落相关,不同发酵等级的垫料具有不同的细菌多样性和群落构成,相同发酵等级如深度发酵垫料,轻度发酵样本ⅰ1和ⅰ3,中度发酵垫料ⅱ1和ⅱ3具有相似的细菌群落.但由于垫料的发酵是连续的过程,不同发酵等级的垫料样本之间有重合,如浅发酵垫料ⅰ2和ⅱ2.
在微生物发酵床垫料与猪粪的混合发酵过程中,不同发酵等级起作用的降解菌不同,细菌含量呈波动变化.浅发酵垫料中的黄单胞杆菌科和间孢囊菌科在发酵初期含量高,是这一时期的主要降解菌.黄单胞杆菌科属于变形菌门,在蚯蚓堆肥系统中起降解作用(Jin et al., 2007; Lin et al., 2016).间孢囊菌科属于放线菌门,此科的某些细菌可分泌碱性纤维素酶,降解纤维素(侯晓娟等,2007),表层垫料中的间孢囊菌科细菌可能参与猪粪及垫料中的纤维素降解.在中度发酵垫料中,变形菌门、拟杆菌门和异常球菌-栖热菌门的含量到达峰值.变形菌门的腐螺旋菌科、拟杆菌门黄杆菌科、放线菌门的棒状杆菌科和异常球菌-栖热菌门的特吕珀菌科在此种垫料中含量最高.腐螺旋菌科可以降解活性污泥中的蛋白质(Yun et al., 2008).黄杆菌科属于拟杆菌门,该科细菌在堆肥中起着重要的作用,该科的很多细菌能够抵御抗生素胁迫,并能降解有机物,是堆肥中的常见菌(Bernardet, 2002),许晓毅等(2015)研究同样表明黄杆菌科具有降解有机物的作用,在环境修复中具有重要应用,因此微生物发酵床中的该科细菌可能参与发酵床猪粪的降解.特吕珀菌科属于异常球菌目,能在碱、盐及高温的条件中生长,在堆肥过程中产生固氮酶,参与固氮过程(Li et al., 2015),同时可利用多种糖类、有机酸和氨基酸(Ivanova et al., 2011).棒状杆菌科细菌是高有机质含量堆肥系统中的主要细菌,参与粪便降解(Silva et al., 2016).在深度发酵垫料中,瘤胃菌科、紫单胞杆菌科、产碱菌科和假单胞菌科含量高于轻度发酵垫料.紫单胞杆菌科属于拟杆菌门,Hahnke等(2016)自沼气反应器中分离了该科的细菌,参与粪便降解.假单胞菌科细菌含量随发酵升高,这一结果与Wang等(2015)的研究一致.假单胞菌科分布广泛,许多细菌具有很强的降解能力,包括降解油脂、烷烃、酚类和有机氮(秦华明等,2003;李军冲等,2010;赵鑫等,2012).产碱菌科的粪产碱菌可抑制大肠杆菌和金黄色葡萄球菌等病原菌(丁友真和张书芳,1997),这可能是致病菌随发酵含量降低的原因.中度和深度发酵垫料中的蛋白质、多糖、有机酸等有机物降解菌含量增加,这与发酵床中猪粪的降解相关.
同时,本研究发现浅发酵垫料中,皮杆菌科和葡萄球菌科含量均高于中度和深度发酵垫料.垫料中微杆菌科细菌可能参与猪粪的生物降解,提高发酵床降解效率.皮杆菌科的某些物种是条件致病菌,导致皮肤感染(Renvoise et al., 2009),垫料中的皮杆菌科细菌可能引起猪皮肤病.在轻度、中度和深度发酵垫料中,此科的平均序列数分别为3933、222和45.随着发酵的进行,含量降低了87倍.葡萄球菌科的某些微生物也是条件致病菌,杨鼎等(2009)研究发现葡萄球菌能引起仔猪急性传染病,包括皮炎、腹泻等.随着发酵的进行,葡萄球菌科细菌的含量降低了100多倍.皮杆菌科和葡萄球菌科细菌数量的变化揭示微生物发酵床对病原菌具有防治作用,保护猪的健康.
3个发酵等级垫料中细菌的主要有机物降解途径预测结果显示:随着垫料等级的递增,细菌群落对氨基酸、碳水化合物和脂类的代谢相关基因的拷贝数呈现递减趋势.同时,本研究发现随着垫料等级的增加,瘤胃菌细菌数量逐渐增高;瘤胃菌科细菌是新鲜猪粪中最主要的细菌,Wang等(2015)发现随着猪粪有机质的降解,瘤胃菌科细菌含量逐渐降低.基于代谢途径预测和瘤胃菌变化趋势,本文推测随着垫料发酵等级的升高,其中的微生物对猪粪的降解能力降低.
5 结论(Conclusions)本研究采用宏基因组技术研究了了微生物发酵床不同发酵等级垫料的细菌组成,揭示了细菌多样性与垫料发酵程度的关系.在3个发酵阶段,微生物发酵床中起主要降解作用的细菌不同.在轻度发酵时期,黄单胞杆菌科和间孢囊菌科细菌是主要的降解菌;在中度发酵时期,变形菌门的腐螺旋菌科、拟杆菌门的黄杆菌科、放线菌门的棒状杆菌科和异常球菌-栖热菌门的特吕珀菌科起主要降解作用;在深度发酵时期,厚壁菌门梭菌目瘤胃菌科、拟杆菌门的紫单胞杆菌科、变形菌门的产碱菌科和假单胞菌科起降解作用.随着垫料等级的增加,粪便中的指示菌-瘤胃菌含量增加,垫料中微生物群落的代谢能力降低.同时,随着发酵等级的增加,垫料中的葡萄球菌科和皮杆菌科细菌含量减少,说明发酵床可以抑制致病菌,保护养殖健康.
Bernardet J F. 2002. International committee on systematics of prokaryotes:Subcommittee on the taxonomy of Flavobacterium and Cytophaga-like bacteria[J]. International Journal of Systematic and Evolutionary Microbiology, 52(3): 1071–1073.
DOI:10.1099/ijs.0.02137-0
|
毕泗伟, 吴祖芳, 虞耀土. 2013. 16S rDNA基因文库技术分析发酵床细菌群落的多样性[J]. 宁波大学学报(理工版), 2013(1): 18–22.
|
Chen Q, Liu B, Wang J, et al. 2017. Diversity and dynamics of the bacterial community involved in pig manure biodegradation in a microbial fermentation bed system[J]. Annals of Microbiology, 67(7): 491–500.
DOI:10.1007/s13213-017-1278-y
|
陈杰, 赵祥杰, 邝哲师, 等. 2014. 利用微生物处理畜禽粪便的研究[J]. 安徽农业科学, 2014, 10(28): 9910–9911.
DOI:10.3969/j.issn.0517-6611.2014.28.097 |
陈世琼, 李平兰, 张篪. 2002. 猪肠道中抑制大肠杆菌的乳酸菌的分离与筛选[J]. 中国农业大学学报, 2002, 7(5): 117–120.
DOI:10.3321/j.issn:1007-4333.2002.05.022 |
丁友真, 张书芳. 1997. 家蝇幼虫体内的粪产碱菌有抑菌能力[J]. 中国媒介生物学及控制杂志, 1997, 8(3): 181–183.
|
侯晓娟, 王卫卫, 李忠玲, 等. 2007. 一株产碱性纤维素酶放线菌的分离及酶学特性[J]. 西北大学学报自然科学版, 2007, 37(5): 781–784.
|
Hahnke S, Langer T, Koeck D E, et al. 2016. Description of Proteiniphilum saccharofermentans sp. nov. Petrimonas mucosa sp. nov. and Fermentimonas caenicola gen. nov. sp. nov. isolated from mesophilic lab-scale biogas reactors and emended description of the genus Proteiniphilum[J]. International Journal of Systematic and Evolutionary Microbiology, 66(3): 1466–1475.
DOI:10.1099/ijsem.0.000902
|
宦海琳, 闫俊书, 周维仁, 等. 2014. 不同垫料组成对猪用发酵床细菌群落的影响[J]. 农业环境科学学报, 2014, 33(9): 1843–1848.
|
Ivanova N, Rohde C, Munk C, et al. 2011. Complete genome sequence of Truepera radiovictrix type strain (RQ-24 T)[J]. Standards in Genomic Sciences, 4(1): 91–92.
DOI:10.4056/sigs.1563919
|
Jami E, Israel A, Kotser A, et al. 2013. Exploring the bovine rumen bacterial community from birth to adulthood[J]. Isme Journal, 7(6): 1069.
DOI:10.1038/ismej.2013.2
|
Jin L, Kim K K, Im W T, et al. 2007. Aspromonas composti gen. nov. sp. nov. a novel member of the family Xanthomonadaceae[J]. International Journal of Systematic and Evolutionary Microbiology, 57(8): 1876–1880.
DOI:10.1099/ijs.0.64472-0
|
Langille M G I, Zaneveld J, Caporaso J G, et al. 2013. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 31(9): 814–821.
DOI:10.1038/nbt.2676
|
蒋建明, 闫俊书, 白建勇, 等. 2013. 微生物发酵床养猪模式的关键技术研究与应用[J]. 江苏农业科学, 2013, 41(9): 173–176.
DOI:10.3969/j.issn.1002-1302.2013.09.069 |
Leconte M C, Mazzarino M J, Satti P, et al. 2011. Nitrogen and phosphorus release from poultry manure composts:the role of carbonaceous bulking agents and compost particle sizes[J]. Biology and Fertility of Soils, 47(8): 897–906.
DOI:10.1007/s00374-011-0591-z
|
Li X, Bond P L, Van Nostrand J D, et al. 2015. From lithotroph-to organotroph-dominant:directional shift of microbial community in sulphidic tailings during phytostabilization[J]. Scientific Reports, 5: 12978.
DOI:10.1038/srep12978
|
Lin Z, Bai J, Zhen Z, et al. 2016. Enhancing pentachlorophenol degradation by vermicomposting associated bioremediation[J]. Ecological Engineering, 87(3): 288–294.
|
蓝江林, 刘波, 唐建阳, 等. 2010. 基于微生物发酵床养猪模式的生态安全探讨[J]. 中国农学通报, 2010, 26(19): 324–326.
|
李军冲, 齐树亭, 石玉新, 等. 2010. 一株假单胞菌降解溶解有机氮条件探讨[J]. 食品研究与开发, 2010, 31(5): 151–153.
DOI:10.3969/j.issn.1005-6521.2010.05.045 |
刘波, 郑雪芳, 朱昌雄, 等. 2008. 脂肪酸生物标记法研究零排放猪舍基质垫层微生物群落多样性[J]. 生态学报, 2008, 28(11): 5488–5498.
DOI:10.3321/j.issn:1000-0933.2008.11.033 |
潘志针, 刘波, 朱育菁, 等. 2016. 基于色差技术的微生物发酵床垫料发酵等级研究[J]. 福建农业学报, 2016, 31(11): 1248–1251.
|
秦华明, 王菊芳, 朱明军, 等. 2003. 假单胞菌降解含油脂废水的研究[J]. 中国油脂, 2003, 28(11): 75–77.
DOI:10.3321/j.issn:1003-7969.2003.11.023 |
Renvoise A, Aldrovandi N, Raoult D, et al. 2009. Helcobacillus massiliensis gen. nov. sp. nov. a novel representative of the family Dermabacteraceae isolated from a patient with a cutaneous discharge[J]. International Journal of Systematic and Evolutionary Microbiology, 59(9): 2346.
DOI:10.1099/ijs.0.003319-0
|
Schloss P D, Westcott S L, Ryabin T, et al. 2009. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied Environmental Microbiology, 75(23): 7537–7541.
DOI:10.1128/AEM.01541-09
|
Silva M E, Lopes A R, Cunhaqueda A C, et al. 2016. Comparison of the bacterial composition of two commercial composts with different physicochemical, stability and maturity properties[J]. Waste Management, 50: 20–30.
DOI:10.1016/j.wasman.2016.02.023
|
苏杨. 2006. 我国集约化畜禽养殖场污染问题研究[J]. 中国生态农业学报, 2006, 14(2): 15–18.
|
Wang H, Li H, Gilbert J A, et al. 2015. Housefly larva vermicomposting efficiently attenuates antibiotic resistance genes in swine manure, with concomitant bacterial population changes[J]. Applied and Environmental Microbiology, 81(22): 7668–7679.
DOI:10.1128/AEM.01367-15
|
Wang Q, Garrity G, Tiedje J, et al. 2007. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied & Environmental Microbiology, 73(16): 5261–5267.
|
王震, 尹红梅, 刘标, 等. 2015. 发酵床垫料中优势细菌的分离鉴定及生物学特性研究[J]. 浙江农业学报, 2015, 27(1): 87–91.
DOI:10.3969/j.issn.1004-1524.2015.01.16 |
许晓毅, 苏攀, 姬宇, 等. 2015. 沉积物中2株多环芳烃降解菌的分离鉴定及其对菲、荧蒽的降解特性[J]. 环境工程学报, 2015, 9(3): 1513–1520.
|
Yun X, Yunhong K, Trine R, et al. 2008. Identification and ecophysiological characterization of epiphytic protein-hydrolyzing Saprospiraceae ("Candidatus Epiflobacter" spp.) in activated sludge[J]. Applied and Environmental Microbiology, 74(7): 2229–2238.
DOI:10.1128/AEM.02502-07
|
杨鼎, 郝永清, 宋长绪. 2009. 猪葡萄球菌脱落毒素研究进展[J]. 动物医学进展, 2009, 30(4): 60–64.
DOI:10.3969/j.issn.1007-5038.2009.04.016 |
赵国华, 方雅恒, 陈贵. 2015. 生物发酵床养猪垫料中营养成分和微生物群落研究[J]. 安徽农业科学, 2015, 60(8): 98–99.
DOI:10.3969/j.issn.0517-6611.2015.08.039 |
赵鑫, 韩妍, 梁文, 等. 2012. 铜绿假单胞菌生物降解特性的研究进展[J]. 基因组学与应用生物学, 2012, 31(4): 406–414.
DOI:10.3969/gab.031.000406 |
郑雪芳, 刘波, 朱育菁, 等. 2016. 养猪发酵床垫料微生物及其猪细菌性病原群落动态的研究[J]. 农业资源与环境学报, 2016, 33(5): 425–432.
|
 2018, Vol. 38
2018, Vol. 38


