多环芳烃(polycyclic aromatic hydrocarbons, PAHs)是由2个或2个以上苯环以线状、角状或簇状排列组合成的一类稠环化合物(秦宁等, 2013; 平立凤等, 2005).PAHs具有高毒、环境持久和长距离迁移等特性, 可在生物体内富集并通过食物链放大, 危害人体和整个生态系统, 被列入《斯德哥尔摩公约》首批控制的持久性有机污染物之一(吴艳阳等, 2013; Jacques et al., 2008).多环芳烃(PAHs)污染修复是当前环境修复中研究的热点, 生物降解是对PAHs污染土壤进行修复的重要途径之一(Poretsky et al., 2005; 尹春芹等, 2012).
对PAHs降解菌的降解机理研究大部分都是从分子生物学水平对微生物的基因及基因簇进行了研究(Michiei et al., 2004), 而从比较转录组的测序与分析以揭示其降解性能的研究工作还较少.随着遗传组成的不同或者降解环境条件的改变, 微生物的代谢网络会发生相应的调节和变化(Seo et al., 2009).若想获得具有稳定PAHs降解效率的菌株, 应该从菌株的整体代谢网络变化来评估, 而转录组学分析技术的引入应用, 可以很大程度上帮助我们实现这种设想(Sadat et al., 2011).通过分析转录组, 可高通量地获得基因表达的RNA水平有关信息.本文筛得一株降解PAHs的菌株克雷白氏杆菌(Klebsiella pneumoniae Tzyx1), 对其转录组进行测序并比较该菌在正常培养和PAHs胁迫培养这两种不同条件下基因表达的差异, 找到基因型与表型相关方式, 以揭示其降解PAHs路径和特性, 这些研究有利于PAHs微生物降解的生物化学和遗传学的研究, 有利于阐明芳香化合物的生物降解机制, 从而分离和培育出降解谱广和降解性能高的菌株, 用于环境污染物的生物治理.
2 材料与方法(Materials and methods) 2.1 菌株及培养处理克雷白氏杆菌(Klebsiellapneumoniae Tzyx1)(CCTCC M 2012239)由本实验室筛选自浙江省台州市某固体废弃物拆解场污染土壤, 并保存于中国典型培养物保藏中心.
葡萄糖-基础培养基:硫酸铵0.2%、柠檬酸钠0.1%、七水合硫酸镁0.02%、磷酸氢二钾0.4%、磷酸二氢钾0.6%、蛋白胨0.1%、葡萄糖0.2%、pH 7.2.
多环芳烃胁迫培养基:硫酸铵0.2%、柠檬酸钠0.1%、七水合硫酸镁0.02%、磷酸氢二钾0.4%、磷酸二氢钾0.6%、蛋白胨0.1%、pH 7.2;灭菌后加入芘、菲、苯并蒽、苯并芘混合多环芳烃化合物的丙酮溶液作为碳源和能源, 置于超净工作台紫外灭菌, 待丙酮挥发后用于菌种的筛选富集.上述加入芘、菲、苯并蒽、苯并芘的量要使配制好的培养液中芘、菲、苯并蒽、苯并芘的最终浓度均为200 mg·L-1.
Tzyx1首先在LB液体培养基中过夜培养至OD600=1, 离心收集菌体后分别重悬至等体积的葡萄糖-基础培养基与多环芳烃胁迫培养基中, 25 ℃, 培养72 h.离心收集菌体, 采用TRIzol法(杨宇虹等, 2006)提取总RNA.葡萄糖-基础培养基培养菌体总RNA样品命名为Tzy-x1c, 多环芳烃胁迫培养基培养菌体总RNA样品命名为Tzy-x1a.
2.2 c DNA文库构建和DNA测序以5 μg总RNA起始量建库, 实验采用IlluminaTruseqTM RNA sample prep Kit试剂盒构建文库(Cock et al., 2010), 实验流程如下:磁珠法去除rRNA(Ribo-Zero Magnetic kit (G+/G-Bacteria)); 离子打断mRNA(TruseqTM RNA sample prep Kit); 双链cDNA合成、补平、3′端加A、连接index接头(Truseq TM RNA sample prep Kit); 文库富集, PCR扩增15个cycles; 2%琼脂糖胶回收目的条带(Certified Low Range Ultra Agarose); TBS380(Picogreen)定量, 按数据比例混合上机; cBot上进行桥式PCR扩增, 生成clusters; Hiseq2500测序平台, 进行2*100bp测序(Erlich et al., 2008).
2.3 转录组数据装配与分析测序后的数据经初步质控分析后, 与Klebsiellapneumoniae KCTC 2242基因组进行比对(GenBank NO. GCA_000220485.1)(Langmead et al., 2012), 比对好的基因信息分别在GO(Gene Ontology)(Trapnell et al., 2010)、COG(Cluster of Orthologous Groups of Proteins)(Mitchell et al., 2009)、KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库(Carlson, 2009)进行分析.
根据Tzy-x1a、Tzy-x1c与参考基因组的比对结果, 计算每个基因/转录本在样本中的FPKM值, 以该值作为基因/转录本在样本中的表达量(Trapnell et al., 2009; Trapnell et al., 2012).最后对所有基因/转录本在各组样本中的表达进行差异显著性分析(Tarazona et al., 2011), 找出相对差异表达的基因/转录本, 并对其进行可视化分析.将FDR控制Q≤0.05得到的表达差异基因进一步对其进行GO显著性分析与KEGG显著性分析.
2.4 荧光定量分析分别从Tzy-x1a、Tzy-x1c中提取总RNA. cDNA合成按照反转录试剂盒说明(Promega, 美国)进行, ABI 7500荧光定量仪用于荧光定量.
3 结果(Results) 3.1 克雷白氏杆菌转录组功能注释分析利用GO数据库, 可以将基因按照它们参与的生物学过程、构成细胞的组分, 实现的分子功能等进行分类.GO数据分类有助于理解物种的基因功能的分布, 从宏观层面描述基因产品的相关生物过程、细胞组分和分子功能(Tang et al., 2008).GO分析结果显示, Klebsiella pneumoniae Tzyx1 unigenes中, 参与生物途径、细胞的组分合成、分子功能因子合成的比例为51%、26%、23% (图 1所示).
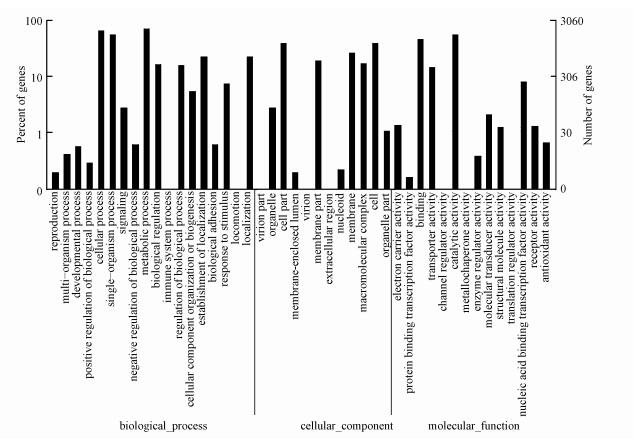 |
| 图 1 克雷白氏肺炎杆菌基因按照GO中分子功能、生物学过程和细胞组分的分类 Fig. 1 GO functional classification of annotated unigenes |
COGs的数据库是根据细菌、藻类、真核生物的完整基因组基因产物进行直系同源分类的数据库, 将K. pneumoniae Tzyx1 unigenes与COG数据库进行比对, 注释结果显示在图 2中.结果表明, 有514条unigenes参与了碳氢化合物的运输与降解.利用KEGG数据库(Grabherr et al., 2011; Camacho et al., 2009)对unigenes进行产物功能及代谢通路分析, 进一步得到pathway-based注释和分析.K. pneumoniae Tzyx1 unigenes中大概有440条unigenes参与了碳氢化合物的代谢与含有苯环结构的异源生物质的生物降解.其参与的苯环结构的异源生物质的生物降解途径包括Benzoate degradation (PATH ko00362), Aminobenzoate degradation (PATH ko00627), Fluorobenzoate degradation (PATH ko00364), Chlorocyclohexane and chlorobenzene degradation (PATH ko00361), Toluene degradation (PATH ko00623), Xylene degradation (PATH ko00622), Nitrotoluene degradation (PATH ko00633), Ethylbenzene degradation (PATH ko00642), Styrene degradation (PATH ko00643), Caprolactam degradation (PATH ko00930), Dioxin degradation (PATH ko00621), Naphthalene degradation (PATH ko00626), Polycyclic aromatic hydrocarbon degradation (PATH ko00624), Metabolism of xenobiotics by cytochrome P450 (PATH ko00980), Drug metabolism-cytochrome P450 (PATH ko00982), Drug metabolism-other enzymes (PATH ko00983).
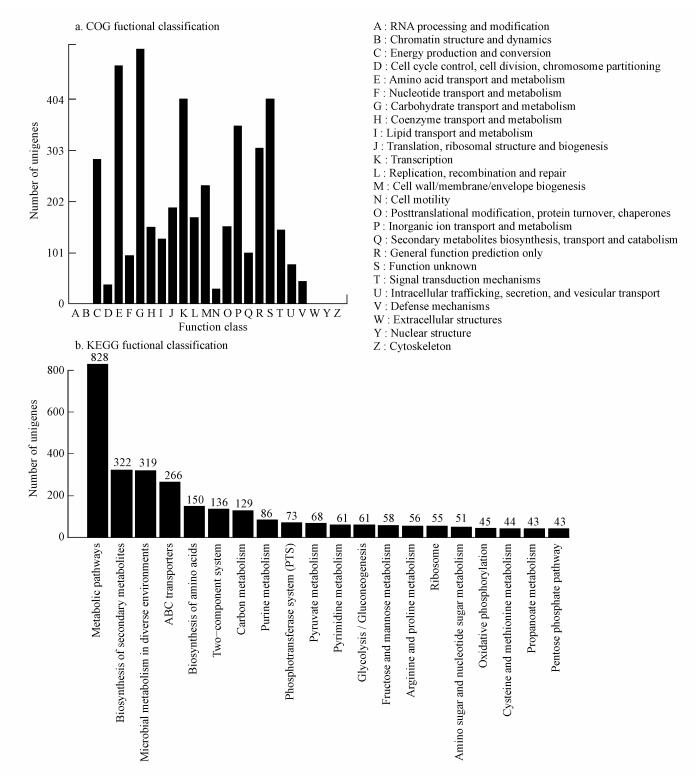 |
| 图 2 COGa和KEGGb分类统计图 (注:a图中每一种柱子代表一个COG的功能分类(用大写字母A~Z表示, 具体含义见右侧标注), 柱子的高度即纵坐标表示具有该类功能的isogene数目COGs的数据库是系统完整的基因组蛋白质编码的分类(ref), 并将其应用于unigenes的功能注释的任务.是显示在图 2a中, 4924注释unigenes显示显著的相似之处26功能类别.b图中从左至右按照包含isogenes数目从高到低依次排列, 柱子越高表明该生物学通路在所测样本中越活跃, 可以根据研究目的的不同选择排序靠前的通路进行后续深入分析.) Fig. 2 COG (a) and KEGG (b) functional classification of annotated unigenes |
Tzy-x1a与Tzy-x1c的转录组数据中找到254个差异表达基因(FDR≤0.05), 占Klebsiellapneumoniae Tzyx1基因组中5264个总基因的5%.在这些差异基因中, 有73个在多环芳烃为碳源时上调表达, 181个下调表达.
根据差异基因的GO富集结果, 其中参与生物途径、细胞的组分合成、分子功能因子合成的差异表达基因比例为43%、38%、20%.使用软件Goatools (https://github.com/tanghaibao/GOatools)进行富集分析, 将差异基因按照其功能进行GO分类.图 3所示为差异表达基因在不同的通路中富集结果, 由图可知, 差异基因在small ribosomal subunit(GO:0015935)富集率最高, 接下来较高的有structural constituent of ribosome(GO:0003735)、cellular component biogenesis(GO:0044085)、ribonucleoprotein complex biogenesis(GO:0022613)等.
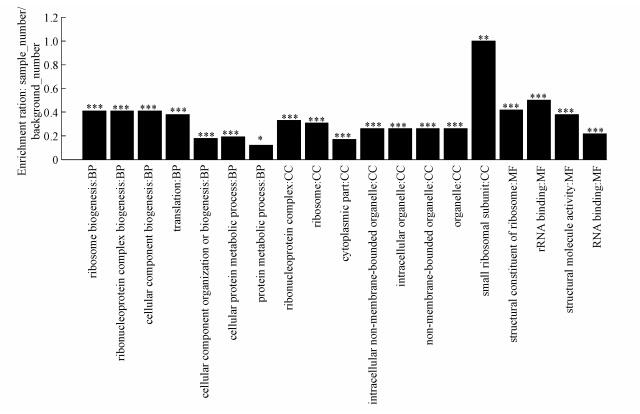 |
| 图 3 GO(Gene Ontology)富集分析散点图(Tzy-x1c vs Tzy-x1a)c (注:图中每个柱子为一个通路, 横坐标文字表示通路的名称和分类, 所属分类描述如左上角所示.柱子的高度即纵坐标表示富集率.显著性即富集率用P表示, 其中P < 0.01的标记为***, P < 0.01的标记为**, P < 0.5的标记为*.) Fig. 3 Scatter plot of the enrichment in the Gene Ontology (GO) analysis (Tzy-x1c vs. Tzy-x1a) |
差异表达基因所属KEGG PATHWAY类型进行分析(Xie et al., 2011), 结果如图 4所示.在Metabolism大类中, 除了“Metabolism of terpenoids and polyketides”“Glycan biosynthesis and metabolism”只存在下调基因外, 其他均存在不同数量的上调基因与下调基因; 在Genetic Information Processing大类中, 所有差异表达基因均为下调, 这是因为多环芳烃胁迫条件会对细胞膜的完整性有影响.Environmental Information Processing与Metabolism类似, 所属通路均存在不同数量的上调基因与下调基因; Organismal Systems与Cellular Processes则各存在一个上调表达基因.
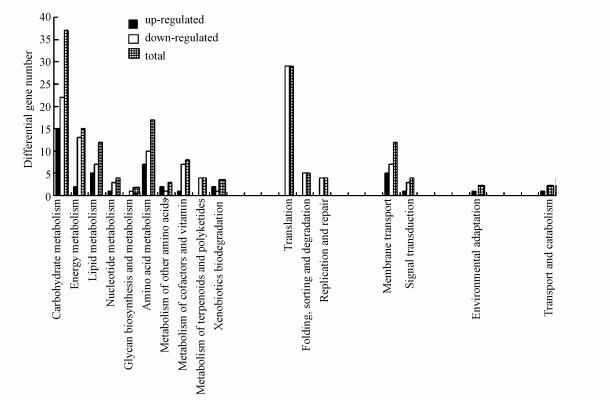 |
| 图 4 K. pneumoniae Tzyx1差异基因按照KEGG数据库所参与的PATHWAY类型进行的分类 Fig. 4 KEGG functional classification of K. pneumoniae Tzyx1 genetic variations |
在转录组中找到254个差异表达基因用荧光定量的方法进行研究, 这些基因来自转录后细胞对外界改变的调控机制、能量供应、基因复制、损伤修复(包括核糖体蛋白、嘌呤和嘧啶代谢)和碳氮代谢等, 将表达模式相近的各组基因在各个样本中的表达趋势用曲线表示, 结果如图 5所示.K. pneumoniae Tzyx1细胞在胁迫状态下乙酰辅酶A合成酶(acetyl-CoA synthetase)、多环芳烃1, 2双加氧酶(benzoate/toluate 1, 2-dioxygenase)及相应的脱氢酶(dihydroxycyclohexadiene carboxylate dehydrogenase)、单加氧酶(p-hydroxybenzoate 3-monooxygenase)、原儿茶酸3, 4-双加氧酶(protocatechuate 3, 4-dioxygenase)相对表达量明显高于Tzy-x1c实验组.根据注释的结果, K. pneumoniae Tzyx1至少存在2种完整的KEGGpathway记录的苯环开环的方式, 第1种是在多环芳烃1, 2-双加氧酶及相应的脱氢酶的作用下生成邻苯二酚, 邻苯二酚进一步在儿茶酚1, 2-双加氧酶的作用下内开环生成粘康酸, 进而经过一系列的反应代谢为乙酰辅酶A和琥珀酰辅酶A, 进入三羧酸循环.第2种是在单加氧酶的作用下生成原儿茶酸, 之后在原儿茶酸3, 4-双加氧酶(的作用下内开环生成3-羧基粘康酸, 进而经过一系列的反应代谢为乙酰辅酶A和琥珀酰辅酶A, 进入三羧酸循环.
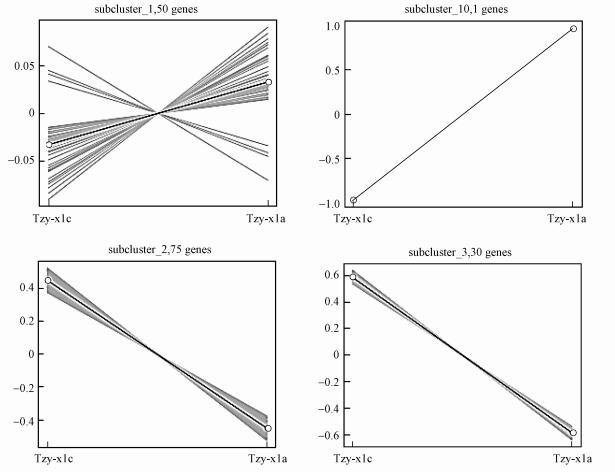 |
| 图 5 差异基因表达趋势线(Tzy-x1c vs x1a) (注:图中为每个子cluster的表达趋势折线图, 横坐标为各比较样本组, 纵坐标为基因在该组样本中的表达量.图中每一条线表示一个基因, 黑色的线表示该子cluster中所有基因的表达量平均值; 每张图展示一种类型的表达模式, 即体现这组基因表达量变化的趋势.) Fig. 5 Differential gene expression trend line (Tzy-x1c vs Tzy-x1a) |
与Tzy-x1c相比, 在多环芳烃胁迫状态下, K. pneumoniae Tzyx1细胞的复制分裂受到了严重的影响.微生物细胞对于芳烃等有毒化学物质会产生应答反应, 例如:脂类物质的代谢方式有了较大的变化(12条基因发生差异表达, 5条上调, 7条下调; 占参与此类代谢途径中基因总数的16%;多糖及肽聚糖表达发生差异(Starch and sucrose metabolism KPN2242_23510, Peptidoglycan biosynthesis KPN2242_07180), 细胞表面蛋白的表达量也发生明显变化(outer membrane protein N KPN2242_10000).此外, 参与细胞内外物质转运的蛋白(Membrane transport, Signal transduction, Transport and catabolism)也表现出不同程度的上调与下调, 共有17条基因发生差异表达, 7条上调, 10条下调, 说明, K. pneumoniae Tzyx1细胞对外界环境改变的调控发生了变化.
4 结论(Conclusions)根据K. pneumoniae Tzyx1转录组功能注释分析, 有514条unigenes参与了碳氢化合物的运输与降解, 45个unigenes可能参与多环芳烃的降解.K. pneumoniae Tzyx1至少存在2种完整的KEGGpathway记录的苯环开环的方式, 同时, K. pneumoniae Tzyx1中可能参与苯环开环后的降解, 但不参与上述两条代谢途径的unigenes, 由此推断K.pneumoniae Tzyx1可能存在其他未报道的参与苯环开环的基因, 根据差异表达基因分析结果, 有相当一部分未知功能的潜在酶基因及调控转运因子上调表达, 也有可能存在其他未报道的苯环开环的方式.
在以多环芳烃为碳源和以葡萄糖为碳源时, Klebsiella pneumoniae Tzyx1 5264个总基因中差异表达的比例为5%, Tzy-x1a与Tzy-x1c的转录组数据中找到254个差异表达基因(FDR≤0.05).由此可知, 以多环芳烃为碳源和以葡萄糖为碳源时对于Klebsiella pneumoniae Tzyx1基因表达的影响范围并不广, 主要集中在碳氢化合物代谢、基因复制、细胞对外界改变的调控3个方面, 其中碳氢化合物代谢、细胞对外界改变的调控相关的差异表达基因上调与下调的比例相当, 由此可见, Klebsiella pneumoniae Tzyx1对于以芳烃化合物为碳源, 其体内存在着较为完整的适应系统.而基因复制相关的差异表达基因均为下调, 因此在芳烃化合物存在的情况下, K. pneumoniae Tzyx1细胞的复制分裂受到了严重的影响.微生物细胞对于芳烃等有毒化学物质的会产生应答反应, 这在K. pneumoniae Tzyx1差异表达KEGG分析结果中均有相应的体现; 参与细胞内外物质转运的蛋白也表现出不同程度的上调与下调.在转录组的差异基因中, 还有一部分基因是未知功能的, 这些基因可能参与K. pneumoniae Tzyx1的特殊多环芳烃降解机制, 值得进一步研究.
Camacho C, Coulouris G, Avagyan V, et al. 2009. BLAST architecture and applications[J]. BMC Bioinformatics, 10(1): 421–429.
DOI:10.1186/1471-2105-10-421
|
Carlson J. 2009. Trinity Health's ch-ch-changes.New C-suite jobs aim to put focus on ambulatory care[J]. Modern healthcare, 39(22): 17–24.
|
Cock P J, Fields C J, Goto N, et al. 2010. The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants[J]. Nucleic Acids Research, 38(6): 1767–1771.
DOI:10.1093/nar/gkp1137
|
Erlich Y, Mitra P P, delaBastide M, et al. 2008. Alta-Cyclic:a self-optimizing base caller for next-generation sequencing[J]. Natural Methods, 5(8): 679–682.
DOI:10.1038/nmeth.1230
|
Grabherr M G, Haas B J, Yassour M, et al. 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 29(7): 644–652.
DOI:10.1038/nbt.1883
|
Jacques R J S, Okeke B C, Bento F M, et al. 2008. Microbial consortium bioaugmentation of a polycyclic aromatic hydrocarbons contaminated soil[J]. Bioresource Technology, 99(7): 2637–2643.
DOI:10.1016/j.biortech.2007.04.047
|
Langmead B, Salzberg S L. 2012. Fast gapped-read alignment with Bowtie 2[J]. Natural Methods, 9(4): 357–359.
DOI:10.1038/nmeth.1923
|
Michiei S, Chantal H, Charles W G. 2004. Two distinct gene clusters encode pyrenedegradationin Mycobacterium sp. strain S65[J]. Microbiology, 48(2): 209–220.
|
Mitchell S G, Khanra S, Miras H N, et al. 2009. The trinity of polyoxometalates:connecting Keggin and Dawson clusters to triangles[J]. Chemical Communications, 40(31): 2712–2714.
|
平立凤, 骆永明. 2005. 有机质对多环芳烃环境行为影响的研究进展[J]. 土壤, 2005, 37(4): 362–369.
|
Poretsky R S, Bano N, Buchan A. 2005. Analysis of microbial gene transcripts in environmental samples[J]. Applied and Environmental Microbiology, 71(7): 4121–4126.
DOI:10.1128/AEM.71.7.4121-4126.2005
|
秦宁, 何伟, 王雁, 等. 2013. 巢湖水体和水产品中多环芳烃的含量与健康风险[J]. 环境科学学报, 2013, 33(1): 230–239.
|
Sadat-Mekmene L, Genay M, Atlan D, et al. 2011. Gagnaire. Original features of cell-envelope proteinases of Lactobacillus helveticus[J]. International Journal of Food Microbiology, 146(1): 1–13.
|
Seo J, Keum Y, Li Q X. 2009. Bacterial degradation of aromatic compounds[J]. Int J Environ Res Public Health, 6(1): 278–309.
|
Tang H, Wang X, Bowers J E, et al. 2008. Unraveling ancient hexaploidy through multiply-aligned angiosperm gene maps[J]. Genome Research, 18(12): 1944–1954.
DOI:10.1101/gr.080978.108
|
Tarazona S, García-Alcalde F, Dopazo J, et al. 2011. Differential expression in RNA-seq:a matter of depth[J]. Genome Research, 21(12): 2213–2223.
DOI:10.1101/gr.124321.111
|
Trapnell C, Pachter L, Salzberg S L. 2009. TopHat:discovering splice junctions with RNA-Seq[J]. Bioinformatics, 25(9): 1105–1111.
DOI:10.1093/bioinformatics/btp120
|
Trapnell C, Williams B A, Pertea G, et al. 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 28(5): 511–515.
DOI:10.1038/nbt.1621
|
Trapnell C, Roberts A, Goff L, et al. 2012. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 7(3): 562–578.
DOI:10.1038/nprot.2012.016
|
吴艳阳, 吴群河, 罗昊, 等. 2013. 沉积物中多环芳烃的生态风险评价法研究[J]. 环境科学学报, 2013, 33(2): 544–556.
|
Xie C, Mao X, Huang J, et al. 2011. KOBAS 2.0:a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 39(Web Server issue): 316–322.
|
尹春芹, 蒋新, 王芳, 等. 2012. 不同碳源刺激对老化污染土壤中PAHs降解研究[J]. 环境科学, 2012, 33(2): 633–639.
|
杨宇虹, 梁东春, 郭刚, 等. 2006. 改进TRIZOL法提取基因组DNA[J]. 医学分子生物学杂志, 2006, 3(2): 105–107.
|
 2018, Vol. 38
2018, Vol. 38


