丛枝菌根(Arbuscular Mycorrhiza, AM)真菌可与陆地上80%的维管植物根系共生, 形成分布最广泛、功能最重要的丛枝菌根(Fan et al., 2014).AM能克服微生物修复和植物修复的不足, 综合利用植物、AM真菌、根际微生物及其相互作用的根际环境而有效地原位修复污染土壤.目前, 越来越多的研究开始关注植物与AM真菌建立的共生体系对污染土壤的修复效果与机制.
阿特拉津(Atrazine)是目前世界上使用最广泛的除草剂之一, 在土壤中的半衰期长, 容易对一些后茬敏感作物产生药害, 已被欧共体、美国和日本等国家列入内分泌干扰物名单, 且对动物细胞具有遗传学毒性(Bohn et al., 2011).现已有大量研究表明, AM真菌可以显著提高对污染土壤中阿特拉津的去除效率.例如, Huang等(2007)研究发现, 玉米接种Glomus caledonium后, 可将阿特拉津去除率从31.4%提高至73.7%;紫花苜蓿与G. mosseae共生对阿特拉津的去除率高达74.65%(Song et al., 2015); G. mosseae与高粱共生修复阿特拉津污染土壤的能力优于G. intraradices(宋福强等, 2010).虽然AM真菌提高阿特拉津去除率的机制迄今仍不是十分清楚, 但有关接种AM真菌在阿特拉津去除过程中对AM共生体的生理指标、污染物的积累及土壤微生物群落结构、代谢活力等方面的影响均已有大量的深入研究(Fan et al., 2014; Rodríguez-Caballero et al., 2017).例如, 接种G. caledonium可以提高阿特拉津在玉米根系中的积累(Huang et al., 2009), 可见, 直接与污染物接触的植物根系和具有庞大菌丝网络结构的AM真菌共生形成的丛枝菌根被视为有机污染物的“先锋区”.那么根际微生物群落对富集的阿特拉津胁迫和驯化是如何响应的?宋福强等(2016)的研究结果表明, AM真菌能够提高在阿特拉津胁迫下根际土壤中蔗糖酶、脲酶、过氧化氢酶和纤维素酶的活性.Huang等(2009)利用磷脂脂肪酸(PLFAs)生物标记技术发现, AM真菌对土壤微生物群落结构具有显著影响.由此可知, AM真菌对阿特拉津胁迫下的根际微生物群落结构和活性均产生影响, 但关于根际细菌群落组成和多样性的相关研究报道还较少.
本研究将紫花苜蓿(M. sativa)接种摩西管梗囊霉(F. mosseae), 形成AM共生体后施加10 mg·kg-1阿特拉津, 当阿特拉津去除率达到50%时, 以16S rRNA基因V4区为分子标靶, 通过lllumina Hiseq高通量测序技术揭示AM真菌和阿特拉津分别对根际细菌群落组成和多样性的影响, 以及F. mosseae菌根根际细菌群落对阿特拉津胁迫的响应, 为探讨丛枝菌根修复阿特拉津污染土壤机制提供理论依据.
2 材料与方法(Materials and methods) 2.1 盆栽设计紫花苜蓿(Medicago sativa)种子购自黑龙江省农业科学院草业研究所; 供试AM真菌为摩西管柄囊霉(Funneliformis mosseae, 曾用名:Glomus mosseae), 接种物由孢子、菌丝及菌根片段组成, 孢子含量约为25个·g-1接种物, 由黑龙江大学修复生态实验室保藏.
盆栽基质由林下土、蛭石和细沙以5: 3: 2(体积比)比例均匀混合而成, 121 ℃、0.1 MPa高温蒸汽灭菌1 h, 以灭活土著AM真菌活性.M. sativa种子经消毒、催芽后播种, 并以每盆培养基质的2%(质量分数)接种F. mosseae接种物和灭活接种物, 分别作为接种处理(AM)与未接种处理(NM), 且每盆(300 mm×100 mm×150 mm, 基质约为3.5 kg)定苗80株.植株培养30 d左右, 利用醋酸墨水染色法检测菌根侵染率.当AM真菌侵染率达到90%以上时, 施加10 mg·kg-1阿特拉津溶液胁迫(实验团队前期研究发现, 当基质中阿特拉津剂量为10 mg·kg-1时, M. sativa的光合作用受到抑制但非致死), 最终形成未接种+未施药(NM.NA)、接种+未施药(AM.NA)、未接种+施药(NM.AT)和接种+施药(AM.AT)4种处理, 每种处理3盆重复.盆栽苗子在黑龙江省植物园内露天常规培养, 但水分完全采用人工控制.
定期对阿特拉津去除率进行检测, 当AM.AT处理中阿特拉津去除率达到50%时, 每盆随机选取40个植株, 用毛刷收集苜蓿根际土壤(<0.5 cm)混合作为1个样品, 每种处理3个重复, 共计12个样品, 然后分别测定土壤理化性质和提取土壤基因组DNA.
2.2 土壤理化性质土壤总氮(TN)采用重铬酸钾-硫酸消化法测定, 铵态氮(AN)和硝态氮(NN)采用氯化钾浸提-钼锑抗分光光度测定, 速效磷(AP)采用碱熔-钼锑抗分光光度法测定, 速效钾(AK)采用中性乙酸铵提取-火焰光度计法测定, 总有机质(TOC)采用重铬酸钾容量法测定, pH值采用2.5: 1的水土比, 用电位计法测定, 以上分析方法见《土壤农业化学分析方法》(鲁如坤, 1999).土壤中阿特拉津残留量采用液相色谱法测定(Song et al., 2015).
2.3 细菌16S rRNA基因测序利用FastDNA® Spin Kit for Soil试剂盒提取土壤基因组DNA, 经1%琼脂糖凝胶电泳定性检测和Nanodrop 2000分光光度计进行DNA纯度和浓度检测(张智猛等, 2017).稀释后的基因组DNA利用通用引物515F(GTGCCAGCMGCCGCGGTAA)和806R(GGACTACHVGGGTWTCTAAT)对16S rRNA基因V4区进行扩增(Caporaso et al., 2011).PCR扩增条件:94 ℃变性30 s, 56 ℃退火30 s, 72 ℃延伸30 s, 25次循环后, 72 ℃终延伸5 min.所得PCR产物经试剂盒(QIAGEN)纯化后并依据浓度等量混合, 委托北京诺禾致源生物信息科技有限公司在Hiseq 2500平台上进行测序.
2.4 数据处理分析使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建.将HiSeq 2500下机测序数据截去Barcode和引物序列后, 使用FLASH(V 1.2.7)进行每个样品的reads拼接, 然后通过Qiime进行Tags截取和长度过滤(Caporaso et al., 2010), 采用UCHIME(Edgar et al., 2011)程序和Gold database数据库去除嵌合体序列, 得到最终有效数据(Effective Tags).
利用Uparse(V 7.0.1001)对所有样品的全部有效数据进行聚类(Edgar et al., 2013), 以97%的一致性将序列聚类成为OTUs(Operational Taxonomic Units), 以出现频数最高的序列作为OTUs的代表序列.用Mothur与SILVA(Quast et al., 2013)的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1), 最后对各样品的数据进行均一化处理.
使用SPSS 19.1进行数据统计分析, 差异显著性检验采用Two-way ANOVA, 相关性分析采用Spearman法.细菌群落α-多样性采用物种数目(Observed-species)、Chao1、Shannon和Simpson指数来计算.利用CANOCO 4.5对土壤理化性质和微生物群落结构进行典范对应分析(CCA).
3 结果(Results) 3.1 土壤理化性质与AM真菌侵染当M. sativa培养到35 d时测定菌根侵染率, 发现F. mosseae与M. sativa可以形成良好的共生关系, 侵染率高达91%以上, 此时在基质中施加阿特拉津进行胁迫.第50 d时检测阿特拉津残留量发现, 接种F. mosseae可将M. sativa对土壤中阿特拉津的去除率由33.4%提高至52.23%, 表明接种AM真菌可以显著提高阿特拉津去除率.此外, 施加阿特拉津显著提高了土壤pH值和土壤速效钾含量(p<0.05), 其他土壤理化指标均未受接种F. mosseae、施加阿特拉津及二者交互作用显著影响(p>0.05)(表 1).
| 表 1 不同处理中根际土壤理化性质与AM真菌侵染率 Table 1 Soil physicochemical properties and AMF colonization rate in different treatments |
在未接种+未施药(NM.NA)、接种+未施药(AM.NA)、未接种+施药(NM.AT)和接种+施药(AM.AT)4种处理根际土壤中, 相对丰度较高的细菌门主要包括变形菌门(Proteobacteria, 37.6%~43.3%)、放线菌门(Actinobacteria, 10.5%~15.9%)、厚壁菌门(Firmicutes, 10.6%~12.9%)、酸杆菌门(Acidobacteria, 8.1%~10.2%)和绿弯菌门(Chloroflexi, 6.8%~10.1%)等(图 1).其中, 变形菌门在4种处理中均为优势类群, 主要包括α-变形菌纲(Alphaproteobacteria, 18.11%~21.95%)、β-变形菌纲(Betaproteobacteria, 10.89%~16.51%)、γ-变形菌纲(Gammaproteobacteria, 3.85%~4.69%)和δ-变形菌纲(Deltaproteobacteria, 2.83%~4.29%).
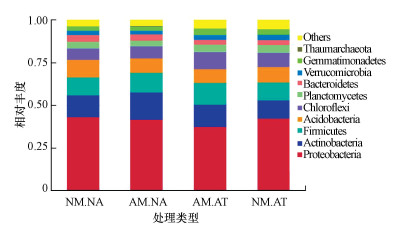 |
| 图 1 不同处理根际土壤细菌在门水平的分类 Fig. 1 Relative abundances of bacterial phyla in different rhizosphere soil |
在4种处理中, 平均相对丰度>1%的菌属依次为芽孢杆菌属(Bacillus, 6.28%~7.85%)、苯基杆菌属(Phenylobacterium, 2.47%~3.78%)、微枝形杆菌属(Microvirga, 1.69%~2.20%)、Tumebacillus(1.17%~1.51%)、新鞘氨醇杆菌属(Novosphingobium, 1.13%~2.01%)、类诺卡氏属(Nocardioides, 1.10%~1.29%)、Ramlibacter(1.08%~1.59%)、节杆菌属(Arthrobacter, 1.02%~3.30%)、Massilia (1.02%~1.77%)、鞘氨醇单胞菌属(Sphingomonas, 0.95%~1.29%)和假单胞菌(Pseudomonas, 0.67%~1.49%), 未分类的菌属丰度均在76%以上(图 2).
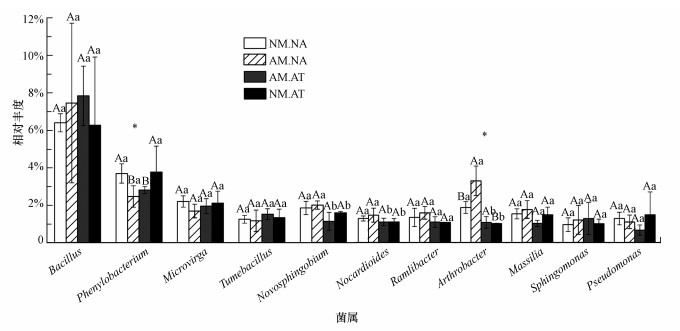 |
| 图 2 接种和施药对根际土壤优势细菌属相对丰度影响 (不同大写字母表示接种处理具有显著性差异(p<0.05), 不同小写字母表示施药处理具有显著性差异(p<0.05), *表示接种与施药存在交互作用) Fig. 2 The effect of inoculation and atrazine on the relative abundance of dominant bacterial genera |
细菌属Bacillus在4种处理中的丰度虽为最高, 但均不受接种F. mosseae、施加阿特拉津及其二者交互作用的显著影响.接种F. mosseae显著提高了Arthrobacter的相对丰度, 但降低了Phenylobacterium的相对丰度.施加阿特拉津均显著降低了Novosphingobium、Nocardioides和Arthrobacter的相对丰度.另外, 接种和施药处理对Phenylobacterium和Arthrobacter的相对丰度存在显著交互作用.由此可见, 本研究中Arthrobacter对接种AM真菌和阿特拉津处理及二者交互作用响应相对敏感.
3.3 细菌群落α-多样性分析本研究通过Observed_species(物种数目)、Shannon指数(多样性指数)、Simpson指数(优势度指数)和Chao1指数(丰度指数)表征细菌群落α-多样性.由表 2可知, 覆盖率(Goods coverage)在4种处理中的数值均达到95%以上, 说明本次测序结果反映了样本中细菌群落的真实情况.细菌群落Shannon指数在接种和施药双处理(AM.AT)中最高.在接种处理(AM.NA与AM.AT)中, 施加阿特拉津引起细菌群落Shannon指数和物种数目的显著增加.在阿特拉津胁迫下, 根际细菌群落丰度与多样性趋势一致, 即AM根际(AM.AT)大于未接种根际(NM.AT).在未胁迫处理下, 接种F. mosseae提高了细菌群落丰度指数, 降低了多样性指数.
| 表 2 根际细菌α-多样性指标及其与AM真菌、阿特拉津相关性 Table 2 Alpha diversity index of bacteria community in different rhizosphere soil |
对不同处理土壤细菌的OTU(97%)组成进行典范对应分析, 结果显示, 每种处理的3个重复聚在一起, 说明本研究的样品重现性较好, 两轴累计贡献率为43.46%(图 3).AM.AT和NM.AT分布在第一排序轴的正方向, AM.NA和NM.NA分布在第一排序轴的负方向, 这表明阿特拉津胁迫是第一排序轴的主要影响因素, 变异解释率为23.25%.AM.AT和AM.NA分布在第二排序轴的正方向, NM.AT和NM.NA分布在第二排序轴的负方向, 表明接种AM真菌是第二排序轴的主要影响因素, 变异解释率为20.21%.
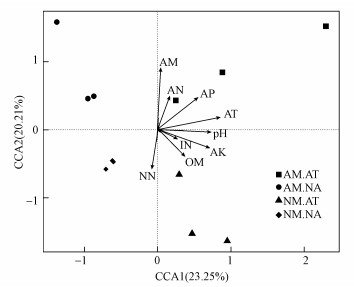 |
| 图 3 细菌群落结构的典范对应分析 Fig. 3 The CCA of bacterial communities changes with environmental variables |
通过排列检验显示, 土壤环境因子OM、TN、AP、AK和pH对土壤细菌群落结构也具有一定的影响, 但没有接种F. mosseae或施加阿特拉津影响显著.
通过对根际土壤中细菌属与AM真菌、阿特拉津胁迫之间的Spearman相关性分析(表 3)可知, F. mosseae与Phenylobacterium、Bryobacter、Variibacter、Pirellula和Ensifer呈显著负相关(p<0.05), F. mosseae与Devosia、Planctomyces呈显著负相关(p<0.01).此外, 阿特拉津与Arthrobacter、Azohydromonas、Noviherbaspirillum、Flavisolibacter呈显著负相关(p<0.01), 仅与Reyranella呈显著正相关(p<0.05).
| 表 3 与F. mosseae、阿特拉津胁迫处理显著相关的细菌属 Table 3 Spearman analysis of bacterial genus with F. mosseae and atrazine in rhizosphere soil |
在本研究中, 接种F. mosseae可以提高苜蓿对土壤中阿特拉津的去除率, 该结果与宋福强等(2010)、Huang等(2007; 2009)的研究结果一致.虽然与上述研究相比, 本研究选用的AM真菌、宿主植物、培养基质、栽培时间、环境因子和阿特拉津浓度等诸多因素存在不同, 但也再次证明了接种AM真菌可以有效地增强宿主植物对阿特拉津污染土壤的修复成果.
近年来, 关于AM真菌与土壤微生物群落之间的互作已开展了大量研究.AM真菌G. mosseae、G. intraradices及混合接种可以显著提高土壤真菌、细菌和放线菌数量(郭欢等, 2013; 宋福强等, 2010; 2015;孙金华等, 2015).郭欢等(2013)研究发现, 接种G. mosseae和G. intraradices提高了红花(Carthamus tinctorius L.)根际土壤微生物群落Shannon指数.然而, 接种G. hoi未对Plantago lanceolata L.根际土壤细菌群落Shannon指数产生显著影响(Nuccio et al., 2013).接种G. caledonium也未导致Chao 1指数的显著性变化(Cao et al., 2016).同时, 对灌木Salvia officinalis L.、Lavandula dentata L.、Thymus vulgaris L.和Santolina chamaecyparissus L.分别接种Rhizophagus intraradices, 也均未显著改变根际细菌群落的α-多样性(Rodríguez-Caballero et al., 2017).由此可见, 有关AM真菌对根际细菌群落α-多样性的影响并未取得一致的结论.在本研究中, 无论是否受到阿特拉津胁迫, 接种F. mosseae对根际细菌群落Simpson指数、Shannon指数和Chao1指数均无显著影响.另外, 本研究关于阿特拉津对土壤细菌群落α-多样性的影响与Fang等(2015)的研究结果一致, 即施加阿特拉津并未对土壤细菌群落Shannon指数产生显著影响.
在Pseudomonas sp. strain ADP和Arthrobacter aurescens TC1首次分离之后, 至今已有大量关于从阿特拉津重度污染生境富集并筛选高降解能力细菌菌株的报道(Zhao et al., 2017), 相继获得了Pseudomonas、Arthrobacter和其他菌属的降解菌, 如Nocardioides(Topp et al., 2000)、Frankia(Rehan et al., 2017)、Chelatobacter(Rousseaux et al., 2001)、Ensifer(Ma et al., 2017)、Acinetobacter(Yang et al., 2016)、Raoultella(Swissa et al., 2014)、Rhodobacter(Du et al., 2011)、Alcaligenes(Aem et al., 2007)、Aminobacter、Stenotrophomonas(Rousseaux et al., 2001)、Methylobacterium等.在本研究的4种处理中, Arthrobacter(1.020%~3.295%)、Pseudomonas(0.668%~1.487%)和Nocardioides(1.096%~1.464%)的相对表达丰度均大于1%.Ensifer(0.283%~0.405%)、Bradyrhizobium(0.276%~0.538%)、Burkholderia(0.117%~0.282%)、Rhodobacter(0.082%~0.145%)、Mycobacterium(0.045%~0.091%)、Frankia(0.046%~0.061%)、Methylobacterium(0.036%~0.051%)、Acinetobacter(0.014%~0.021%)和Stenotrophomonas(0.003%~0.026%)的相对丰度依次降低.在阿特拉津胁迫下, F. mosseae使得Arthrobacter的相对丰度由1.020%升至1.075%, Mycobacterium的相对丰度由0.063%升至0.091%, Frankia的相对丰度由0.046%升至0.061%, Stenotrophomonas的相对丰度由0.003%升至0.007%.由此可见, 虽然阿特拉津胁迫降低了根际土壤中Arthrobacter、Frankia和Stenotrophomonas的相对丰度, 但接种F. mosseae在一定程度上提高了具有阿特拉津降解潜力菌属的相对丰度.
此外, 接种F. mosseae提高了阿特拉津胁迫下土壤中具有阿特拉津降解潜力菌属Arthrobacter、Mycobacterium、Frankia和Stenotrophomonas的相对丰度, 其中Arthrobacter受F. mosseae、阿特拉津及其二者交互作用的显著性影响.
5 结论(Conclusions)1) 不同处理的根际细菌在门水平相对丰度较高(>10%)的为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和绿弯菌门(Chloroflexi).
2) 苜蓿单独接种F. mosseae或施加阿特拉津均未对根际土壤细菌群落α-多样性指数产生显著影响, 但先接种F. mosseae形成丛枝菌根后, 施加阿特拉津可引起细菌群落Shannon指数显著增加.
3) 接种F. mosseae提高了M. sativa根际土壤中Arthrobacter、Mycobacterium、Frankia和Stenotrophomonas等具有阿特拉津降解潜力菌属的相对丰度.
Aem C, Ritter W F, Radosevich M. 2007. Isolation of a selected microbial consortium from a pesticide-contaminated mix-load site soil capable of degrading the herbicides atrazine and alachlor[J]. Soil Biology & Biochemistry, 39(12): 3056–3065.
|
Bohn T, Cocco E, Gourdol L, et al. 2011. Determination of atrazine and degradation products in Luxembourgish drinking water:origin and fate of potential endocrine-disrupting pesticides[J]. Food Additives & Contaminants, 28(8): 1041–1054.
|
Cao J, Feng Y, Lin X, et al. 2016. Arbuscular mycorrhizal fungi alleviate the negative effects of iron oxide nanoparticles on bacterial community in rhizospheric soils[J]. Frontiers in Environmental Science.
DOI:10.3389/fenvs.2016.00010
|
Caporaso J G, Kuczynski J, Stombaugh J, et al. 2010. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 7(5): 335–336.
DOI:10.1038/nmeth.f.303
|
Caporaso J G, Lauber C L, Walters W A, et al. 2011. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences, 108: 4516–4522.
DOI:10.1073/pnas.1000080107
|
Du J, Zhang Y, Ma Y, et al. 2011. Simulation study of atrazine-contaminated soil biodegradation by strain W16[J]. Procedia Environmental Sciences, 11(1): 1488–1492.
|
Edgar R C. 2013. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 10(10): 996–998.
DOI:10.1038/nmeth.2604
|
Edgar R C, Haas B J, Clemente J C, et al. 2011. UCHⅡME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 27(16): 2194–2200.
DOI:10.1093/bioinformatics/btr381
|
Fan X X, Song F Q. 2014. Bioremediation of atrazine:recent advances and promises[J]. Journal of Soils and Sediments, 14(10): 1727–1737.
DOI:10.1007/s11368-014-0921-5
|
Fang H, Lian J, Wang H, et al. 2015. Exploring bacterial community structure and function associated with atrazine biodegradation in repeatedly treated soils[J]. Journal of Hazardous Materials, 286(11): 457–465.
|
郭欢, 曾广萍, 刘红玲, 等. 2013. 丛枝菌根真菌对红花根围微生物多样性特征的影响[J]. 微生物学通报, 2013, 40(7): 1214–1224.
|
Huang H, Zhang S, Shan X, et al. 2007. Effect of arbuscular mycorrhizal fungus (Glomus caledonium) on the accumulation and metabolism of atrazine in maize (Zea mays L.) and atrazine dissipation in soil[J]. Environmental Pollution, 146(2): 452–457.
DOI:10.1016/j.envpol.2006.07.001
|
Huang H, Zhang S, Wu N, et al. 2009. Influence of Glomus etunicatum/Zea mays mycorrhiza on atrazine degradation, soil phosphatase and dehydrogenase activities, and soil microbial community structure[J]. Soil Biology & Biochemistry, 41(4): 726–734.
|
刘驰, 李家宝, 芮俊鹏, 等. 2015. 16S rRNA基因在微生物生态学中的应用[J]. 生态学报, 2015, 35(9): 2769–2788.
|
鲁如坤. 1999. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社.
|
Ma L, Chen S, Yuan J, et al. 2017. Rapid biodegradation of atrazine by Ensifer sp.strain and its degradation genes[J]. International Biodeterioration & Biodegradation, 116: 133–140.
|
Nuccio E E, Hodge A, Pett-Ridge J, et al. 2013. An arbuscular mycorrhizal fungus significantly modifies the soil bacterial community and nitrogen cycling during litter decomposition[J]. Environmental Microbiology, 15(6): 1870–1881.
DOI:10.1111/emi.2013.15.issue-6
|
Quast C, Pruesse E, Yilmaz P, et al. 2013. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J]. Nucleic Acids Research, 41: 590–596.
|
Rehan M, Fadly G E, Farid M, et al. 2017. Opening the s-triazine ring and biuret hydrolysis during conversion of atrazine by Frankia sp.strain EuI1c[J]. International Biodeterioration & Biodegradation, 117: 14–21.
|
Rodríguez-Caballero G, Caravaca F, Fernández-González A J, et al. 2017. Arbuscular mycorrhizal fungi inoculation mediated changes in rhizosphere bacterial community structure while promoting revegetation in a semiarid ecosystem[J]. Science of the Total Environment, 584-585(15): 838–848.
|
Rousseaux S, Hartmann A, Soulas G. 2001. Isolation and characterisation of new Gram-negative and Gram-positive atrazine degrading bacteria from different French soils[J]. FEMS Microbiology Ecology, 36(2/3): 211–222.
|
Song F, Li J, Fan X, et al. 2015. Transcriptome analysis of Glomus mosseae/Medicago sativa mycorrhiza on atrazine stress[J]. Scientific Reports, 6: 20245.
|
宋福强, 程蛟, 常伟, 等. 2015. 黑土农田施加AM菌剂对大豆根际菌群结构的影响[J]. 土壤学报, 2015, 52(2): 390–398.
|
宋福强, 丁明玲, 董爱荣, 等. 2010. 丛枝菌根(AM)真菌对土壤中阿特拉津降解的影响[J]. 水土保持学报, 2010, 24(3): 189–193.
|
宋福强, 范晓旭, 常伟, 等. 2016. 苜蓿菌根对土壤中阿特拉津降解及酶活性影响[J]. 中国农学通报, 2016, 32(30): 182–187.
DOI:10.11924/j.issn.1000-6850.casb16040027 |
孙金华, 毕银丽, 王建文, 等. 2015. 接种AM菌对西部黄土区采煤沉陷地柠条生长和土壤的修复效应[J]. 生态学报, 2015, 37(7): 2300–2306.
|
Swissa N, Nitzan Y, Langzam Y, et al. 2014. Atrazine biodegradation by a monoculture of Raoultella planticola isolated from a herbicides wastewater treatment facility[J]. International Biodeterioration & Biodegradation, 92: 6–11.
|
Topp E, Mulbry W M, Zhu H, et al. 2000. Characterization of S-triazine herbicide metabolism by a Nocardioides sp.isolated from agricultural soils[J]. Applied & Environmental Microbiology, 66(8): 3134–3141.
|
Yang F, Jiang Q, Zhu M, et al. 2016. Effects of biochars and MWNTs on biodegradation behavior of atrazine by Acinetobacter lwoffii DNS32[J]. Science of the Total Environment, 577(15): 54–60.
|
张智猛, 慈敦伟, 张冠初, 等. 2017. 山东地区盐碱土花生种子际土壤微生物群落结构的研究[J]. 微生物学报, 2017, 57(4): 582–596.
|
Zhao X, Ma F, Feng C, et al. 2017. Complete genome sequence of Arthrobacter sp.ZXY-2 associated with effective atrazine degradation and salt adaptation[J]. Journal of Biotechnology, 248: 43–47.
DOI:10.1016/j.jbiotec.2017.03.010
|
 2018, Vol. 38
2018, Vol. 38


