2. 宁波大学建筑工程与环境学院, 宁波 315211
2. Faculty of Architectural, Civil Engineering and Environment, Ningbo University, Ningbo 315211
近20年来, 以硝酸盐为代表的氮素大量地排入海洋, 导致近岸海域出现了不同程度的富营养化, 极大地限制了海洋生物资源的可持续利用.海洋微生物是水生生态系统的重要组成部分, 在海洋碳、氮、磷、硫等生物地球化学循环中起关键作用(Falkowski et al., 2008), 且能够对环境变化迅速做出响应(Paerl et al., 2003), 维持海洋生态系统的平衡.因此, 有必要深入了解过量氮输入对海洋微生物群落的影响.
海洋环境中营养物质的浓度与微生物群落结构和功能密切相关(Arrigo, 2005), 水体营养水平的变化除了能改变微生物的丰度和多样性外(Nogales et al., 2011; Carlsson et al., 2012), 还能显著影响微生物群落功能(Bell et al., 2009).目前, 关于氮输入对微生物群落功能影响的研究主要集中在细菌生产力、酶活性和功能微生物多样性等方面, 已有研究表明, 德国Pomeranian湾细菌生产力和生长率随着水体营养程度的升高而提高(Feuerpfeil et al., 2004), 富营养化的加剧也使得微生物蛋白质和碳水化合物分解酶的活性上升(Meyer-Reil et al., 2000).此外, nirS作为反硝化细菌的重要分子标记, 已被用于太平洋东南部海水反硝化细菌群落的研究, 发现其群落变化与溶解氧、硝酸盐和亚硝酸盐密切相关(CAstro-González et al., 2005), 以nifH作为固氮细菌分子标记的研究发现, 南海北部寡营养海水中固氮细菌的组成和丰度与硝酸盐有一定相关性(Kong et al., 2011).然而, 目前对于海洋环境中的过量氮输入引起细菌群落整体代谢潜力变化的研究还很缺乏.
研究微生物群落功能的方法很多, 如美国Biolog公司开发的Biolog-Eco板是基于微生物对碳源的利用情况来表征微生物群落局部代谢多样性的一种方法, 已被广泛用于微生物群落代谢特征研究(Lebaron et al., 2001; Almutairi, 2009).近年来, 宏基因组和宏转录组测序等高通量技术的发展更是为全面了解微生物群落功能潜力和活性提供了强大的手段, 但目前这些技术的测试成本依然很高.而PICRUSt(Phylogenetic investigation of communities by reconstruction of unobserved states)则是基于16S rRNA基因序列对原核生物群落进行功能预测的生物信息学工具(Langille et al., 2013), 它通过已测得基因组的相近物种来预测原核生物群落的宏基因组信息, 已被广泛应用到微生物组学研究中(Metcalf et al., 2016; Wang et al., 2016; Polónia et al., 2014), 为深入研究特定功能基因提供了初步依据.本研究通过构建寡营养海水生态微宇宙, 采用一次性输入和连续性输入2种方式, 分别向微宇宙中输入等浓度的硝酸盐, 并基于Biolog-Eco技术和PICRUSt预测方法, 研究寡营养海水中过量氮输入引起的细菌群落代谢潜力的变化规律.本课题组前期研究已经发现, 过量氮输入显著改变了细菌群落的物种多样性和结构(Dong et al., 2017), 本研究将进一步探索过量氮输入对细菌群落代谢多样性的影响, 为明确近岸海域细菌群落对过量氮输入的响应模式提供基础数据.
2 材料与方法(Materials and methods) 2.1 生态微宇宙构建及氮输入处理从象山东海域(122°39′36.86″E、29°21′54.72″ N, 2013年9月29日)采集表层(50 cm)海水, 在6 h内带回实验室.用200 L的塑料桶分批将海水样品混合均匀后, 以35 L·桶-1分装入容积为40 L的塑料桶(用1:30(体积比)的硫酸溶液浸泡, 并用超纯水润洗)中, 内置海水造浪泵, 稳定3 d后进行氮输入处理.共设置3个处理, 即对照组(CK)、一次性输入组(One-off Loading, 简写为OL, 一次性输入硝酸盐至体系中硝氮水平为1.0 mg·L-1)和连续性输入组(Periodic Loading, 简写为PL, 每隔2 d输入一次硝酸盐, 使体系中硝氮水平每次增加0.125 mg·L-1, 共输入8次, 直到与一次性输入组达到相同水平), 每组设4个平行, 以硝酸钠母液(1000 mg·L-1, 用0.2 μm滤膜过滤并灭菌后的清洁海水配置)作为氮源模拟氮输入, 根据采样季节设置昼夜比为14 h:10 h, 光强为340 Lx, 控制培养室温度为(27±1) ℃, 连续培养16 d.
2.2 水样采集和环境参数测定分别在实验的第0(只采集对照组样品)、2、4、8和16 d输入硝酸盐溶液前, 从12个水体积为35 L、体系容积为40 L的微宇宙中分别取3 L水样, 用0.2 μm的聚碳酸酯滤膜(Millipore Type GTTP, USA)过滤0.5 L水样, 并用灭菌PE管收集滤膜保存在-80 ℃冰箱中, 待提取DNA.采用标准方法(GB/T12763.4—2007和GB 17378.4—2007)测定水样pH、溶解氧(DO)、硝氮(NO3-)、铵氮(NH4+)、亚硝氮(NO2-)、磷酸盐(PO43-)、总磷(TP)、化学需氧量(CODMn)和悬浮颗粒物(SS)(Dong et al., 2017).
2.3 基于Biolog-Eco板的细菌群落功能多样性检测利用含有31种碳源的Biolog-Eco板(Biolog Inc., Hayward, CA, USA)进行细菌群落功能多样性检测.在实验第1 d和第16 d从每个微宇宙中分别采集水样, 将Biolog-Eco板预热至25 ℃, 用排枪从采集水样中分别取150 μL未过滤水样滴于Biolog-Eco板各孔中, 并在28 ℃下恒温连续培养240 h, 每隔12 h利用酶标仪(Molecular Device, Sunnyvale, CA)测其590 nm和750 nm下的光密度值, 并计算平均每孔颜色变化率(Average Well Color Development, AWCD)、Shannon多样性指数(席劲瑛等, 2003)和六大类碳源的平均AWCD值(孙凤霞等, 2010).
2.4 DNA提取和16S rRNA基因扩增子Illumina MiSeq测序利用MO BIO公司的Power Soil DNA Isolation Kit试剂盒提取0.2 μm孔径滤膜上的总DNA, 利用带Barcode的引物515F(5′-GTGCCAGCMGCCG CGGTAA-3′)和806R(5′-GGACTACHVGGGTWT CTAAT-3′)对16S rRNA基因V4区进行扩增, 扩增产物纯化后用Agilent 2100(Agilent, USA)生物测定仪检测片段大小, 并用Qubit 2.0荧光光度计(Life Technologies, USA)定量.最后, 对PCR产物进行等物质的量合并, 在Illumina MiSeq平台上进行测序(Illumina, USA).
所得序列已上传到DDBJ数据库(http://www.ddbj.nig.ac.jp/, 登录号DRA004922)(Dong et al., 2017).首先使用FLASH对双末端序列进行拼接(Magoč et al., 2011), 随后利用QIIME 1.7.0对拼接后的序列进行质控(Caporaso et al., 2010), 利用USEARCH筛除嵌合体(Edgar et al., 2011), 再利用pick_closed_reference_otus.py脚本针对Greengenes 13.5数据库进行OTU聚类(序列相似性大于97%), 去除古菌、叶绿体和不能分类到细菌界的序列.
2.5 基于PICRUSt算法的群落功能基因预测利用PICRUSt中的normalize_by_copy_number.py脚本对OTU表进行拷贝数标准化, 然后用predict_metagenomes.py脚本进行功能基因预测, 生成KEGG(Kyoto Encyclopedia of Genes and Genomes)同源基因(KEGG Orthologs, KOs)表, 再利用categorize_by_function.py脚本将KO表转化为第3层次的功能基因家族表进行下游分析.
2.6 统计分析方法由于环境样品比较复杂, 而处于AWCD曲线“拐点”处的数据, 其分析结果相对比较稳定(Cookson et al., 2008; Han et al., 2007; Stenrod et al., 2006).因此, 本研究中选用了Biolog-Eco板培养144 h后的数据进行多样性指数、碳源利用率和主成分分析(Principal Component Analysis, PCA).基于最小显著差别(Least Significant Difference, LSD)进行AWCD值的方差分析(Analysis of Variance, ANOVA), 检验不同氮输入条件下碳源利用能力的差异, 利用双因素方差分析(Two-Way ANOVA)判断采样时间和氮输入对碳源利用率的影响.利用PAST软件针对31种碳源的相对吸光值进行PCA分析来表征各组细菌群落的代谢特征差异(Giaramida et al., 2013), 并利用Primer-E v5软件进行相似性分析(Analysis of Similarity, ANOSIM)检验各组碳源利用特征差异的显著性.利用主坐标分析(Principal Coordinates Analysis, PCoA)表征基于PICRUSt预测的功能基因家族组成(KEGG第3层次)差异, 并在Primer-E v5软件中进行双因素交互相似性分析(Two-way crossed ANOSIM), 检验氮输入和采样时间对功能基因家族组成的影响.根据KEGG氮代谢通路, 筛选出氮代谢功能基因, 利用R中的vegan包去除与氮代谢基因组成无显著相关(p>0.05)和膨胀因子大于20的环境因子, 筛选出与氮代谢基因组成密切相关的环境因子进行冗余分析(Redundancy Analysis, RDA);通过双因素方差分析检验氮输入和采样时间对氮代谢基因相对丰度的影响, 利用t检验分析对照组与氮输入组之间氮代谢基因相对丰度的差异, 并对氮代谢基因相对丰度与硝酸盐浓度进行相关性分析.方差分析、t检验和相关性分析均在SPSS16.0软件中完成, 文中所有图件均利用OriginPro 8.5软件制作.
3 结果分析(Results and analysis) 3.1 主要水体理化因子的相关性整个实验周期内, NO3-只与NH4+显著相关, 而NH4+则与很多环境因子相关, 如与NO3-、NO2-和DO等显著正相关, 与TP、CODMn和pH显著负相关(表 1).
| 表 1 主要环境因子间的Pearson相关系数 Table 1 Pearson correlation coefficient between the main environmental factors |
Biolog-Eco板通过AWCD值来反映微生物的碳源利用能力(谭兆赞等, 2006), 其值越高表明细菌群落代谢活性越高.在实验的第1 d, AWCD值在培养120 h后升幅较大, 但组间均无显著差异(图 1);第16 d的样品在培养60 h后AWCD值迅速升高, 对照组与一次性输入组的AWCD值随培养时间呈相似的变化规律, 并于培养后期达到饱和状态, 而连续性输入组在培养后期则呈线性增长趋势, 180 h后逐渐高于其余两组, 且在204和228 h时呈显著差异(p<0.05).
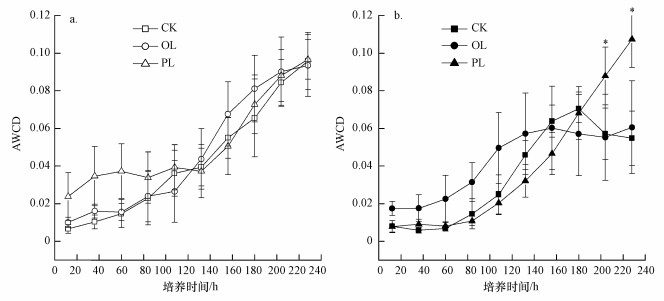 |
| 图 1 实验第1 d (a)和16 d (b)时Biolog-Eco板AWCD值(数据为平均值±标准差(n=4);*表示PL组与对照组或OL组间有显著性差异(p<0.05, 基于LSD)) Fig. 1 The average well color development (AWCD) of Biolog-Eco plates at days 1 (a) and 16 (b) during the experiment |
根据实验第1 d和第16 d的样品在Biolog-Eco板上培养144 h时的Shannon指数(表 2), 发现氮输入方式对细菌群落代谢多样性没有显著影响.
| 表 2 细菌群落代谢多样性 Table 2 Metabolic diversity of bacterial communities |
微生物群落对碳源的利用情况反映了微生物群落组成的变化(夏雪等, 2011).对Biolog-Eco板培养144 h后6类碳源的AWCD值进行双因素方差分析(结果未显示), 发现总体上碳源利用率未受到采样时间和氮输入的明显影响(图 2), 但对照组和连续性输入组中细菌群落对酯类的利用率随采样时间显著升高(p<0.05).
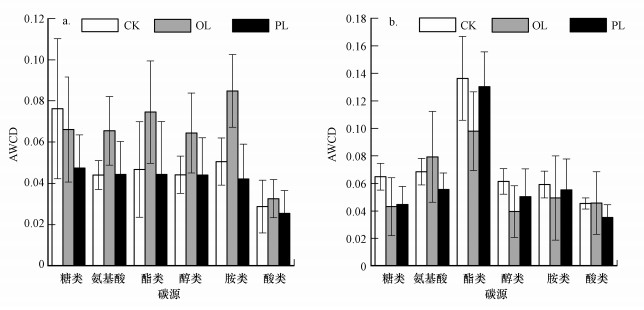 |
| 图 2 实验第1 d (a)和16 d (b)时细菌群落对6类碳源的利用情况(基于Biolog-Eco板培养144 h后的数据, 数据为平均值±标准差(n = 4)) Fig. 2 The utilization pattern of bacterial communities for six carbon sources at days 1(a) and 16(b)during the experiment |
主成分分析结果表明, 对PC1贡献较大的碳源主要是酯类, 对PC2贡献较大的主要是氨基酸、胺类和酸类(图 3).相似性分析发现, 对照组和连续性输入组的细菌群落碳源利用特征随采样时间明显变化(p<0.05, 数据未在图表中展示).
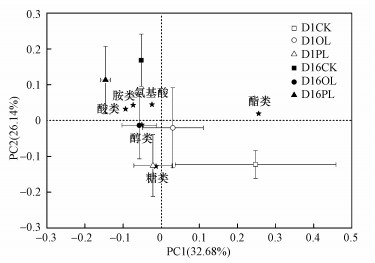 |
| 图 3 细菌群落碳源利用特征变化的主成分分析(基于Biolog-Eco板培养144 h后的数据, 数据为平均值±标准差(n=4);D1CK、D1OL、D1PL分别表示第1天对照组、一次性输入组、连续性输入组;D16CK、D16OL、D16PL分别为第16天相应处理组, 下同) Fig. 3 Principal component analysis (PCA) for carbon source utilization pattern of bacterial communities |
经PICRUSt预测后每个样品的NSTI值为0.120~0.193, 平均值为0.155.整个实验周期内细菌群落功能基因家族组成呈明显的时间变化, 且2 d后各实验组随采样时间呈相对一致的变化规律(图 4).基于Bray-Curtis非相似性的双因素交互相似性分析表明, 功能基因家族组成有显著的时间变异(Global r=0.651, p=0.001), 而氮输入总体上对功能基因组成没有显著影响(Global r=0.097, p=0.101);成对比较发现, 虽然一次性输入组与对照组没有显著差异, 但连续性输入组与对照和一次性输入组均有显著差异(表 3).
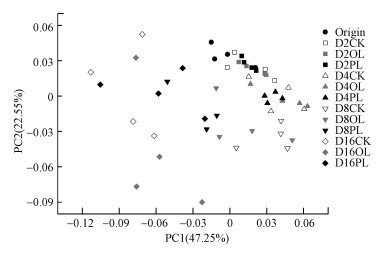 |
| 图 4 基于PICRUSt预测的细菌群落功能基因家族组成变化的主坐标分析 Fig. 4 Principal coordinates analysis (PCoA) illustrating variation in functional gene family composition of bacterial communities based on PICRUSt prediction |
| 表 3 基于PICRUSt预测的细菌群落功能基因家族组成的双因素交互相似性分析 Table 3 Two-way crossed ANOSIM for functional gene family composition of bacterial communities based on PICRUSt prediction |
微生物介导的氮代谢是地球氮循环的主要驱动力(李丹等, 2015), 氮代谢主要包括固氮、氨氧化、硝化、同化硝酸盐还原、异化硝酸盐还原、反硝化、厌氧氨氧化等过程.本研究利用PICRUSt预测了部分氮代谢关键基因的相对丰度, 主要有nifDKH、hao、nxrAB、napA、napB、narGHI、nirK、norBC、nosZ、nrfA、nirBD、nirA和nasAB, 其中, narGHI、nirK、norBC、nirBD和nasAB的相对丰度较高(图 5).双因素方差分析表明, 氮代谢相关基因的相对丰度随时间显著变化(p<0.001), 氮输入仅对nrfA的相对丰度有显著影响(p<0.05), 时间和氮输入的交互作用对napB和nrfA的相对丰度有显著影响(p<0.05)(表 4).由此可见, 氮代谢基因相对丰度的时间变化超过氮输入的影响.RDA分析结果显示, 时间和氮输入对氮代谢基因家族组成的影响, 主要与NO2-、NH4+、PO43-、CODMn、DO、pH和TP密切相关(图 6).
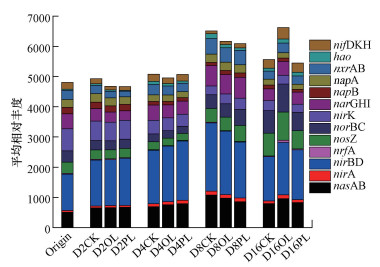 |
| 图 5 基于PICRUSt预测的氮代谢相关基因的平均相对丰度 Fig. 5 The average relative abundances of genes relevant with N metabolism based on PICRUSt prediction |
| 表 4 不同采样时间和氮输入方式下基于PICRUSt预测的氮代谢基因相对丰度的双因素方差分析 Table 4 Two-way analysis of variance (ANOVA) of the relative abundances of genes relevant with N metabolism based on PICRUSt prediction across different sampling times and N loading modes |
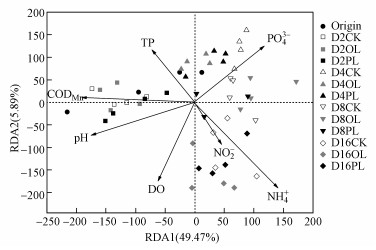 |
| 图 6 基于PICRUSt预测的氮代谢功能基因家族组成的冗余分析 Fig. 6 Redundancy analysis (RDA) of functional gene family composition relevant with N metabolism of bacterial communities based on PICRUSt prediction |
通过系统地比较2种氮输入条件下氮代谢基因相对丰度与对照组的差异(图 7), 发现实验前2 d氮输入对氮代谢基因相对丰度的影响较小, 只有nifDKH的相对丰度显著降低;而4 d后氮输入对更多的基因产生影响, nifDKH、napB、nirK、nrfA、nirBD和nasAB的相对丰度在不同时间点发生显著变化;相对于一次性输入组而言, 连续性输入组与对照组间氮代谢基因相对丰度的差异更为普遍.Spearman相关性分析发现, napB和nirK的相对丰度与NO3-浓度呈显著负相关, nirBD、nirA和nasAB的相对丰度与NO3-浓度呈显著正相关(表 5).
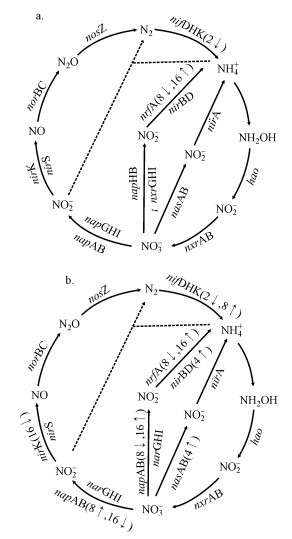 |
| 图 7 对照与氮输入组间氮代谢基因相对丰度差异(a.对照组vs.一次性输入组, b.对照组vs.连续性输入组;括号内的数字表示采样时间(d), ↑(↓)表示该基因相对丰度显较对照组著性升高或降低(t检验, p<0.05)) Fig. 7 Differences in relative abundances of the genes related to N metabolism between CK and N loading groups(a.CK vs. OL, b.CK vs. PL) |
| 表 5 氮代谢基因相对丰度与硝酸盐浓度的Spearman相关性 Table 5 Spearman rank correlations between the relative abundances of genes relevant with N metabolism and nitrate concentration |
Biolog-Eco板的AWCD值可以揭示微生物群落的局部代谢能力, 是反映微生物活性、描述微生物群落碳源利用多样性的一个重要指标(梅冰等, 2016).连续输入氮至过量时, 提高了细菌群落碳源利用能力.已有研究表明, 营养物质的输入会促进微生物代谢活性(秦旭等, 2014), 在实验培养后期(第16 d), 不同的氮输入条件下Biolog-Eco板在培养204 h后AWCD值呈现出一定差异, 其中, 连续性氮输入组的AWCD值显著升高, 证明了间歇性的持续氮输入会提高寡营养海水中细菌的代谢活性, 相似的结果也在海水中可溶性有机氮与细菌活性相关性的研究中被报道(Stepanauskas et al., 1999).此外, 在微宇宙中单一地、连续性地输入氮源, 造成寡营养条件下的氮磷比出现持续性的严重失衡, 可能导致了细菌群落对碳源的利用模式发生变化(Leflaive et al., 2008).
基于碳源利用情况表征微生物群落功能多样性的短期变化具有较好的灵敏性(Gomez et al., 2006), 然而Biolog-Eco板的颜色变化主要取决于丰度或生长速率较高的物种(Almutairi, 2009), 且该方法只能通过代表性碳源来相对地评价潜在代谢多样性(Gomez et al., 2006), 31种单一碳源不能精确地表征生态系统中底物的复杂性, 因此, 可能会低估细菌群落的实际代谢水平(章家恩等, 2004).细菌群落存在功能冗余(Allison et al., 2008), 许多物种同时具有利用特定碳源的潜力, 因此, 碳源利用模式可能对细菌群落物种组成的变化不敏感, 导致不同的氮输入条件下细菌群落代谢多样性没有显著变化(表 2), 这与前人(Konopka et al., 1998)研究发现基于碳源利用模式表征的代谢多样性可能变化很小的结果相类似.微宇宙中单一的氮输入既改变了氮磷比和碳氮比, 也改变了物种共存的环境, 进而可能导致微宇宙中海水细菌群落的功能多样性发生变化, 例如, 有研究利用Biolog-Eco技术发现, 氮磷比调控改变了中度营养湖泊微宇宙(Microcosm)系统中细菌群落的功能多样性(Leflaive et al., 2008), 而在对虾-蟹混养系统中不同的碳氮比对水体细菌群落的功能多样性有明显影响(李杰等, 2015).寡营养环境中过量营养物质的输入对一些原本具有较高代谢能力的特定类群可能产生一定的抑制作用(Allison et al., 2008), 而Biolog-Eco板培养的细菌只是整个群落中的一部分, 仅表征快速生长或丰度较高的细菌的活性, 无法全面表征寡营养海水中普遍存在的、生长缓慢的细菌的活性, 因此, 只能揭示细菌群落局部的代谢特性(Wang et al., 2008).这些因素可能部分地解释了本研究中细菌群落代谢能力和多样性总体上对过量氮输入的不敏感性.
4.2 过量氮输入对氮代谢相关基因的影响PICRUSt预测的功能基因家族组成随采样时间显著变化且与水环境因子显著相关, 这可能与水环境因子的时间变化有关, 这些环境因子在驱动微生物类群组成随时间演替的同时(Dong et al., 2017), 也可能导致功能基因家族组成的变异, 从而对整个群落的代谢潜力产生影响(Lebaron et al., 2001).此外, 尽管总体上过量氮输入对除nrfA以外的氮代谢基因没有显著影响(表 4), 但在实验4 d后氮输入对nifDKH、nirK、nasAB等氮代谢基因有显著影响, 且连续氮输入的影响较一次性输入更为普遍(图 7), 这说明氮输入对氮代谢基因相对丰度的影响有时间性, 且高频率氮输入可能比高强度氮输入的微生态效应更大.
氮输入在一定程度上影响了微宇宙中的固氮、硝化、异化硝酸盐还原、反硝化、异化硝酸盐还原到铵和同化硝酸盐还原途径的关键基因(图 7).实验起始阶段, 由于本研究所取海水未进行预过滤, 浮游动植物死亡造成铵氮浓度显著升高(Dong et al., 2017), 进而可能导致固氮基因nifDKH的相对丰度显著下降.硝酸盐作为异化硝酸盐还原酶的限制因子, 是异化硝酸盐还原酶的诱导剂(李小平等, 2010), 本研究中持续性氮输入使得微宇宙中硝酸盐含量持续增多, 可能导致实验中期(第8 d)连续性输入组中异化硝酸盐还原酶编码基因napAB相对丰度显著升高;但实验后期(第16 d)微宇宙内过量的硝酸盐积累可能抑制了异养还原菌的生长, 从而使得napAB相对丰度显著降低(图 7b).溶解氧、碳氮比和碳源都是异化硝酸盐还原反应的影响因子, 在好氧条件下, 碳氮比和碳源成为影响异化硝酸盐还原反应的关键因子(刘佳, 2007).已有研究发现, 在非低氧条件下也有一些微生物拥有异化硝酸盐还原的相关基因(Roberts et al., 2014; Roco et al., 2016).尽管有研究利用宏转录组测序发现, 在近岸非低氧区相关功能基因也能表达(Satinsky et al., 2014), 但本研究结果仅代表该群落可能有相关功能潜力.已有研究表明, 同化硝酸盐还原酶是生物体对于硝酸盐新陈代谢过程中的主要调节酶, 在硝氮适度输入时会被诱导合成(孟智奇, 2010), 这与本研究中连续氮输入早期(第4 d)同化硝酸盐还原酶编码基因nasAB相对丰度显著高于对照组相符.此外, 持续氮输入导致微宇宙中碳氮比逐渐减小, 使反硝化相关基因无法获得足够的碳源合成反硝化酶系(李卫芬等, 2014), 这可能部分解释了实验第16 d反硝化关键基因nirK的相对丰度较对照组显著下降(图 7b).
PICRUSt预测结果的准确度可以用NSTI值表征, 其准确性通常随NSTI值的增加而降低.但有研究发现土壤样品的NSTI平均值为0.17时, 其预测的准确度仍较高, 而在未知多样性较高的环境中, 如高盐环境中NSTI平均值为0.23(Langille et al., 2013), 其预测结果准确度则较差.本研究中NSTI平均值为0.155, 加上海洋环境中可能蕴含大量的未知细菌多样性, 因而预测结果可能较粗略.本研究结果只初步探索和预测了过量氮输入对寡营养海水细菌群落局部代谢活性和潜力的影响, 今后的工作将进一步借助荧光定量PCR、宏基因组和宏转录组测序等技术深入研究过量氮输入对海洋细菌群落功能代谢潜力和活性的影响.
5 结论(Conclusions)1) 本研究通过基于寡营养海水生态微宇宙实验发现, 连续性氮输入改变了细菌群落的碳源利用能力, 其碳源利用特征随时间变化.
2) 过量氮输入影响了固氮、反硝化和硝酸盐还原相关基因的相对丰度, 而连续性氮输入下氮代谢相关基因的变化较一次性输入更为普遍, 为探明近岸海域过量氮输入的微生态效应提供了基础数据.
3) 本研究中建立的小型生态微宇宙, 尽管每次输入氮源时考虑了培养体积的减小, 但培养体积减小可能会导致微宇宙系统稳定性降低、时间变异增大等问题.利用Biolog-Eco板仅能表征细菌群落的局部功能活性, 而16S rDNAPICRUSt预测仅能大略表征群落功能潜力.针对这些不足之处, 今后将利用原位中宇宙实验来对本研究结果加以验证, 并利用宏基因组和宏转录组测序来深入研究细菌群落的功能潜力和活性.
Allison S D, Martiny J B H. 2008. Resistance, resilience, and redundancy in microbial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 105(S1): 11512–11519.
|
Almutairi N Z. 2009. Variable distributional characteristics of substrate utilization patterns in activated sludge plants in Kuwait[J]. Bioresource Technology, 100(4): 1524–1532.
DOI:10.1016/j.biortech.2008.06.014
|
Arrigo K R. 2005. Marine microorganisms and global nutrient cycles[J]. Nature, 437(7057): 349–355.
DOI:10.1038/nature04159
|
Bell C W, Acostamartinez V, Mcintyre N E, et al. 2009. Linking microbial community structure and function to seasonal differences in soil moisture and temperature in a Chihuahuan Desert grassland[J]. Microbial Ecology, 58(4): 827–842.
DOI:10.1007/s00248-009-9529-5
|
Carlsson P, Granéli E, Granéli W, et al. 2012. Bacterial and phytoplankton nutrient limitation in tropical marine waters, and a coastal lake in Brazil[J]. Journal of Experimental Marine Biology and Ecology, s 418-419(3): 37–45.
|
Castro-González M, Braker G, Farías L, et al. 2005. Communities of nir S-type denitrifiers in the water column of the oxygen minimum zone in the eastern South Pacific[J]. Environmental Microbiology, 7(9): 1298–1306.
DOI:10.1111/emi.2005.7.issue-9
|
Cookson W R, Murphy D V, Roper M M. 2008. Characterizing the relationships between soil organic matter components and microbial function and composition along a tillage disturbance gradient[J]. Soil Biology and Biochemistry, 40(3): 763–777.
DOI:10.1016/j.soilbio.2007.10.011
|
Falkowski P G, Fenchel T, Delong E F. 2008. The microbial engines that drive Earth's biogeochemical cycles[J]. Science, 320(5879): 1034–1039.
DOI:10.1126/science.1153213
|
Feuerpfeil P, Rieling T, Estrum-Youseff S R, et al. 2004. Carbon budget and pelagic community compositions at two coastal areas that differ in their degree of eutrophication, in the Southern Baltic Sea[J]. Estuarine Coastal and Shelf Science, 61(1): 89–100.
DOI:10.1016/j.ecss.2004.04.006
|
Giaramida L, Manage P M, Edwards C, et al. 2013. Bacterial communities' response to microcystins exposure and nutrient availability:Linking degradation capacity to community structure[J]. International Biodeterioration and Biodegradation, 84: 111–117.
DOI:10.1016/j.ibiod.2012.05.036
|
Gomez E, Ferreras L, Toresani S. 2006. Soil bacterial functional diversity as influenced by organic amendment application[J]. Bioresource Technology, 97(13): 1484–1489.
DOI:10.1016/j.biortech.2005.06.021
|
Han X M, Wang R Q, Liu J, et al. 2007. Effects of vegetation type on soil microbial community structure and catabolic diversity assessed by polyphasic methods in North China[J]. Journal of Environmental Sciences, 19(10): 1228–1234.
DOI:10.1016/S1001-0742(07)60200-9
|
Kong L, Jing H, Kataoka T, et al. 2011. Phylogenetic diversity and spatio-temporal distribution of nitrogenase genes (nif H) in the northern South China Sea[J]. Aquatic Microbial Ecology, 65(1): 15–27.
DOI:10.3354/ame01531
|
Konopka A, Oliver L, Turco R F. 1998. The use of carbon substrate utilization patterns in environmental and ecological microbiology[J]. Microbial Ecology, 35(2): 103–115.
DOI:10.1007/s002489900065
|
Langille M G I, Zaneveld J, Caporaso J G, et al. 2013. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 31(9): 814–821.
DOI:10.1038/nbt.2676
|
Lebaron P, Servais P, Troussellier M, et al. 2001. Microbial community dynamics in Mediterranean nutrient-enriched seawater mesocosms:changes in abundances, activity and composition[J]. FEMS Microbiology Ecology, 34(3): 255–266.
DOI:10.1111/fem.2001.34.issue-3
|
Leflaive J, Danger M, Lacroix G, et al. 2008. Nutrient effects on genetic and functional diversity of aquatic bacterial communities[J]. FEMS Microbiology Ecology, 66(2): 379–390.
DOI:10.1111/fem.2008.66.issue-2
|
李丹, 李忠磊, 邵雷, 等. 2015. 细菌硝酸盐还原中的关键酶影响细菌耐药性的研究进展[J]. 中国抗生素杂志, 2015, 40(7): 481–485.
|
李杰, 田相利, 董双林, 等. 2015. 碳菌调控对虾、蟹混养系统微生物群落功能多样性的影响[J]. 河北渔业, 2015(8): 3–11.
|
李卫芬, 郑佳佳, 张小平, 等. 2014. 反硝化酶及其环境影响因子的研究进展[J]. 水生生物学报, 2014, 38(1): 166–170.
|
李小平, 方涛, 敖鸿毅, 等. 2010. 东湖沉积物中dNaR活性和硝酸盐还原菌的垂向分布[J]. 中国环境科学, 2010, 30(2): 228–232.
|
刘佳. 2007. 微生物好氧硝酸盐还原产铵研究[D]. 成都: 四川大学
http://cdmd.cnki.com.cn/Article/CDMD-10610-2008028005.htm |
梅冰, 彭绪亚, 谢影. 2016. 采用Biolog法分析餐厨垃圾厌氧消化微生物群落多样性[J]. 中国沼气, 2016, 34(1): 14–18.
|
孟智奇. 2010. 长三角地区饮用水源污染水体的微生物修复研究[D]. 杭州: 浙江大学
http://cdmd.cnki.com.cn/article/cdmd-10335-2010057136.htm |
Metcalf J L, Xu Z Z, Weiss S, et al. 2016. Microbial community assembly and metabolic function during mammalian corpse decomposition[J]. Science, 351(6269): 158–162.
DOI:10.1126/science.aad2646
|
Meyer-Reil L A, Köster M. 2000. Eutrophication of marine waters:Effects on benthic microbial communities[J]. Marine Pollution Bulletin, 41(1/6): 255–263.
|
Nogales B, Lanfranconi M P, Piñavillalonga J M, et al. 2011. Anthropogenic perturbations in marine microbial communities[J]. FEMS Microbiology Reviews, 35(2): 275–298.
DOI:10.1111/j.1574-6976.2010.00248.x
|
Paerl H W, Juliannedyble, Moisander P H, et al. 2003. Microbial indicators of aquatic ecosystem change:current applications to eutrophication studies[J]. FEMS Microbiology Ecology, 46(3): 233–246.
DOI:10.1016/S0168-6496(03)00200-9
|
Polónia A R M, Cleary D F R, Freitas R, et al. 2014. The putative functional ecology and distribution of archaeal communities in sponges, sediment and seawater in a coral reef environment[J]. Molecular Ecology, 24(2): 409–423.
|
秦旭, 高文胜, 周文杰, 等. 2014. 控释肥对苹果园土壤微生物碳源利用能力的影响[J]. 果树学报, 2014(5): 809–814.
|
Roberts K L, Kessler A J, Grace M R, et al. 2014. Increased rates of dissimilatory nitrate reduction to ammonium (DNRA) under oxic conditions in a periodically hypoxic estuary[J]. Geochimica et Cosmochimica Acta, 133(6): 313–324.
|
Roco C A, Bergaust L L, Shapleigh J P, et al. 2016. Reduction of nitrate to nitrite by microbes under oxic conditions[J]. Soil Biology and Biochemistry, 100: 1–8.
DOI:10.1016/j.soilbio.2016.05.008
|
Satinsky B M, Crump B C, Smith C B, et al. 2014. Microspatial gene expression patterns in the Amazon River Plume[J]. Proceedings of the National Academy of Sciences of the United States of America, 111(30): 11085–11090.
DOI:10.1073/pnas.1402782111
|
Stenrod M, Charnay M P, Benoit P, et al. 2006. Spatial variability of glyphosate mineralization and soil microbial characteristics in two Norwegian sandy loam soils as affected by surface topographical features[J]. Soil Biology and Biochemistry, 38(5): 962–971.
DOI:10.1016/j.soilbio.2005.08.014
|
Stepanauskas R, Edling H, Tranvik L J. 1999. Differential dissolved organic nitrogen availability and bacterial aminopeptidase activity in limnic and marine waters[J]. Microbial Ecology, 38(3): 264–272.
DOI:10.1007/s002489900176
|
孙凤霞, 张伟华, 徐明岗, 等. 2010. 长期施肥对红壤微生物生物量碳氮和微生物碳源利用的影响[J]. 应用生态学报, 2010, 21(11): 2792–2798.
|
谭兆赞, 刘可星, 廖宗文. 2006. 土壤微生物BIOLOG分析中特征碳源的判别[J]. 华南农业大学学报, 2006, 27(4): 10–13.
DOI:10.7671/j.issn.1001-411X.2006.04.003 |
Wang G, Liu J, Qi X, et al. 2008. Effects of fertilization on bacterial community structure and function in a black soil of Dehui region estimated by Biolog and PCR-DGGE methods[J]. ActaEcologica Sinica, 28(1): 220–226.
|
Wang K, Ye X, Zhang H, et al. 2016. Regional variations in the diversity and predicted metabolic potential of benthic prokaryotes in coastal northern Zhejiang, East China Sea[J]. Scientific Reports: 6.
DOI:10.1038/srep38709
|
席劲瑛, 胡洪营, 钱易. 2003. Biolog方法在环境微生物群落研究中的应用[J]. 微生物学报, 2003, 43(1): 138–141.
|
夏雪, 谷洁, 车升国, 等. 2011. 施氮水平对塿土微生物群落和酶活性的影响[J]. 中国农业科学, 2011, 44(8): 1618–1627.
DOI:10.3864/j.issn.0578-1752.2011.08.010 |
章家恩, 蔡燕飞, 高爱霞, 等. 2004. 土壤微生物多样性实验研究方法概述[J]. 土壤, 2004, 36(4): 346–350.
|
 2018, Vol. 38
2018, Vol. 38


