近年来, 随着中国经济迅速发展, 纺织印染行业也逐渐壮大, 有关研究报告称, 世界染料总产量超过80万t·a-1, 种类超过10万种, 中国年产量占世界年产量的40%, 而且每年以30%的比例持续增长(Fan et al., 2009).偶氮染料不仅种类繁多, 而且应用广泛.目前, 偶氮染料已被广泛应用于纺织印染、食品、造纸印刷等行业(Ulson de Souza et al., 2007), 占总纺织染料使用量的70%左右(Lang et al., 2013), 是各行业中应用最为广泛的一类合成染料.
含有偶氮键的染料统称为偶氮染料, 是染料中最多的一类.以杂环化合物作为重氮组分或偶合组分的偶氮染料, 主要有分散染料、酸性染料和活性染料.偶氮染料色彩鲜艳、分子吸光系数大、牢度较好, 是当前分散、酸性染料、活性染料的主要发展方向.一般说来, 偶氮化合物的分子中含有1~3个“―N=N―”, 键上连有苯或苯基, 苯或苯基又连有―Cl、―NH2、―CH3、―SO3、―NO2及―OH等基团(Saratale et al., 2011).偶氮分子的发色基是偶氮键, 助色基是氨基、羟基、甲基和磺酸基等, 当光线射入后发生选择性吸收而产生视觉所感受到的各种颜色.偶氮染料的使用量在纺织印染行业最大, 产生的纺织印染废水量也最多.含偶氮染料的纺织印染废水具有色度高、化学稳定性强和生物可降解性差的特点(Supaka et al., 2004), 而且其本体及其降解产物苯胺类中间体具有毒性、致突变和致癌性(Pandey et al., 2007), 是国内外公认的难处理的废水之一.这些有毒物质长期存在于水环境中, 对人类及动植物的健康会造成极大危害(Lima et al., 2007), 因而筛选能够高效降解偶氮染料的微生物, 并将其应用到印染废水的处理已经成为国内外研究的热点之一.
国内外研究人员已发现了多种微生物对偶氮染料有较强的脱色能力, 细菌、真菌、酵母菌、藻类及某些植物都被检测到可以降解或矿化各种染料使其脱色.偶氮染料分子的发色基是偶氮键(—N=N—), 偶氮染料的脱色是通过偶氮键断裂来实现(Nam et al., 2000).一般认为, 微生物并不能把大分子偶氮染料当作基质吸收利用, 而是通过合成一种特定的偶氮还原酶, 在其催化作用下将发色基团偶氮键还原成无色的胺类物质(严滨等, 2008; dos Santos et al., 2007).脱色细菌因其在环境中普遍存在, 培养条件简单, 繁殖迅速, 在工业微生物领域应用广泛而被高度重视.例如, Dawkar等(2009)报道的偶氮染料脱色细菌Bacillus sp. VUS, 在18 h的脱色过程中, 可以对50 mg·L-1的偶氮染料Navy Blue 2G进行有效脱色, 且脱色率达到94%;Jadhav等(2008)报道的偶氮染料脱色细菌Comamonas sp. UVS, 在13 h的脱色过程中, 可以对1.1 g·L-1的偶氮染料Direct Red 5B进行有效脱色, 且脱色率高达到100%;Kalyani等(2009)报道的偶氮染料脱色菌Pseudomonas sp. SUK1, 在6 h的脱色过程中, 可以对高浓度(5 g·L-1)的偶氮染料Reactive Red 2进行有效脱色, 且脱色率达到96%;Saratale等(2009)报道的Micrococcus glutamicus NCIM-2168, 在42 h脱色过程中, 可以对50 mg·L-1的偶氮染料Reactive Green 19 A进行有效脱色, 且脱色率达到100%;Amar等(2008)报道的 Rhizobium radiobacter MTCC 8161, 在48 h脱色过程中, 可以对50 mg·L-1的偶氮染料Reactive Red 141进行有效脱色, 且脱色率达到90%.但关于嗜水气单胞菌属(Aeromonas hydrophila)对偶氮染料酸性橙7的脱色研究鲜有报道.
因此, 本实验利用平板划线分离法从杭州七格污水处理厂二沉池的活性污泥中筛选得到一株对偶氮染料酸性橙7(Acid Orange 7)具有高效脱色功能的菌株嗜水气单胞菌(Aeromonas hydrophila strain JL-1), 通过16S rRNA基因序列分析鉴定, 并初步探讨该菌株的最佳脱色条件及其脱色性能, 为进一步丰富高效偶氮脱色菌的基因库, 进行印染工业废水的处理及偶氮染料降解机理的研究提供参考依据.
2 材料与方法(Materials and methods) 2.1 偶氮染料酸性橙7(Acid Orange 7, 简称AO7), 又称为金橙Ⅱ(OrangeⅡ), CAS号633-96-5, 分子式为C16H11N2NaO4S, 相对分子为350.32, 特征波长λmax=484 nm, 分子结构示意图如图 1所示.本产品购置于杭州某化学药剂公司, 纯度>85%, 属于偶氮酸性染料, 主要用于酸碱指示剂、生物染色剂、抽提和分光测定阳离子表面活性剂.
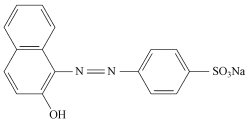 |
| 图 1 酸性橙7(AO7)分子结构示意图 Fig. 1 Chemical structure of Acid Orange 7 (AO7) |
选用LB培养基(Luria-Bertani medium)用于菌株的培养, 再选取另外5种不同类型培养基用于菌株的脱色实验, 其培养基组成成分如表 1所示.所使用的培养基均由50 g·L-1的NaHCO3溶液调节pH至7.0, 并在121 ℃下灭菌20 min, 待培养基降到60 ℃后, 加入偶氮染料AO7.
| 表 1 菌株JL-1的脱色培养基组成 Table 1 Decolorization Culture composition of strain JL-1 |
菌体在液体培养基培养至对数期, 取1 mL菌液,使用上海申能博彩生物科技有限公司生产的3S柱离心式环境样品DNA抽提试剂盒提取总DNA.扩增菌株16S rRNA基因, 使用细菌通用引物对(正向引物27F 5′-AGAGTTYGATCCTGGCTCAG-3′; 反向引物1492R 5′-AAGGAGGTGATCCAGCCGCA-3′).PCR反应条件为:95 ℃预变性5 min; 95 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸1 min 30 s, 30个循环; 72 ℃保温10 min, 4 ℃保存.将测序所得基因序列提交至NCBI数据库,使用BLAST进行同源性比对, 并下载相关序列, 利用系统发育树分析软件MEGA5.0计算序列相似度, 同时用其中的Neighbor Joining(NJ)方法构建菌株的系统发育树并对序列进行亲缘性及系统发育分析.
2.4 脱色实验配置100 mL含有100 mg·L-1偶氮染料AO7的培养基于250 mL的锥形瓶中, 按6%的接种量接种菌株JL-1(接种液OD600=1.5), 在温度为30 ℃、转速为150 r·min-1的恒温振荡器中进行接种培养24 h.探讨环境条件对菌株JL-1脱色性能及其生长情况的影响, 包括初始染料浓度(20、50、100、150和200 mg·L-1)、接种量(2%、4%、6%、8%和10%)、pH(5、6、7、8和9)和NaCl浓度(0、5、10、30和50 g·L-1).
2.5 分析方法使用721可见分光光度计对偶氮染料AO7与菌株JL-1进行定量分析, 分析方法参照文献(Chen, 2002).所取混合样品一部分直接在600 nm的波长下测定吸光度OD(optical density), 另一部分经10 min、13000 r·min-1离心(H1850R台式高速冷冻离心机), 上清液在波长600 nm和λmax=484 nm下分别进行吸光度测定, 菌株浓度用样品离心前(式(1))、后(式(2))混合液在600 nm下的吸光度的差值表征, 染料浓度用样品离心后上清液在λmax下的吸光度表征(式(3)).
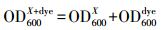
|
(1) |
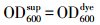
|
(2) |
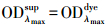
|
(3) |
式中, OD600X+dye为样品混合液在600 nm下吸光度值; OD600X为细菌菌液在600 nm下吸光度值, 用于表征细菌浓度; OD600sup与OD600dye为样品上清液在600 nm下染料的吸光度值; ODλmaxsup与ODλmaxdye为样品上清液在最大吸收波长下染料的吸光度值;
用脱色率表征菌株JL-1对染料AO7的脱色能力(吸附与降解能力), 根据公式(4)计算脱色率η.
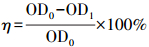
|
(4) |
式中, OD0为培养基初始的吸光度值; OD1为反应一段时间后的吸光度值.
3 结果与讨论(Results and discussion) 3.1 菌株的分离筛选采用平板划线分离法从杭州七格污水处理厂二沉池的活性污泥中筛选得到一株对偶氮染料酸性橙7(AO7)具有高效脱色功能的菌株, 编号为JL-1.该菌株在LB琼脂培养基上生长良好, 兼性厌氧, 形成边缘整齐、表面湿润、光滑的圆形菌落, 菌落呈半透明、灰白色, 有特殊气味, 菌落的大小与培养时间、温度有关, 菌落小的只有针尖大小, 大的直径可达3~4 mm, 菌落照片如图 2所示.
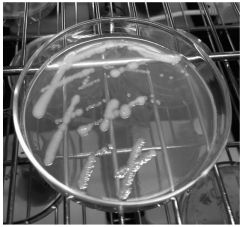 |
| 图 2 菌株JL-1菌落照片 Fig. 2 Colony image of strain JL-1 |
测序获得菌株JL-1的16S rRNA基因序列(1153 bp), 将测序获得的正反序列经RNAMAN序列拼接后, 在NCBI数据库中进行同源性比对, 获得Blast序列登录号为XMACAS5301R.选取Blast中已有的嗜水气单胞菌(Aeromonas hydrophila)序列相近的标准菌株, 并下载相关序列, 利用软件MEGA5.0计算序列相似度, 同时用其中的Neighbor Joining(NJ)方法构建菌株的系统发育树并对序列进行亲缘性及系统发育分析.该菌株的16S rRNA基因序列的系统发育树如图 3所示.结果表明, 菌株JL-1基因与Aeromonas hydrophila strain S5-14相似度最高, 同源性高达99%.并且在所构建的系统发育树中, 菌株JL-1与Aeromonas hydrophila strain S5-14也处于同一分支上, 因此, 结合其形态特征, 将该菌鉴定并初步命名为Aeromonas hydrophila strain JL-1.
 |
| 图 3 菌株JL-1的16S rRNA基因序列系统发育树(括号内序号为Blast登录号, 数字为同源性) Fig. 3 Phylogenetic tree derived from 16S rRNA gene sequence of strain JL-1(The serial number in the parentheses denotes the Blast accession Number, and numeral represents comparability) |
由图 4可知,菌株JL-1对100 mg·L-1偶氮染料酸性橙7(AO7)脱色因选用培养基不同而脱色率各异, 在10 h脱色过程中, Czapek′s medium、Martin Broth、Mineral salt medium、M9 medium、Sabourand medium 5种培养基的脱色率达不到40%, 而Luria-Bertani medium的脱色率高达94.7%.究其原因, 可能是因为Luria-Bertani medium所含营养物质(胰蛋白胨、酵母浸膏)均是复杂的有机含氮化合物, 能提供菌株生长所需的碳源和氮源, 而且酵母浸膏作为合作基质可以促进偶氮染料脱色(Muhammad et al., 2016), 但其余5种培养基所含的营养物质均为无机氮源, 不利于菌株JL-1对偶氮染料AO7脱色.因此, 在后续的实验中, 选择Luria-Bertani medium作为偶氮染料的脱色培养基.
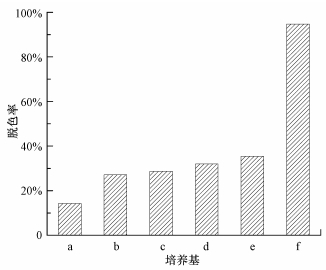 |
| 图 4 培养基对菌株JL-1脱色性能的影响(a.Czapek′s medium, b.Martin Broth, c.Mineral salt medium, d.M9 medium, e.Sabourand medium, f. Luria-Bertani medium) Fig. 4 Effect of medium on decolorization of Acid Orange 7 by strain JL-1 |
本实验为了探讨菌株JL-1对偶氮染料AO7的脱色能力, 设计了5组不同初始AO7浓度的LB脱色培养基, 用于观察初始染料浓度对菌株脱色性能及其生长的影响.菌株JL-1对不同浓度的偶氮染料AO7脱色情况如图 5a所示, 增殖情况如图 5b所示.图 5a表明, 在10 h脱色过程中, 菌株JL-1对偶氮染料AO7的脱色效果受初始染料浓度的影响, 对于低浓度(20 mg·L-1)和中低浓度(50 mg·L-1)的偶氮染料AO7, 菌株JL-1在4 h内完成78.3%和88.7%的脱色, 在后续6 h脱色过程中脱色率几乎没有变化; 对于中等浓度(100 mg·L-1)的偶氮染料AO7, 菌株JL-1在8 h内完成93.8%的脱色, 在后续2 h培养过程中脱色率几乎没有变化; 对于中高浓度(150 mg·L-1)和高浓度(200 mg·L-1)的偶氮染料AO7, 菌株JL-1在10 h内只完成84.7%和68.3%的脱色.当初始染料浓度在20~100 mg·L-1之间时, 菌株JL-1的脱色效率随着AO7浓度增加而增加, 初始染料浓度在100~200 mg·L-1之间时, 菌株JL-1的脱色效率随着AO7浓度增加而减小.图 5b表明, 在前4 h培养过程中, AO7浓度对菌株JL-1的生长没有明显影响, 在后6 h培养过程中, 随着初始染料AO7浓度的升高, 其OD600值增加幅度逐渐减小, 在高浓度(200 mg·L-1)的AO7环境下生长的菌株增殖速率下降最快.
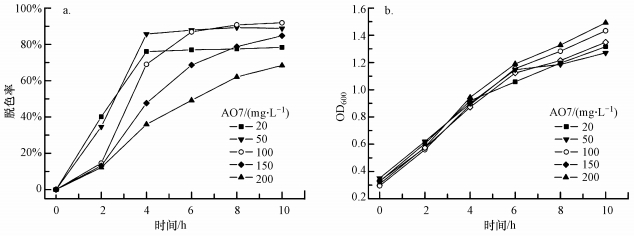 |
| 图 5 初始染料浓度对菌株JL-1脱色(a)及生长(b)的影响 Fig. 5 Effect of initial dye (Acid Orange 7) concentration on decolorization(a) and growth(b) of strain JL-1 |
菌株JL-1对100 mg·L-1偶氮染料AO7在8 h的脱色率达到93.8%, 相比于Tan等(2013;2014;2016)研究中所述降解20 mg·L-1偶氮染料AO7的脱色菌株, 菌株JL-1的脱色能力与效率明显均要高于前者.Chen(2003)研究的菌株Aeromonas hydrophila DEC1对100 mg·L-1偶氮染料AO7在24 h的脱色率达到79%, 其脱色菌株DEC1与菌株JL-1均属于嗜水气单胞菌属, 但针对同一浓度(100 mg·L-1)偶氮染料AO7的降解, 其脱色效率要比菌株JL-1逊色许多.
脱色菌株JL-1对于各种浓度梯度的偶氮染料AO7均达不到100%脱色, 而且低浓度AO7染料脱色会受到抑制, 随着初始染料浓度越高, 脱色速率会越小, 抑制现象也越明显, 脱色到一定程度后将很难再进行下去.探究其原因可能是本体偶氮染料及其分解产生的具有毒性的中间体抑制了脱色菌株生长和偶氮还原酶合成(Tony et al., 2009; Jadhav et al., 2011), 使得其脱色能力下降, 图 5b的菌株增殖趋势也证实这一点, 随着初始偶氮染料AO7浓度的增加, 菌株JL-1的增殖速率在不断下降, 偶氮染料AO7会抑制菌株的生长.在偶氮染料诱导之下, 脱色菌被诱导合成偶氮还原酶, 将AO7分解成对氨基苯磺酸和1-氨基-2-萘酚(García-Martínez et al., 2015).染料浓度越高, 分解的代谢产物量将越多, 对菌株活性的抑制也更强, 脱色率下降将显而易见.因此, 本实验选取100 mg·L-1偶氮染料AO7作为菌株JL-1最佳脱色的初始染料浓度.
3.5 接种量对菌株脱色性能及生长的影响菌体初始接种量的大小不仅直接影响生物脱色效率, 而且与工程应用的成本息息相关.为了保证染料有效脱色, 同时又能把成本降到最低, 本实验设置了一系列的接种量用于探讨pH对菌株JL-1脱色性能及生长的影响.不同接种量的菌株JL-1对100 mg·L-1偶氮染料酸性橙7(AO7)脱色情况如图 6a所示, 生长情况如图 6b所示.在8 h的脱色过程中, 接种量为2%时的脱色率仅为68.7%, 接种量为4%时的脱色率为77.3%, 接种量为6%时的脱色率为80.5%, 接种量为8%时的脱色率达到90.8%, 接种量为10%时的脱色率达到92.7%.结果表明, 菌株JL-1的脱色效率受接种量大小的影响, 接种量越大, 菌株生长速率越快, 脱色效果也越好.菌株JL-1以二分裂方式增殖, 接种量越大, 菌体的初始浓度也将越高, 其脱色率和增殖速率也会越高(Tuttolomondo et al., 2014), 为了保证脱色效果, 接种量至少应为8%, 这与Tan等(2014)得出的结论相吻合.因此, 本实验选取8%的接种量作为菌株JL-1的最佳接种量.
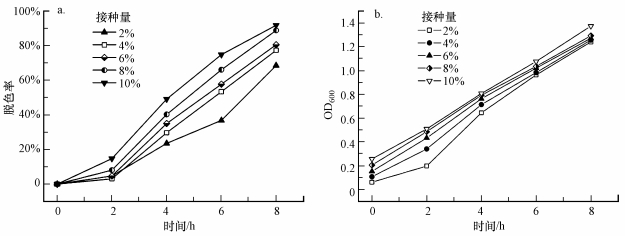 |
| 图 6 接种量对菌株JL-1脱色(a)及生长(b)的影响 Fig. 6 Effect of inoculation size on decolorization(a) and growth(b) of strain JL-1 |
考虑到纺织印染废水pH值较高, 本实验设置了5组pH梯度用于探讨pH对菌株JL-1脱色性能及生长的影响.所处不同pH环境下的菌株JL-1对100 mg·L-1偶氮染料酸性橙7(AO7)的脱色情况如图 7a所示, 生长情况如图 7b所示.在8 h的脱色过程中, pH为5的培养基脱色率达到92.1%, pH为6的培养基脱色率达到91.6%, pH为7的培养基脱色率达到75.9%, pH为8的培养基脱色率达到69.2%, pH为9的培养基脱色率仅有29.1%.结果表明, 菌株JL-1更适合在偏酸性环境对偶氮染料AO7进行脱色, 在偏碱性环境下它的生长与脱色都会受到抑制(于文娟等, 2012).这一结论与Chen等(2003)报道的嗜水气单胞菌在偏酸性环境对活性深蓝NR脱色结果相似.因此, 本实验选取pH为5的偏酸性环境作为菌株JL-1的最佳脱色环境.
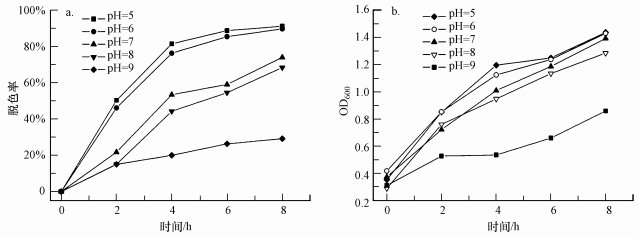 |
| 图 7 pH对菌株JL-1脱色(a)及生长(b)的影响 Fig. 7 Effect of pH on decolorization(a) and growth(b) of strain JL-1 |
微生物对染料的脱色主要是通过生物吸附和生物降解两个过程实现, 这两个过程与pH值的大小密切相关, 它不仅可以改变染料结构和电荷而影响微生物细胞对染料的吸附(Wang et al., 2008), 而且还可以通过改变脱色菌合成的偶氮还原酶活性间接影响脱色效率.在偏酸性的环境下, 偶氮染料AO7的磺酸基更容易与H+结合, 使得偶氮染料AO7分子间产生静电斥力, 从而促进微生物细胞对染料的吸附作用(Konsowa et al., 2011; Qu et al., 2010); 在偏碱性环境下, 较高的pH值会使得菌株JL-1产生偶氮还原酶失去活性, 从而影响到菌株的脱色效率.
3.7 盐度对菌株脱色性能及生长的影响考虑到纺织印染废水具有很高的含盐量, 因此, 本实验设置5组含有不同NaCl浓度梯度的LB培养基, 用NaCl含量表示盐度的高低, 探讨盐度对菌株JL-1脱色性能及生长的影响.所处不同盐度环境下的菌株JL-1对100 mg·L-1偶氮染料酸性橙7(AO7)的脱色情况如图 8a所示, 生长情况如图 8b所示.在10 h的脱色过程中, 未加NaCl(即盐度为0)的培养基脱色率仅有63.1%, NaCl浓度为5 g·L-1(盐度为5%)的培养基脱色率达到76.7%, NaCl浓度为10 g·L-1(盐度为10%)的培养基脱色率达到93.6%, NaCl浓度为30 g·L-1(盐度为30%)的培养基脱色率仅有55.5%, NaCl浓度为50 g·L-1(盐度为50%)的培养基脱色率仅有20.2%.显而易见, 过低或过高浓度的NaCl都会影响菌株JL-1的脱色效果, 在NaCl浓度较低(0~10 g·L-1)时, 菌株JL-1对偶氮染料AO7的脱色能力随着NaCl浓度升高而加强, 这与Gou等(2009)发现盐度从0增加到6%时青霉菌QQ对偶氮染料活性深红X-3B脱色效果明显增强的现象一致; 在NaCl浓度较高(10~50 g·L-1)时, 菌株JL-1对偶氮染料AO7的脱色能力随着NaCl浓度升高而减弱, NaCl浓度为10 g·L-1的培养基脱色效果最佳.推测其原因可能是菌株JL-1在高浓度NaCl的培养基里活性受到抑制, 盐析作用增强, 脱氢酶的活性下降, 微生物本身活性受阻, 新陈代谢作用减缓, 导致脱色率下降.这一推测也从图 8b得到证实, 菌株JL-1在NaCl浓度为50 g·L-1的培养基里的OD600值达不到1.2, 明显低于其他NaCl浓度下的OD600值, 表明菌株JL-1在高盐度条件下的生长受到抑制.另外, 脱色菌JL-1合成的偶氮还原酶活性高低将直接影响偶氮染料的脱色效率, 盐度过高会使得偶氮还原酶变性.因此, 本实验选取NaCl浓度为10 g·L-1的低盐度环境作为菌株JL-1的最佳脱色环境.
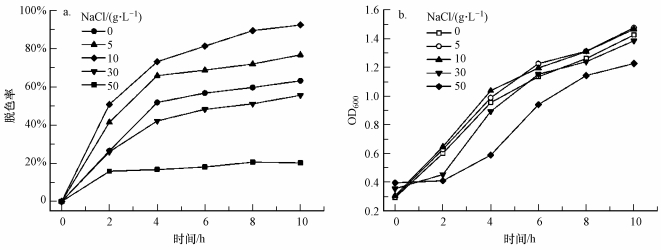 |
| 图 8 NaCl浓度对菌株JL-1脱色(a)及生长(b)的影响 Fig. 8 Effect of NaCl concentration on decolorization(a) and growth (b) of strain JL-1 |
事实上, 盐度对微生物细胞的渗透压有极大影响, 高盐度环境会抑制细菌生长, 甚至导致细胞死亡(Kolekar et al., 2008).染料的制造和印染工序产生的废水盐度一般在15%~20%之间(Manu et al., 2003), 一般来说, NaCl浓度超过3 g·L-1就可以对大多数细菌活性造成中等程度的抑制(郭建博等, 2007; 邹聪慧等, 2011; Nachiyar et al., 2007; Gou et al., 2010).然而, 菌株JL-1在NaCl浓度为10 g·L-1条件下生长良好, 对偶氮染料AO7的脱色率高达93.6%, 不存在抑制现象, 由此可见, 菌株JL-1具有一定程度的耐盐性.
4 结论(Conclusions)1) 采用平板划线分离法从杭州七格污水处理厂二沉池的活性污泥中筛选得到一株对偶氮染料酸性橙7具有高效脱色功能的细菌, 编号为JL-1, 根据16S rRNA基因序列分析结果, 鉴定并命名为Aeromonas hydrophila strain JL-1.
2) 菌株JL-1可在缺氧振荡培养条件下对偶氮染料酸性橙7 (Acid Orange 7)进行脱色; 培养基、初始染料浓度、接种量、pH和NaCl浓度对菌株JL-1脱色性能及生长有明显影响; pH为5.0、盐度为10%、温度为30 ℃、转速为150 r·min-1的LB培养基(Luria-Bertani medium)为菌株JL-1最佳的脱色及生长环境, 在此环境条件下, 100 mg·L-1的偶氮染料AO7脱色率达95%以上.该菌株耐盐性良好, 能在高达30%盐度下对染料进行有效的脱色.
Amar T. 2008. Kinetics and mechanism of reactive Red 141 degradation by a bacterial isolate Rhizobium radiobacter MTCC 816[J]. Acta Chimica Slovenica, 55(2): 320–329.
|
Chen B Y. 2002. Understanding decolorization characteristics of reactive azo dyes by Pseudomonas luteula:toxicity and kinetics[J]. Process Biochemistry, 38: 437–446.
DOI:10.1016/S0032-9592(02)00151-6
|
Chen K C, Wu J Y, Liou D J, et al. 2003. Decolorization of the textile dyes by newly isolated bacterial strains[J]. Journal of Biotechnology, 101: 57–68.
DOI:10.1016/S0168-1656(02)00303-6
|
Dawkar V V, Jadhav U U, Ghodake G S, et al. 2009. Effect of inducers on the decolorization and biodegradation of textile azo dye Navy Blue 2 GL by Bacillus sp.VUS[J]. Biodegradation, 20: 777–787.
DOI:10.1007/s10532-009-9266-y
|
dos Santos A B, Cervantes F J, van Lier J B. 2007. Review paper on current technologies for decolourisation of textile wastewaters:perspectives for anaerobic biotechnology[J]. Bioresource Technology, 98: 2369–2385.
DOI:10.1016/j.biortech.2006.11.013
|
Fan L, Zhu S N, Liu D Q, et al. 2009. Decolorization of 1-amino-4-bromoanthraquinone-2-sulfonic acid by a newly isolated strain of Sphingomonas herbicidovorans[J]. International Biodeterioration & Biodegradation, 63: 88–92.
|
García-Martínez Y, Bengoa C, Stüber F, et al. 2015. Biodegradation of acid orange 7 in an anaerobic-aerobic sequential treatment system[J]. Chemical Engineering and Processing:Process Intensification, 94: 99–104.
DOI:10.1016/j.cep.2014.12.011
|
郭建博, 周集体, 王栋, 等. 2007. 降解偶氮染料耐盐菌GTY的分离鉴定及特性研究[J]. 环境科学学报, 2007, 27(2): 201–205.
|
Gou M, Qu Y Y, Zhou J, et al. 2009. Azo dye decolorization by a new fungal isolate, Penicillium sp.QQ and fungal-bacterial cocultures[J]. Journal of Hazardous Materials, 170: 314–319.
DOI:10.1016/j.jhazmat.2009.04.094
|
Jadhav U U, Dawkar V V, Ghodake G S, et al. 2008. Biodegradation of direct Red 5B, a textile dye by newly isolated Comamonas sp.UVS[J]. Journal of Hazardous Materials, 158: 507–516.
DOI:10.1016/j.jhazmat.2008.01.099
|
Kalyani D C, Patil P S, Jadhav J P, et al. 2008. Biodegradation of reactive textile dye Red BLI by an isolated bacterium Pseudomonas sp.SUK1[J]. Bioresource Technology, 99: 4635–4641.
DOI:10.1016/j.biortech.2007.06.058
|
Kalyani D C, Telke A A, Dhanve R S, et al. 2009. Ecofriendly biodegradation and detoxification of Reactive Red 2 textile dye by newly isolated Pseudomonas sp.SUK1[J]. Journal of Hazardous Materials, 163: 735–742.
DOI:10.1016/j.jhazmat.2008.07.020
|
Konsowa A H, Abd El-Rahman H B, Moustafa M A. 2011. Removal of azo dye acid orange 7 using aerobic membrane bioreactor[J]. Alexandria Engineering Journal, 50(1): 117–125.
DOI:10.1016/j.aej.2011.01.014
|
Lang W, Sirisansaneeyakul S, Ngiwsara L. 2013. Characterization of a new oxygen-insensitive azoreductase from Brevibacillus laterosporus TISTR1911:Toward dye decolorization using a packed-bed metal affinity reactor[J]. Bioresource Technology, 150: 298–306.
DOI:10.1016/j.biortech.2013.09.124
|
Lima R O A, Bazo A P, Salvadori D M, et al. 2007. Mutagenic and carcinogenic potential of a textile azo dye processing plant effluent that impacts a drinking water source[J]. Mutation Research, 626: 53–60.
DOI:10.1016/j.mrgentox.2006.08.002
|
Manu B, Chaudhari S. 2003. Decolorization of indigo and azo dyes in semicontinuous reactors with long hydraulic retention time[J]. Process Biochemistry, 38: 1213–1221.
DOI:10.1016/S0032-9592(02)00291-1
|
Muhammad I, Muhammad A, Negm F, et al. 2016. Yeast extract promotes decolorization of azo dyes by stimulating azoreductase activity in Shewanella sp.strain IFN4[J]. Ecotoxicology and Environmental Safety, 124: 42–49.
DOI:10.1016/j.ecoenv.2015.09.041
|
Nachiyar C V, Vijayalakshmi K, Muralidharan D, et al. 2007. Mineralization of metanilic acid by Pseudomonas aeruginosa CLRI BL22[J]. World Journal of Microbiology and Biotechnology, 23: 1733–1738.
DOI:10.1007/s11274-007-9422-7
|
Nam S, Renganathan V. 2000. Non-enzymatic reduction of azo dyes by NADH[J]. Chemosphere, 40(4): 351–357.
DOI:10.1016/S0045-6535(99)00226-X
|
Pandey A, Singh P, Iyengar L. 2007. Bacterial decolorization and degradation of azo dye[J]. International Biodeterioration & Biodegradation, 59: 73–84.
|
Qu Y Y, Shi S N, Ma F, et al. 2010. Decolorization of Reactive Dark Blue K-R by the synergism of fungus and bacterium using response surface methodology[J]. Bioresource Technology, 101: 8016–8023.
DOI:10.1016/j.biortech.2010.05.025
|
Saratale R G, Saratale G D, Chang J S, et al. 2009. Ecofriendly degradation of sulfonated diazo dye C.I.Reactive Green 19A using Micrococcus glutamicus NCIM-2168[J]. Bioresource Technolog, 100: 3897–3905.
DOI:10.1016/j.biortech.2009.03.051
|
Saratale R G, Saratale G D, Chang J S, et al. 2011. Bacterial decolorization and degradation of azo dyes:A review[J]. Institute of Chemical Engineers, 42: 138–157.
DOI:10.1016/j.jtice.2010.06.006
|
Supaka N, Juntongjin K, Damronglerd S, et al. 2004. Microbial decolorization of reactive azo dyes in a sequential anaerobic-aerobic system[J]. Chemical Engineering Journal, 99(2): 169–176.
DOI:10.1016/j.cej.2003.09.010
|
Tan L, Ning S X, Zhang X W, Shi S N. 2013. Aerobic decolorization and degradation of azo dyes by growing cells of a newly isolated yeast Candida tropicalis TL-F1[J]. Bioresource Technology, 138: 307–313.
DOI:10.1016/j.biortech.2013.03.183
|
Tan L, Li H, Ning S X, Xu B W. 2014. Aerobic decolorization and degradation of azo dyes by suspended growing cells and immobilized cells of a newly isolated yeast Magnusiomyces ingens LH-F1[J]. Bioresource Technology, 158: 321–328.
DOI:10.1016/j.biortech.2014.02.063
|
Tan L, He M Y, Li S, et al. 2016. Aerobic decolorization, degradation and detoxification of azo dyes by a newly isolated salt-tolerant yeast Scheffersomyces spartinae TLHS-SF1[J]. Bioresource Technology, 203: 287–294.
DOI:10.1016/j.biortech.2015.12.058
|
Tony B D, Goyal D, Khanna S. 2009. Decolorization of textile azo dyes by aerobic bacterial consortium[J]. International Biodeterioration & Biodegradation, 63: 462–469.
|
Tuttolomondo M V, Alvarez G S, Desimone M F, et al. 2014. Removal of azo dyes from water by sol-gel immobilized Pseudomonas sp[J]. Environ Chem Eng, 2: 131–136.
DOI:10.1016/j.jece.2013.12.003
|
Ulson de Souza S M A G, Forgiarini E, Ulson A A. 2007. Toxicity of textile dyes and their degradation by the enzyme horseradish peroxidase (HRP)[J]. Journal of Hazardous Materials, 147: 1073–1078.
DOI:10.1016/j.jhazmat.2007.06.003
|
Wang B E, Hu Y Y, Xie L, et al. 2008. Biosorption behavior of azo dye by inactive CMC immobilized Aspergillus fumigatus beads[J]. Bioresource Technol, 99: 794–800.
DOI:10.1016/j.biortech.2007.01.043
|
严滨, 周集体, 柳广飞, 等. 2008. 偶氮染料生物降解机理的研究[J]. 环境科技, 2008, 21(5): 1–5.
|
于文娟, 谢学辉, 洪武林, 等. 2012. 嗜水气单胞菌RB5-M1的分离鉴定及脱色条件研究[J]. 微生物学通报, 2012, 39(4): 458–467.
|
邹聪慧, 徐方成, 陈新华. 2011. 海洋产电菌Shewanella marisflavi EP1的脱色特性[J]. 微生物学通报, 2011, 38(1): 2–7.
|
 2018, Vol. 38
2018, Vol. 38


