2. 中国科学院生态环境研究中心, 北京 100085
2. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085
抗生素目前被广泛应用于人类和动物感染性疾病的控制, 并可作为亚治疗剂和促生长剂添加在饲料中(Chee-Sanford et al., 2009).中国是全球最大的抗生素生产和消费国(余忻, 2014), 抗生素的广泛使用导致其在土壤、地表水、地下水及饮用水等各种环境介质中的频繁检出(Qiao et al., 2018).环境中残留的抗生素不仅对环境造成污染, 还会诱导抗生素抗性细菌(Antibiotic Resistant Bacteria, ARB)和抗生素抗性基因(Antibiotic Resistance Genes, ARGs)的产生, ARGs可通过基因的水平转移进行传播, 从而对生态环境和人类健康造成威胁(Furuya et al., 2006; Wei et al., 2013).细菌的存在是ARGs传播的重要载体(Rysz et al., 2004).因此, 关注微生物在ARGs传播中发挥的作用具有重要意义.但是, 目前研究对于抗性基因与微生物, 尤其是与反映水体中微生物污染状况的指示微生物之间的相关性研究尚缺乏统一认识.
在我国环渤海区域, 以污水处理厂出水等非常规水源补给为主的河、湖逐渐成为城市群景观水体的主导类型;而随着“海绵城市”建设的全国推进, 以污水处理厂尾水为补给水源的城市湿地公园近年来亦大量涌现.城市景观水体在生态生境、城市景观、游憩活动等多种生态经济服务中发挥着重要的作用.圆明园作为再生水补给型城市景观水体的典型代表, 其用水均来自清河污水处理厂处理后的再生水(彭自然等, 2011), 日用水量达2×104~3×104 m3·d-1.大量文献研究指出, 污水处理厂现有的处理工艺并不能完全去除抗生素, 出水中残留的抗生素、ARB和ARGs可导致在下游环境中的传播(Rizzo et al., 2013; Wang et al., 2014).此外, 由于圆明园的水文系统处于封闭状态, 缺乏流动, 封闭的湖泊可能会为ARGs的积累提供适宜的传播条件.随着滞留时间的增加, 污染物的循环放缓, 其对生态系统和人类健康造成的生态风险很可能高于其他流动性强的自然水体.然而, 目前的研究多集中于城市河流、水库中ARGs的分布情况(Jiang et al., 2014; Qiu et al., 2019), 关于典型景观水体ARGs的分布状况同样值得关注.
因此, 本研究选取北京市典型景观水体圆明园, 通过荧光定量PCR技术, 分析圆明园内抗生素抗性基因的污染特征.本研究选取了2种大环内酯类(mefA和ermB)、2种四环素类(tetM和tetQ)和2种磺胺类(sulI和sulII)ARGs, 1种可转移元件intI1进行研究.并通过传统微生物学方法, 分析园区内3种指示微生物的浓度水平, 明确ARGs和指示微生物的相关性, 以期为城市景观水体ARGs的削减和相关风险评价提供基础科学研究支撑.
2 材料与方法(Materials and methods) 2.1 样品采集与处理圆明园地处温带属季风性气候, 夏季炎热多雨, 园区在2018年7月未有再生水进入, 无法考察再生水补水条件下园区内ARGs的丰度水平, 因此本研究选择有再生水补入的9月开展调查.此外, 圆明园内约有27个大小不同的湖泊, 水系情况复杂.随着城市发展和环境的恶化, 圆明园历史上的供水渠道现在都已断绝, 如今圆明园使用的仅为从园区北侧入水口进入园内的再生水.基于圆明园的水系流向, 我们从圆明园进水口开始采集样品, 采样点覆盖园区内最大的湖泊——福海, 重要景观水域——鉴碧亭、凤麟洲, 生态修复区域——方河, 采样点如图 1所示.其中, 采样点S1是园区进水口, S2和S3分别位于后海和福海, S4位于鉴碧亭, S5是凤麟洲, S6位于末端方河.所有水样均采用采水器在从距离水面0.5 m处采集样品, 并保存于无菌聚乙烯瓶中.采集后立即保存于4 ℃冰箱, 迅速送至实验室后在24 h内进行处理.水样的温度和pH用便携式仪器(YSI, 美国)在野外现场测定.水样带回实验室后, 用纳氏试剂比色法测定氨氮(NH3-N);过硫酸钾氧化紫外分光光度法测定总氮(TN);钼酸铵分光光度法测定水样中的总磷(TP);快速消解分光光度法测定化学需氧量(COD);用总有机碳分析仪(岛津TOC-L, 日本)测定水样中的总有机碳(TOC).
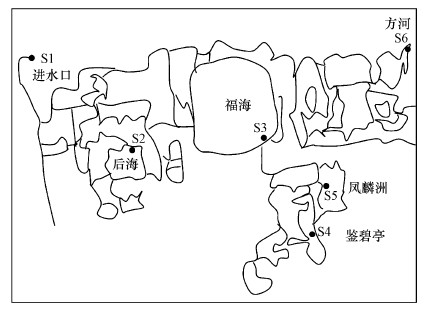 |
| 图 1 圆明园采样点空间位置示意 Fig. 1 Sketch of sampling sites in Yuanmingyuan |
目前, 国内外主要以指示菌的浓度水平来反映水体中微生物的污染状况.美国、世界卫生组织、欧盟采用的细菌指示微生物为大肠杆菌(Escherichia coli, 简称EC)、肠球菌(Enterococcus, 简称ENT), 而我国目前常采用的是粪大肠菌群(Fecal Coliform, 简称FC).因此, 本研究指示微生物以FC、EC和ENT作为监测指标, 培养方法参照我国行业标准HJ-T347—2007滤膜法、美国环保署(USEPA)标准EPA Method1603和EPA Method1600.
2.2 水样DNA提取将样品通过0.2 μm聚偏二氟乙烯膜过滤后, 用铝箔纸(避光)包裹置于-20 ℃冰箱中保存.提取DNA时, 用预先灭菌的剪刀将滤膜剪成片, 置于Lysing Matrix E tube离心管中后, 加入配套的978 μL PBS缓冲液.依据Fast DNA SPIN试剂盒(MP bio, 美国)中的方法提取总DNA.提取后的DNA通过紫外分光光度计检测其浓度和纯度.
2.3 荧光定量PCR使用StepOne Plus实时荧光定量PCR仪(ABI, 美国)对目标基因进行SYBR Green绝对定量PCR反应, 每个样品3个平行.所用引物见表 1.在无菌96孔板中配置20.0 μL的PCR反应体系:2× SYBR 10 μL;前引物、后引物各0.4 μL;DNA样品1 μL;ROX染料0.4 μL;无菌水7.8 μL.PCR反应条件为:95 ℃预变性时间30 s;95 ℃变性时间5 s, 60 ℃退火延伸30 s, 总共40个循环.程序质粒标准曲线的校准曲线呈现良好的线性关系(R2= 0.99).PCR扩增效率范围为95%~110%, 表明qPCR的扩增效率满足要求.
| 表 1 ARGs所用引物序列 Table 1 Primer sequences used for PCR amplification of ARGs |
采用StepOne Plus荧光定量PCR仪测定样品的16S rRNA基因, 采用标准质粒外标法对其丰度进行绝对定量.标准质粒原始浓度为2.81×1010 copies·L-1.将以10倍浓度梯度稀释的质粒用于制作绝对定量标准曲线, 标准曲线范围:2.81×104~2.81×109copies·L-1.PCR扩增的总反应体系为20 μL.反应体系中各试剂的最终浓度为:2×的SYBR Premix Ex TaqTM试剂;10 μmol·L-1的上下游引物;10 ng·μL-1的DNA模板;ROX染料.PCR反应条件为:95 ℃下预变性, 30 s;95 ℃变性, 5 s;退火时间30 s;72 ℃延伸时间30 s.依据仪器程序自动添加的熔解曲线进行分析.
2.5 数据分析基因的相对拷贝数由公式(1)计算, 绝对丰度由公式(2)计算(Looft et al., 2012; 张丹丹等, 2018).
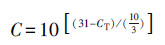
|
(1) |
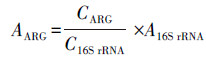
|
(2) |
式中, C为基因的相对拷贝数, CT为PCR反应收集到特定荧光时的循环次数, CARG为样本中ARGs的相对拷贝数, C16S rRNA为样本中16S rRNA基因的相对拷贝数, 样本中ARGs的相对丰度即为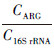
利用Microsoft Excel 2010进行计算分析, Origin Pro9.0制图, 采用SPSS 22.0中的配对T检验和相关性分析进行数据相关性的分析.当p值小于0.05时, 均认为存在显著相关性.使用Canoco 5进行ARGs和环境因子的相关性分析.
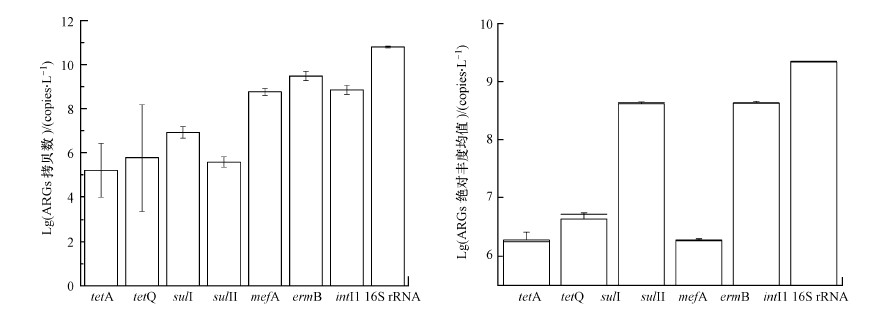
3 结果与讨论(Results and discussion) 3.1 圆明园水体中不同种类ARGs分布特征本研究所测的大环内酯类、四环素类和磺胺类ARGs在圆明园水体中均有检出, 说明圆明园水环境已受到ARGs污染(图 2a).圆明园中ARGs的绝对丰度在n.d.(未检出)~1.08×109 copies·L-1间, 其中, sulII绝对丰度为3.15×105~1.08×109 copies·L-1, sulII在检测ARGs中占据主导地位, 即ARGs的绝对丰度依次为:sulII>sulI> mefA> tetQ> tetM>ermB(图 2b).这一结果与上海城市河网(朱春红, 2018)、北京温榆河(Liu et al., 2019)中ARGs的检出结果一致;区别于海河流域(杨继平等, 2017), 海河流域的结果显示sulI的丰度高于其余ARGs.sulII为圆明园优势ARGs, 推测一方面是与sulI和sulII位于不同的移动元件上有关:sulI位于整合子上, 而sulII位于宿主广泛的质粒上;两种磺胺类ARGs均编码于革兰氏阴性肠道菌中对磺胺类耐药的二氢叶酸合成酶(DHPS)上, 但是, sulII的DHPS似乎在区分底物方面非常敏锐, 使得sulII的分布更为广泛(Su et al., 2012; Zhang et al., 2016).另一方面, 刘璐等(2018)研究发现, 高浓度的氮磷对sulI基因有显著的抑制作用, 由于9月园区部分点位氮磷浓度达劣五类水平, 由此可能也抑制了园内sulI基因的浓度水平.
 |
| 图 2 圆明园内ARGs的丰度分布(a)及ARGs的绝对丰度(b) Fig. 2 Distribution of ARGs in Yuanmingyuan(a) and absolute abundance of ARGs(b) |
园区内大环内酯类和四环素类ARGs较磺胺类相比低1~2个数量级, 这可能与本地抗生素的使用类型、残留的环境选择压力相关.通常认为, 环境中存在的抗生素对ARGs的诱导、产生和维持都存在影响(沈群辉等, 2012; 马业萍, 2015).Zhang等(2015)的研究指出, 中国磺胺类、四环素类和大环内酯类抗生素的消耗量占总使用量的38%.而人类和动物消耗的抗生素很大一部分随排泄物进入到生活污水中(Sarmah et al., 2006).圆明园的景观用水均来自清河污水处理厂处理后的再生水, 清河污水处理厂也是北京市污水处理厂中规模较大的, 主要收集清河流域排放的生活污水.而现有的污水处理工艺对抗生素的去除效果有限, 大量未被去除的抗生素经城市污水厂进入水环境中(徐建等, 2018).因此, 圆明园内磺胺类ARGs的检出, 也表明该区域磺胺类抗生素的广泛使用.而ARGs的检出除了与该地抗生素的用药习惯有关外, 也可能是由于可移动遗传元件促进的ARGs的传播和扩散.本研究也证实了sulI与intI1间显著相关性(p < 0.05), 这与先前Luo等(2010)的研究结果相似, 表明sulI受intI1的促进传播, 其主要原因是sulI基因序列位于intI1基因盒序列中(Nigro et al., 2013).
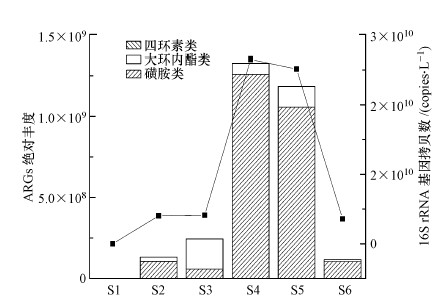
3.2 圆明园水体中ARGs的空间分布特征图 3为各采样点ARGs的变化情况.结果显示, 不同点位ARGs的丰度存在差异.ARGs的浓度水平除受抗生素使用量的影响外, 还与细菌生物量、水生植物等多种因素有关.由于细菌的存在是ARGs传播的重要载体, Wu(2010)的研究指出, ARGs的拷贝数与16S rRNA的基因拷贝数显著相关, 因此推测从园区进水口至末端ARGs出现升高后降低可能是由于细菌含量变化引起的.有研究表明, 人工湿地中ARGs去除差异可能和水生植物的种类有关(Chen, 2013; Fang et al., 2017), 即种植的水生植物可能对水体中ARGs的去除有一定的效果.采样点S1位于园区进水口附近, 为净化水源, 附近种植有水生植物, 而S4(鉴碧亭)和S5(凤麟洲)处种植的黄花鸢尾、千屈菜等水生植物受人为破坏严重, 使得园内ARGs污染主要在鉴碧亭和凤麟洲区域.此外, 园区进水口目标ARGs的丰度低于末端, 表明封闭的水体可能会为ARGs的积累提供适宜环境.
 |
| 图 3 各采样点ARGs绝对丰度的变化 Fig. 3 The variation of absolute abundance of ARGs in each sample |
圆明园水体环境因子检测结果如表 2所示.由表 2可知, 同一时间内各采样点水温变化不大, 所有采样点的pH均在7~9之间.园区COD为14.68~60.12 mg·L-1, 根据《地表水环境标准》(GB3838—2002), 9月圆明园COD均值超Ⅴ类水质标准, COD在S5处达到最大值, 可能是由于人们在园区游览中, 存在向水体中投入饵料的行为, 使得水体受到有机污染, COD值升高.此外, 进水口处TN污染严重, 而在园区末端TN值有明显下降, 这可能与园区内植物的吸收利用有关.磷作为水体富营养化的关键因素, 9月园区除S3外TP浓度都较低, S3处TP含量升高的原因可能与福海区域底泥磷的释放有关.
| 表 2 圆明园水质参数统计特征 Table 2 Summary statistics of measured parameters in Yuanmingyuan |
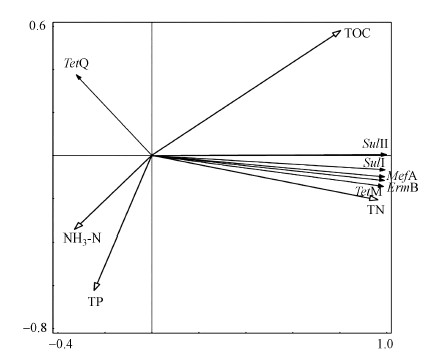
环境中ARGs的存在不仅与抗生素有关, 环境因子同样会影响ARGs的水平.因此, 本研究将各环境因子和目标ARGs的浓度进行冗余分析(Redundancy analysis, RDA), 结果如图 4所示.TN与目标ARGs(除tetQ外)间的夹角较小, 呈现出显著相关性(p < 0.01), 表明氮的存在对ARGs在环境中的迁移和转化有显著的影响.Liaqat等(2016)研究指出, 氮通过为微生物提供能量进而影响环境中微生物含量, 从而导致ARGs变化.在本研究中, 尽管TOC、TP和目标ARGs并未表现出显著相关性, 但Demaneche等(2001)的研究指出, TOC与ARGs的产生和传播有关, 并且可保护其免受降解;而磷等无机盐和细胞结构组成、能量转移等功能有关, 因此ARGs的变化与氮磷等营养元素、有机物等存在一定的相关性.综上, 由于环境因子可极大地影响微生物群落组成, 因此它们将会引起城市水体环境中ARGs的变化.
 |
| 图 4 ARGs与环境因子的RDA分析 Fig. 4 Redundancy analysis(RDA) of ARGs and environmental factors |
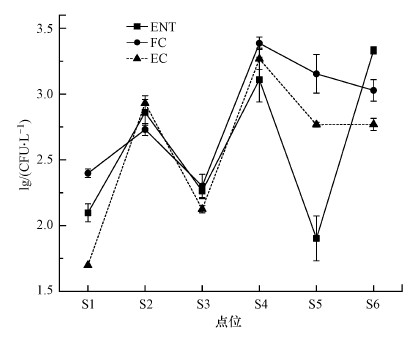
各采样点3种指示微生物的平均浓度如图 5所示.由图可知, 不同点位的微生物赋存状况不同.S1、S3、S4、S5的FC浓度最高, 为50~1.85×103 CFU·L-1;而采样点S2的EC浓度最高, 为8.5×102 CFU·L-1;园区末端S6的ENT污染严重, 浓度为2.15×103 CFU·L-1.根据我国《地表水环境质量标准》(GB 3838—2002)的规定, Ⅲ类水FC浓度限值为10000 CFU·L-1(国家环保总局, 2002).因此, 圆明园内FC浓度在Ⅲ类水质标准允许范围内.此外, 3种指示菌的变化趋势存在一致性.以区段为尺度分析空间差异性显示, 园区末端方河指示菌污染状况显著高于进水口区段.这一结果的得出是由于微生物在末端的蓄积作用导致的(Brookes et al., 2004; Wang et al., 2017).
 |
| 图 5 圆明园指示微生物浓度分布图 Fig. 5 Concentration of indicator microorganisms in Yuanmingyuan |
研究发现6种ARGs中, sulI和sulII与粪大肠菌群和大肠杆菌具有显著相关性(表 3), 其余ARGs与3种指示菌均未见相关性.此结果的出现可能是由于ARGs可借助可移动遗传元件(如质粒、转座子、整合子以及插入序列等)在不同的微生物个体间进行基因水平转移(Ma et al., 2017).例如, tetE基因被发现存在于渔业养殖环境菌Aeromonas. spp的可转移性质粒上, 已被证明可以转移到Escherichia coli上(Agersø et al., 2007);ermB基因可以通过质粒和转座子从其主要宿主菌葡萄球菌、链球菌和肠球菌向其他菌属转移(张卓然等, 2007).但已有研究表明, 经排泄进入环境的肠道菌可能会与土著菌发生ARGs的水平转移, 进而使得抗性在环境中传播(高盼盼等, 2011; 张婉茹等, 2014).因此, 关注肠道菌群和抗性基因之间相关性的研究仍然十分必要.
| 表 3 ARGs丰度与指示微生物间的相关性分析 Table 3 Correlation analysis between ARGs and indicator microorganisms |
1) 在检测的目标ARGs中, 磺胺类ARGs在圆明园中呈现最高的绝对丰度.
2) 园区进水口目标ARGs的丰度低于末端, 表明封闭的水体可能为ARGs的积累提供了适宜环境.
3) 环境因子中的总氮可显著影响ARGs在城市景观水体中的传播;sulI和sulII与粪大肠菌群和大肠杆菌存在显著相关性, 说明指示菌在ARGs的空间分布及扩散的过程中可能发挥着至关重要的作用.
Agersø Y, Bruun M S, Dalsgaard I, et al. 2007. The tetracycline resistance gene tet(E) is frequently occurring and present on large horizontally transferable plasmids in Aeromonas spp. from fish farms[J]. Aquaculture, 266(1): 47-52. |
Aminov R I, Garrigues-Jeanjean N, Mackie R I. 2001. Molecular ecology of tetracycline resistance: Development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied and Environmental Microbiology, 67(1): 22-23. DOI:10.1128/AEM.67.1.22-32.2001 |
Aminov R I, Mackie R I, Koike S, et al. 2007. Monitoring and source tracking of tetracycline resistance genes in lagoons and groundwater adjacent to swine production facilities over a 3-year period monitoring and source tracking of tetracycline resistance genes in lagoons and groundwater adjacent to swine production facilities over a 3-year period[J]. Applied & Environmental Microbiology, 73(15): 4813. |
Brookes J D, Antenucci J, Hipsey M, et al. 2004. Fate and transport of pathogens in lakes and reservoirs[J]. Environment International, 30(5): 741-759. DOI:10.1016/j.envint.2003.11.006 |
Chee-Sanford J C, Mackie R I, Koike S, et al. 2009. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
Chen H, Zhang M. 2013. Effects of advanced treatment systems on the removal of antibiotic resistance genes in wastewater treatment plants from Hangzhou, China[J]. Environmental Science and Technology, 47: 8157-8163. |
Chen J, Yu Z, Michel F C, et al. 2007. Development and application of real-time PCR assays for quantification of erm genes conferring resistance to macrolides-lincosamides-streptogramin B in livestock manure and manure management systems[J]. Applied and Environmental Microbiology, 73(14): 4407-4416. DOI:10.1128/AEM.02799-06 |
Demaneche S, Jocteur-Monrozier L, Quiquampoix H, et al. 2001. Evaluation of Biological and Physical Protection against Nuclease Degradation of Clay-Bound Plasmid DNA[J]. Applied and Environmental Microbiology, 67(1): 293-299. DOI:10.1128/AEM.67.1.293-299.2001 |
Fang H, Zhang Q, Nie X, et al. 2017. Occurrence and elimination of antibiotic resistance genes in a long-term operation integrated surface flow constructed wetland[J]. Chemosphere, 173: 99-106. DOI:10.1016/j.chemosphere.2017.01.027 |
Furuya E Y, Lowy F D. 2006. Antimicrobial-resistant bacteria in the community setting[J]. Nature Reviews Microbiology, 4(1): 36. DOI:10.1038/nrmicro1325 |
高盼盼, 罗义, 毛大庆. 2011. 天津水产业磺胺类耐药细菌及其分布[J]. 生态毒理学报, 6(1): 74-79. |
国家环保总局, 国家质量监督检验检疫总局. 2002.地表水环境质量标准(GB3838-2002) [S].北京: 中国标准出版社
|
Jiang Y, Li M, Guo C, et al. 2014. Distribution and ecological risk of antibiotics in a typical effluent-receiving river (Wangyang River) in north China[J]. Chemosphere, 112: 267-274. DOI:10.1016/j.chemosphere.2014.04.075 |
Liaqat A, Chen J, Wang Y, et al. 2016. Nutrient-induced antibiotic resistance in Enterococcus faecalis in the eutrophic environment[J]. Journal of Global Antimicrobial Resistance, 7: 78-83. DOI:10.1016/j.jgar.2016.07.014 |
刘璐, 刘萱, 王华, 等. 2018. 水环境因子对磺胺类抗性基因Sul1含量的影响[J]. 农业环境科学学报, 37(3): 515-519. |
Liu X, Zhang G, Liu Y, et al. 2019. Occurrence and fate of antibiotics and antibiotic resistance genes in typical urban water of Beijing, China[J]. Environmental Pollution, 246: 163-173. DOI:10.1016/j.envpol.2018.12.005 |
Looft T, Johnson T A, Allen H K E A. 2012. Infeed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 109(5): 1691-1696. DOI:10.1073/pnas.1120238109 |
Luo Y, Mao D, Rysz M, et al. 2010. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science and Technology, 44(19): 7220-7225. DOI:10.1021/es100233w |
Luo Y, Mao D, Rysz M, et al. 2010. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environ Sci Technol, 44: 7220-7225. DOI:10.1021/es100233w |
Ma L, Li A, Yin X, et al. 2017. The prevalence of integrons as the carrier of antibiotic resistance genes in natural and man-made environments[J]. Environmental Science & Technology, 51(10): 5721-5728. |
Mulvey M, Martin M I, Alfa M, et al. 2001. Multiplex PCR for the detection of tetracycline resistant genes[J]. Molecular and Cellular Probes, 15(4): 209-215. DOI:10.1006/mcpr.2001.0363 |
Nigro P, Pompilio G, Capogrossi M C. 2013. Cyclophilin A:a key player for human disease[J]. Cell Death &Disease, 4(10): e888. |
彭自然, 何文辉, 高佳慧, 等. 2011. 圆明园景观水体生态修复过程中水质变化的初步研究[J]. 上海海洋大学学报, 20(03): 457-461. |
Qiao M, Ying G, Singer A C, et al. 2018. Review of antibiotic resistance in China and its environment[J]. Environment International, 110: 160-172. DOI:10.1016/j.envint.2017.10.016 |
Qiu W, Sun J, Fang M, et al. 2019. Occurrence of antibiotics in the main rivers of Shenzhen, China: Association with antibiotic resistance genes and microbial community[J]. Science of the Total Environment, 653: 334-341. DOI:10.1016/j.scitotenv.2018.10.398 |
Rizzo L, Manaia C, Merlin C, et al. 2013. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: A review[J]. Science of the Total Environment, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
Rysz M, Alvarez P J J. 2004. Amplification and attenuation of tetracycline resistance in soil bacteria: aquifer column experiments[J]. Water Research, 38(17): 3705-3712. DOI:10.1016/j.watres.2004.06.015 |
Sarmah A K, Meyer M T, Boxall A B A. 2006. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics (VAs) in the environment[J]. Chemosphere, 65(5): 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
沈群辉, 冀秀玲, 傅淑珺, 等. 2012. 黄浦江水域抗生素及抗性基因污染初步研究[J]. 生态环境学报, 21(10): 1717-1723. DOI:10.3969/j.issn.1674-5906.2012.10.015 |
Su H, Ying G, Tao R, et al. 2012. Class 1 and 2 integrons, sul resistance genes and antibiotic resistance in Escherichia coli isolated from Dongjiang River, South China[J]. Environmental Pollution, 169: 42-49. DOI:10.1016/j.envpol.2012.05.007 |
Sutcliffe J, Grebe T, Tait-Kamradt A, et al. 1996. Detection of erythromycin-resistant determinants by PCR[J]. Antimicrobial Agents and Chemotherapy, 40(11): 2562-2566. DOI:10.1128/AAC.40.11.2562 |
Wang F, Qiao M, Lv Z, et al. 2014. Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China[J]. Environmental Pollution, 184: 247-253. DOI:10.1016/j.envpol.2013.08.038 |
Wang Y, Chen Y, Zheng X, et al. 2017. Spatio-temporal distribution of fecal indicators in three rivers of the Haihe River Basin, China[J]. Environmental Science and Pollution Research, 24(10): 9036-9047. DOI:10.1007/s11356-015-5907-3 |
Wei Q, Jiang X, Li M, et al. 2013. Diversity of Gene Cassette Promoter Variants of Class 1 Integrons in Uropathogenic Escherichia coli[J]. Current Microbiology, 67(5): 543-549. DOI:10.1007/s00284-013-0399-1 |
Wu Nan, Qiao M, Zhang B, et al. 2010. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China[J]. Environmental Science and Technology, 44(18): 6933-6939. DOI:10.1021/es1007802 |
徐建, 胡鹏, 吕佳佩, 等. 2018. 水环境中抗生素和抗性基因污染特征及控制措施[J]. 科技导报, 36(15): 13-23. |
杨继平, 邱志刚, 袁兆康, 等. 2017. 天津海河流域抗生素抗性基因分布特征及与指示微生物的关系[J]. 环境与健康杂志, 34(4): 313-316. |
余忻. 2014.抗生素废水的生物毒性与微生物耐药性及其控制技术研究[D].北京: 清华大学 http://cdmd.cnki.com.cn/Article/CDMD-10003-1015039172.htm
|
张丹丹, 郭亚平, 任红云, 等. 2018. 福建省敖江下游抗生素抗性基因分布特征[J]. 环境科学, 39(6): 2600-2606. |
Zhang Q, Ying G, Pan C, et al. 2015. A comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modelling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 49(11): 6772-6782. |
Zhang X H, Xu Y B, He X L, et al. 2016. Occurrence of antibiotic resistance genes in landfill leachate treatment plant and its effluent-receiving soil and surface water[J]. Environ Pollut, 218: 1255-1261. DOI:10.1016/j.envpol.2016.08.081 |
张婉茹, 那广水, 陆紫皓, 等. 2014. 北黄海近岸海域磺胺类抗生素及其抗性Escherichia coli分布[J]. 应用与环境生物学报, 20(3): 401. |
张卓然, 夏梦岩, 倪语星. 2007. 微生物耐药的基础与临床[M]. 北京: 人民卫生出版社.
|
朱春红. 2018.上海市城市河网抗生素、抗性基因时空分布特征及其与环境因子相关性研究[D].上海: 华东师范大学
|
 2019, Vol. 39
2019, Vol. 39


