2. 山东省土地生态修复工程技术研究中心, 滨州 256600;
3. 浙江财经大学中国政府管制研究院, 杭州 310018
2. Land Ecological Restoration Engineering Technology Research Center of Shandong Province, Binzhou 256600;
3. China Institute of Regulation Research, Zhejiang University of Finance & Economics, Hangzhou 310018
盐碱土是盐土和碱土及各种盐化和碱化土的总称, 主要存在于全球的干旱和半干旱地区, 至少影响了世界范围内75个国家的土壤健康和农业发展.据联合国教科文组织(UNESCO)和粮农组织(FAO)不完全统计, 全世界盐碱地面积为9.54亿hm2(Qadir et al., 2008).盐碱土在中国广泛分布于西北、华北、东北及东部沿海地区, 总面积达3600万hm2, 占全国可利用土地面积的4.88%(杨劲松, 2008).土壤含盐量急增会改变土壤理化和生物学性质, 降低土壤质量, 从而打破原有植被群落结构, 进一步影响土壤理化和生物学性质, 造成恶性循环, 导致土壤碳库急剧下降, 对气候和环境产生不利影响(孔涛等, 2014).
土壤微生物群落结构受到土壤理化性质、植物类型、环境污染等因素的影响(Ramirez et al., 2012; Bell et al., 2015).盐碱地中不同的土地利用方式会导致各种土壤微生物与植物在长期的共生环境中形成独特的耐盐机制, 以应对盐碱对自身的不利影响(Lúcio et al., 2013).研究表明, 盐碱环境条件下的土壤含盐量和pH比非盐碱土高, 土壤盐分对土壤微生物群落的活性、多样性和结构存在显著的影响(Chandrasekaran et al., 2014).目前国际上已有较多对林地、草地、湿地、耕地等不同土地利用方式下的土壤微生物种群多样性的研究(Ma et al., 2018;白雪娟等, 2018;王颖等, 2018;包明等, 2018), 主要是从化肥投入、种植方式、经营管理模式及土壤环境条件等方面对土壤微生物的影响进行相关性分析(Wang et al., 2015;夏昕等, 2015;张彦军, 2018;刘开辉等, 2018;李倩等, 2018), 并取得了较为丰富的成果.但针对盐碱地不同土地利用方式下土壤微生物群落组成之间的特征差异及其影响因子分析等的相关研究较少.
黄河三角洲是我国乃至世界上造陆速度最快的河口三角洲之一, 不断的造陆提供了丰富的土地资源, 但土壤盐碱化也严重限制了该区域农业的发展(Ottinger et al., 2013).因此, 本文选择黄河三角洲进行盐碱地不同土地利用方式下土壤细菌群落多样性差异分析, 以填补该区域盐碱地土壤细菌研究工作的缺失, 并为该区域乃至华北平原盐碱地土地利用方式的优化提供参考, 最终促进该区域农业发展.
2 材料和方法(Materials and methods) 2.1 研究区概况研究区位于山东省滨州市潘家村, 靠近山东省最北端, 地处沿海, 地理坐标为东经117°64′、北纬37°76′, 属于黄河三角洲综合开发的重点区域, 以小麦等粮食作物和冬枣等优质水果闻名.该地区属北温带东亚季风区域大陆性气候, 夏热多雨, 冬寒季长, 全村土壤盐分高、碱性强, 0~60 cm土壤的平均电导率为230.89 μS·cm-1, 平均pH值为8.69, 平均含水量为6.57%.
2.2 样品采集选择研究区内不同土地利用方式的盐碱地进行研究.于2018年4月23日上午8:00—10:00进行土壤采集, 采样时当地平均气温为22 ℃.选取不同土地利用方式的盐碱地(耕地、园地、荒地), 为了最大限度地控制外因对土壤的影响, 本研究选取大小为10 m×10 m且远离道路的地块进行采样.每种地类挑选5个试验点, 采集不同深度(0~15、15~30、30~60 cm)土壤剖面样品, 每个试验点随机选取5个剖面采样, 然后将相同深度的样品混合均匀.现场挑选出杂质(石子、碎草等), 将20 g左右的样品用锡纸包装, 然后用聚乙烯塑料袋分装, 并在袋上做好标记, 放进干冰保温箱中, 运回实验室后保存于-80 ℃的冰箱.用于土壤理化性质测定的土样, 通过风干过筛后, 于干燥环境中常温保存.
2.3 样品处理 2.3.1 土壤理化性质测定土壤水分运用电热恒温烘箱于105 ℃条件下烘烤6 h后进行平行测定;土壤pH值通过pH计直接读取数据;土壤电导率通过电导率仪运用电极法测定;土壤有机质含量通过总碳分析仪运用燃烧氧化-非分散红外法测定;土壤有效磷运用紫外/可见光分光光度计进行比色测定;土壤速效钾运用火焰光度计法测定.
2.3.2 DNA提取与基因组DNA测序取5 g采集的土样于加入液氮的研钵中研磨, 按照Power Soil DNA Kit(MoBio, USA)试剂盒的说明, 提取土壤微生物基因组DNA, 置于-20 ℃的冰箱中保存.
对16S rRNA基因的V4、V5区域进行扩增, 引物序列为515F(5′-GTGCCAGCMGCCGCGG-3′)、907R(5′-CCGTCAATTCMTTTRAGTTT-3′).PCR反应体系(62 μL)如下:DNA模板4 μL, 2×Taq Master Mix 31 μL, 10 μmol·L-1正反向引物各1 μL, 加ddH2O至62 μL.扩增条件为:94 ℃预变性5 min;94 ℃变性30 s, 55 ℃复性30 s, 72 ℃延伸30 s, 28个循环;最后72 ℃彻底延伸5 min.利用Agarose Gel DNA Fragment Recovery Kit试剂盒(TaKa Ra)切胶纯化所获得的扩增产物, 经1%琼脂糖凝胶电泳鉴定, 割胶回收后, 通过Ion Torrent Personal Genome Machine(PGM)测序仪进行建库测序.
2.4 数据分析通过Ion Torrent PGM测序平台, 在97%相似度下使用Mothur软件包获得物种操作分类单元(OTU).采用RDP classifier 2.12对OTUs进行分类学分析, 得到每个OTU对应的物种分类信息, 并在界、门、纲、目、科、属水平上统计各个样品的细菌群落组成.利用QIIME1.8软件进行样品α多样性分析, 计算Chao1、Shannon、Simpson等物种多样性指数.通过HemI 1.0软件制作细菌群落热图用于分析物种相对丰度.通过基于距离矩阵算法的主坐标分析(PCoA)展示细菌的β多样性, 从而比较不同样品之间的菌群组成的差异.采用相关分析找出影响细菌物种群落结构的主要环境因子.
3 结果与分析(Results and analysis) 3.1 土壤理化性质分析土壤样品理化性质测定结果见表 1.耕地和园地的土壤pH值均低于荒地土壤, 这主要是耕地和园地受到人为灌溉的结果, 导致酸碱度比荒地要低一些(Lüneberg et al., 2018).浅表层土壤中, 耕地和园地的土壤电导率和含水量明显高于荒地土壤, 但随着土壤深度的增加, 荒地土壤电导率和含水量增长较快.有效磷、速效钾及有机质含量在耕地和园地土壤中随深度变化而呈现出较大的波动幅度, 这主要是化肥施用的结果(张双双等, 2019).
| 表 1 不同土地利用方式下土壤细菌菌群丰度、多样性指标及土壤理化性质 Table 1 Bacterial flora abundance and diversity indexes and physical and chemical properties of soil under different land use patterns |
细菌群落结构丰富度和多样性主要是由代表OTUs统计的Observed spieces、表征菌群丰度的Chao1指数及反映菌群多样性的Shannon和Simpson指数来表示.研究区3种土地利用方式下不同深度的土壤共9个样品, 其物种数都超过了5000, Shannon多样性指数均大于11, 表明该研究区内盐碱地土壤细菌群落结构较为稳定.从耕地、园地和荒地3种土地利用方式下的土壤细菌物种数和Shannon多样性指数上看, 在深度为15~30 cm和30~60 cm的条件下, 荒地土壤细菌物种数和Shannon多样性指数明显高于耕地和园地, 说明荒地土壤细菌群落丰富度和多样性更优于耕地和园地土壤细菌群落, 这与已有研究(刘旻霞等, 2018)的结果一致.通过比较不同深度相同土地利用方式下的土壤物种数和Shannon多样性指数, 发现耕地和园地的土壤物种数和Shannon多样性指数都随着土壤深度的增加呈下降趋势, 而荒地则与之相反.这主要是由于耕地和园地受到人为的经营管护, 表层土的有机质含量较高, pH值较低, 更适合微生物的生存和繁衍(Xu et al., 2017; Lu et al., 2018).通过比较同一深度不同土地利用方式下的土壤物种数和Shannon多样性指数, 发现园地浅表层的土壤物种数和Shannon多样性指数数值最高, 但随着土壤深度的增加, 荒地的这两项指标数值逐渐超过耕地和园地.通过细菌群落结构heatmap图也可以直观地看出, 荒地中土壤细菌在门类上的物种相对丰度略高于耕地和园地(图 1).
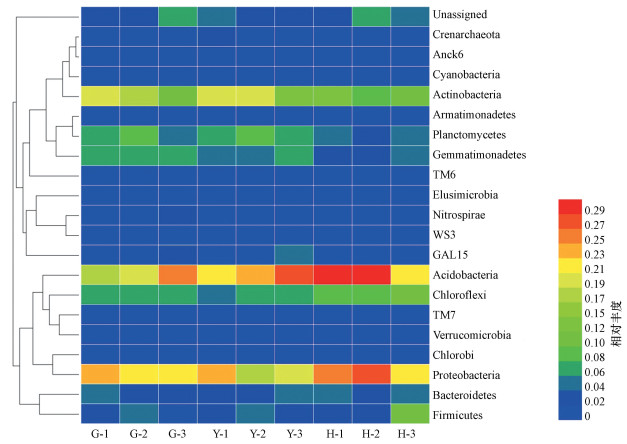 |
| 图 1 土壤细菌群落相对丰度聚类分析 Fig. 1 Cluster analysis of relative abundance of soil bacterial communities |
PCoA分析, 即通过各土壤细菌样品序列间的进化信息来测算样品间距离, 从而将不同样品间的细菌群落进行区分以观察群体之间的相似性与差异性.图 2中形状相同的点代表同一分组的样品, 两个点之间的距离越近, 说明它们之间的细菌群落相似性越高, 反之则差异性越高.本次检测的样本受主成分PC1、PC2、PC3的影响最大, 贡献率越大则说明解释样品细菌群落结构的能力就越强.其中, PC1可以解释样品中所有差异的32.41%, 代表最能区分所有样品的主坐标轴;PC2可以解释样品中所有差异的20.51%, 代表最能区分所有样品的第二主坐标轴;PC3可以解释样品中所有差异的27.23%.由图 2可知, 代表耕地和园地不同深度土样的点之间的距离都比较近, 而代表荒地土样的点却离的比较远.由此推断得出, 种植人工作物的土壤中细菌群落结构相似度较高, 与受人为干扰较少的荒地土壤细菌种群之间存在较大的差异, 这与已有的研究结果一致(Gomez et al., 2007; 王影等, 2013).
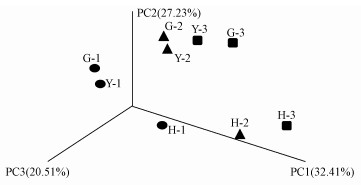 |
| 图 2 PCoA分析图 Fig. 2 PCoA analysis diagram |
各个样品在门分类水平上具有较高的多样性(图 3a), 达到20个门以上, 主要包括酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)、浮霉菌门(Planctomycetes)、变形菌门(Proteobacteria)等, 其中以放线菌门、变形菌门、酸杆菌门、绿弯菌门及浮霉菌门的细菌为主, 约占总测序序列的69%~83%.优势菌门类中, 放线菌门和变形菌门在各样品中均占较大的比例(18%~31%), 这符合以往对盐碱地微生物的研究结论(Chatterjee et al., 2017; Zhang et al., 2018).其中, 土壤放线菌对一些有害真菌表现出明显的抑制效果, 具有广谱抗生性及促进植株种子萌发、胚根生长的作用(刘灵芝等, 2013).而变形菌门下的根瘤菌能与许多植物之间组成共生固氮系统, 从而提高土壤肥力、改善土壤物质循环和增加土壤微生物活性, 进而提高土壤质量(Mitchell et al., 2009).酸杆菌门、绿弯菌门和浮霉菌门细菌在样品中的含量也较高, 分别占总测序序列的10%~21%、5%~11%和3%~10%, 它们都是土壤中比较重要的细菌种群, 其普遍性及相对丰度也表明它们在土壤生物化学循环系统中所起的关键作用(万盼等, 2018).
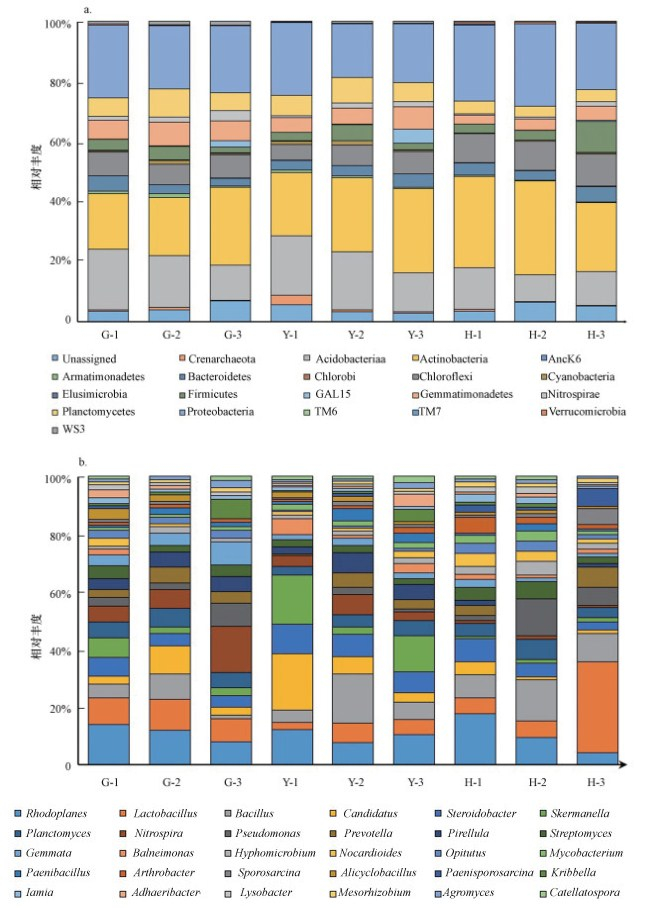 |
| 图 3 门水平(a)和属水平(b)上土壤细菌菌群组成结构 Fig. 3 Composition of soil bacterial flora at phylum level (a) and genus level (b) |
在属水平上的细菌群落达230多属, 图 3b中展示了主要菌属的结构图, 优势菌属主要来自酸杆菌门、变形菌门、厚壁菌门、浮霉菌门、芽单胞菌门及绿弯菌门.酸杆菌属在土壤中的相对丰度最高, 为5.76%, 在3种地类中, 荒地土壤的酸杆菌属相对丰度最低.因为酸杆菌属具有良好的分解凋落物能力, 在园地和耕地等具有丰富植株的土壤中更容易得到滋养, 从而有效推动土壤碳循环系统运作.此外, 变形菌门下的红游动菌属也具有较高的相对丰度, 该菌属是一种具有脱氮作用的含氮化合物功能菌, 对提升土壤有机质含量有较大促进作用(陈重军等, 2016;梁志婷等, 2017).厚壁菌门下的乳酸菌属也具有较高的相对丰度, 在耕地不同深度的土壤中大量存活, 该菌属能产生大量有机酸, 并不断释放出土壤中的迟效态氮、磷、钾, 从而有效提高土壤有机质含量, 改善盐碱地土壤的理化和生物性状(高鹏飞等, 2014).
4 讨论(Discussion) 4.1 土壤理化性质相关性土壤pH值是土壤细菌群落多样性的重要影响因素, 对土壤有效磷、速效钾及有机质的合成和分解具有重要作用(Ndour et al., 2008).通过相关性分析(表 2)可见, pH值与土壤有机质之间显著负相关, 随着pH值的升高, 有机质含量将会明显下降.这是因为有机物分解为有机质的最适宜pH值接近中性, 随着pH值的升高, 微生物或是物理化学转化有机质的活动将会受到限制.
| 表 2 土壤理化性质指标之间的相关性 Table 2 Correlation between soil physical and chemical properties |
3种土地利用方式下的土壤含水量都较低, 这是因为土样采集的时间处于春季, 期间干旱多风, 造成土壤水分含量总体较低.但仍然可以看出园地的土壤含水量略高于耕地和荒地, 这是由于园地受到灌溉并且枣树的根系具有较好的蓄水能力(缪凌等, 2016).土壤电导率可以直接测算出土壤中所含的总盐量, 是表征土壤盐碱化程度的主要指标(刘旻霞等, 2018).通过相关分析(表 2)可见, 土壤电导率与含水量之间显著正相关, 这是由于深层土壤积累了灌溉或雨水冲洗下来的盐分, 使得水分和盐分在同一深度的变化情况极为相似.
有效磷、速效钾及有机质是表征土壤养分的主要指标.通过相关分析(表 2)发现, 有效磷与速效钾、有机质之间显著正相关.这主要是耕地与园地受施肥作用的影响, 土壤养分随着深度的增加呈下降趋势, 而荒地由于未受到施肥影响, 其三者的含量也随深度变化呈现相似的变化情况, 这与已有文献(冯晨等, 2018;刘兴华等, 2018)的研究结果一致.
4.2 土壤理化性质与土壤细菌群落结构多样性通过对不同土地利用方式下的土壤细菌多样性与土壤理化因子之间相互关系的分析发现(表 3), 含水量与Shannon指数呈显著负相关;有效磷与Chao1指数、observed_species、Shannon指数及Simpson指数都呈显著正相关;速效钾与Chao1指数、observed_species及Simpson指数呈显著正相关;有机质与Chao1指数、Simpson指数呈显著正相关.由于本次采集的土壤均属于盐碱土, 因此, pH值与电导率对土壤细菌群落多样性指标未形成显著性影响.可见, 有效磷、速效钾和有机质是影响土壤细菌多样性指数的主要环境因子.
| 表 3 土壤理化性质与土壤细菌多样性指数之间的相关性 Table 3 Correlation analysis between soil bacterial diversity index and soil physical and chemical properties |
土壤细菌群落结构对土壤理化性质的变化较为敏感, 尤其是土壤养分和酸碱度对土壤细菌群落结构的影响最为明显(徐志霞等, 2018;张媚等, 2018).本研究通过细菌群落与土壤理化性质的相关性分析(表 4)发现, pH值与酸杆菌门细菌存在显著负相关, 但却与浮霉菌门细菌呈显著正相关.这是由于酸杆菌门细菌属于嗜酸菌, 更适宜在酸性环境中生存, 而浮霉菌门则易于在碱性环境中存活, 这与先前的研究相一致(Sait et al., 2006).有机质含量较高的土壤细菌丰富度将显著高于低有机质含量的土壤, 尤其是纤维素分解菌(Yang et al., 2014).酸杆菌门细菌具有大量编码纤维素酶和淀粉水解酶的基因序列, 这充分说明了酸杆菌门细菌在土壤中具有分解动植物残体产生有机质的重要作用(Pattanop et al., 2011).这与本次实验得出的结果相吻合, 通过相关分析发现, 有机质与酸杆菌门细菌显著正相关.而绿弯菌门细菌需要消耗有机质来供应代谢活动所需的能量(Bowman et al., 2013), 因此, 研究区的土壤有机质含量与绿弯菌门细菌呈现显著负相关.
| 表 4 门水平上主要细菌群落与土壤理化性质的相关关系 Table 4 Correlation analysis among physico-chemistry characteristics and bacteria on phylum |
根据目前已有的研究发现, 不同土地利用方式对土壤细菌群落结构多样性具有较大的影响(秦红等, 2017;张双双等, 2019).黄河三角洲盐碱地不同土地利用方式下的土壤细菌群落结构多样性同样存在显著差异, 本文选择的耕地、园地及荒地具有一定代表性.实验结果表明, 荒地中的放线菌门和变形菌门细菌相对丰度均高于园地和耕地, 而这两种菌群具有提高土壤质量、促进植株生长的作用(Mitchell et al., 2009;刘灵芝等, 2013), 可见较少的人为干扰可以有效保护土壤生态系统的韧性.
此外, 酸杆菌门、绿弯菌门和浮霉菌门细菌在样品中的含量也较高, 分别占总测序序列的10%~21%、5%~11%和3%~10%, 它们都是土壤中比较重要的细菌种群, 其普遍性及相对丰度也表明它们在土壤生物化学循环系统中所起的关键作用(万盼等, 2018).酸杆菌门细菌可以分解植物凋落物, 改善土壤碳循环系统(Eichorst et al., 2007), 浮霉菌可以净化和改善土壤水环境(Chen et al., 2015).这两种菌群在耕地和园地土壤中的丰富度远高于荒地, 可见人工施肥和秸秆还田在提高土壤养分的同时也有益于酸杆菌门和浮霉菌门细菌的生长.
5 结论(Conclusions)黄河三角洲盐碱地不同土地利用方式对土壤细菌群落结构具有较大的影响, 不同植物通过根系活动和凋落物等改变土壤环境, 以主动选择提高或降低特定土壤细菌群落的相对丰度和多样性.半自然状态下的荒地土壤细菌相对丰度及多样性均高于人工种植状态下的耕地和园地, 可见人为干扰是影响土壤细菌群落的关键因素.种植人工作物的耕地和园地土壤中细菌群落结构相似度较高, 与受人为干扰较少的荒地土壤细菌种群之间存在较大的差异.荒地土壤中放线菌门和变形菌门这两种菌类的相对丰度均高于园地和耕地, 更有利于其健康土壤生态环境的营造.在不同的土地利用方式下, 土壤有效磷、速效钾和有机质含量是显著影响土壤细菌多样性指数的环境因子.
白雪娟, 曾全超, 安韶山, 等. 2018. 子午岭人工林土壤微生物生物量及酶活性[J]. 应用生态学报, 2018, 29(8): 2695–2704.
|
包明, 何红霞, 马小龙, 等. 2018. 化学氮肥与绿肥对麦田土壤细菌多样性和功能的影响[J]. 土壤学报, 2018, 55(3): 734–743.
|
Bell C W, Asao S, Calderon F, et al. 2015. Plant nitrogen uptake drives rhizosphere bacterial community assembly during plant growth[J]. Soil Biology and Biochemistry, 85: 170–182.
DOI:10.1016/j.soilbio.2015.03.006
|
Bowman K, Nobre M, Da M C. 2013. Dehalogenimonas alkenigignens sp.nov.a chlorinated-alkane-dehalogenating bacterium isolated from groundwater[J]. International Journal of Systematic & Evolutionary Microbiology, 64(4): 1492–1498.
|
Chandrasekaran M, Boughattas S, Hu S, et al. 2014. A meta-analysis of arbuscular mycorrhizal effects on plants grown under salt stress[J]. Mycorrhiza, 24(8): 611–625.
DOI:10.1007/s00572-014-0582-7
|
Chatterjee P, Samaddar S, Anandham R, et al. 2017. Beneficial soil bacterium pseudomonas frederiksbergensis OS261 augments salt tolerance and promotes red pepper plant growth[J]. Frontiers in Plant Science, 8: 1–9.
|
陈重军, 张海芹, 汪瑶琪, 等. 2016. 基于高通量测序的ABR厌氧氨氧化反应器各隔室细菌群落特征分析[J]. 环境科学, 2016, 37(7): 2652–2658.
|
Chen Y, Wen Y, Tang Z, et al. 2015. Effects of plant biomass on bacterial community structure in constructed wetlands used for tertiary wastewater treatment[J]. Ecological Engineering, 84: 38–45.
DOI:10.1016/j.ecoleng.2015.07.013
|
Eichorst S A, Breznak J A, Schmidt T M. 2007. Isolation and characterization of soil bacteria that define terriglobus gen.nov., in the phylum acidobacteria[J]. Applied and Environmental Microbiology, 73(8): 2708–2717.
DOI:10.1128/AEM.02140-06
|
冯晨, 孙占祥, 王宝荣, 等. 2018. 辽西地区覆膜条件对不同土层土壤氮磷钾累积量的影响[J]. 中国土壤与肥料, 2018(5): 129–133.
|
高鹏飞, 姚国强, 赵树平, 等. 2014. 乳酸菌在农产品种植及其质量安全中的研究进展[J]. 中国农业科技导报, 2014, 16(6): 143–148.
|
Gomez E, Pioli R, Conti M. 2007. Fungal abundance and distribution as influenced by clearing and land use in a vertic soil of Argentina[J]. Biology and Fertility of Soils, 43(3): 373–377.
|
孔涛, 张德胜, 徐慧, 等. 2014. 盐碱地及其改良过程中土壤微生物生态特征研究进展[J]. 土壤, 2014, 46(4): 581–588.
DOI:10.3969/j.issn.1674-5906.2014.04.006 |
李倩, 马琨, 冶秀香, 等. 2018. 不同培肥方式对土壤有机碳与微生物群落结构的影响[J]. 中国生态农业学报, 2018, 26(12): 1–8.
|
梁志婷, 邓建强, 王自奎, 等. 2017. 陇东旱塬区不同粮草轮作模式下土壤细菌群落组成特征[J]. 草业学报, 2017, 26(8): 180–191.
|
刘开辉, 张波, 丁小维, 等. 2018. 基于高通量测序的花马盐湖原核微生物及其耐盐基因[J]. 微生物学报, 2018, 58(10): 1743–1753.
|
刘灵芝, 秦嗣军, 吕德国, 等. 2013. 樱桃园土壤优势放线菌的分离及其促长功能研究[J]. 中国农业科学, 2013, 46(6): 1221–1229.
DOI:10.3864/j.issn.0578-1752.2013.06.016 |
刘旻霞, 李瑞, 张灿, 等. 2018. 兰州市南山季节性土壤微生物特征及影响因素[J]. 中国环境科学, 2018, 38(7): 2722–2730.
DOI:10.3969/j.issn.1000-6923.2018.07.044 |
刘兴华, 章海波, 李远, 等. 2018. 黄河三角洲滩涂-湿地-旱地土壤团聚体有机质组分变化规律[J]. 土壤学报, 2018, 56(2): 375–386.
|
缪凌, 董建国, 汪有科, 等. 2016. 黄土丘陵区不同土地利用类型下的深层土壤水分变化特征[J]. 水土保持研究, 2016, 29(2): 13–18.
|
Lu X, Nicol G W, Neufeld J D. 2018. Differential responses of soil ammonia-oxidizing archaea and bacteria to temperature and depth under two different land uses[J]. Soil Biology and Biochemistry, 120: 272–282.
DOI:10.1016/j.soilbio.2018.02.017
|
Lúcio W D S, Lacerda C F D, Mendes Filho P F, et al. 2013. Crescimento e respostas fisiológicas do meloeiro inoculado com fungos micorrízicos arbusculares sob estresse salino[J]. Semina:Ciências Agrárias, 34(4): 1587–1602.
|
Lüneberg K, Schneider D, Siebe C, et al. 2018. Drylands soil bacterial community is affected by land use change and different irrigation practices in the Mezquital Valley, Mexico[J]. Scientific Reports, 8(1): 1–15.
DOI:10.1038/s41598-017-17765-5
|
Ma B, Zhao K, Lv X, et al. 2018. Genetic correlation network prediction of forest soil microbial functional organization[J]. The ISME Journal, 12(10): 2492.
DOI:10.1038/s41396-018-0232-8
|
Mitchell J S, Ruess R W. 2009. N2 fixing alder (Alnus viridis spp.fruticosa) effects on soil properties across a secondary successional chronosequence in interior Alaska[J]. Biogeochemistry, 95(2/3): 215–229.
|
Ndour N Y B, Achouak W, Christen R, et al. 2008. Characteristics of microbial habitats in a tropical soil subject to different fallow management[J]. Applied Soil Ecology, 38(1): 51–61.
|
Ottinger M, Kuenzer C, Liu G, et al. 2013. Monitoring land cover dynamics in the Yellow River Delta from 1995 to 2010 based on Landsat 5 TM[J]. Applied Geography, 44: 53–68.
DOI:10.1016/j.apgeog.2013.07.003
|
Pattanop K, Tanaporn U, Ukrit R. 2011. Insights into the phylogeny and metabolic potential of a primary tropical peat swamp forest microbial community by metagenomic analysis[J]. Microbial Ecology, 61: 518–528.
DOI:10.1007/s00248-010-9766-7
|
秦红, 李昌晓, 任庆水. 2017. 不同土地利用方式对三峡库区消落带土壤细菌和真菌多样性的影响[J]. 生态学报, 2017, 37(10): 3494–3504.
|
Qadir M, Tubeileh A, Akhtar J, et al. 2008. Productivity enhancement of salt-affected environments through crop diversification[J]. Land Degradation & Development, 19(4): 429–453.
|
Ramirez K S, Craine J M, Fierer N. 2012. Consistent effects of nitrogen amendments on soil microbial communities and processes across biomes[J]. Global Change Biology, 18(6): 1918–1927.
DOI:10.1111/gcb.2012.18.issue-6
|
Sait M, Davis K E R, Janssen P H. 2006. Effect of pH on isolation and distribution of members of subdivision 1 of the phylum Acidobacteria occurring in soil[J]. Applied and Environmental Microbiology, 72(3): 1852–1857.
DOI:10.1128/AEM.72.3.1852-1857.2006
|
万盼, 胡艳波, 张弓乔, 等. 2018. 甘肃小陇山油松与柴胡栽培土壤细菌群落特征[J]. 生态学报, 2018, 38(17): 1–8.
|
王颖, 宗宁, 何念鹏, 等. 2018. 青藏高原高寒草甸不同海拔梯度下土壤微生物群落碳代谢多样性[J]. 生态学报, 2018, 38(16): 1–8.
|
王影, 张志明, 李晓慧, 等. 2013. 土地利用方式对土壤细菌、泉古菌和氨氧化古菌丰度的影响[J]. 生态学杂志, 2013, 32(11): 2931–2936.
|
Wang X, Wang Q, Guo X, et al. 2015. Functional genomic analysis of Hawaii marine metagenomes[J]. Science Bulletin, 60(3): 348–355.
DOI:10.1007/s11434-014-0658-y
|
夏昕, 石坤, 黄欠如, 等. 2015. 长期不同施肥条件下红壤性水稻土微生物群落结构的变化[J]. 土壤学报, 2015, 52(3): 697–705.
|
徐志霞, 王璇, 李慧敏, 等. 2018. 不同林龄木麻黄林地土壤细菌及与土壤因子的相关性分析[J]. 基因组学与应用生物学, 2018, 37(2): 780–788.
|
Xu M, Li X, Cai X, et al. 2017. Land use alters arbuscular mycorrhizal fungal communities and their potential role in carbon sequestration on the Tibetan Plateau[J]. Scientific Reports, 7(1): 1–11.
DOI:10.1038/s41598-016-0028-x
|
杨劲松. 2008. 中国盐渍土研究的发展历程与展望[J]. 土壤学报, 2008, 45(5): 837–845.
DOI:10.3321/j.issn:0564-3929.2008.05.010 |
Yang J, Zhang J, Yu H. 2014. Community composition and cellulase activity of cellulolytic bacteria from forest soils planted with broad-leaved deciduous and evergreen trees[J]. Applied Microbiology and Biotechnology, 98(3): 1449–1458.
DOI:10.1007/s00253-013-5130-4
|
张媚, 林马水, 曹秀秀, 等. 2018. 不同经营模式山核桃林地土壤pH值、养分与细菌多样性的差异[J]. 生物多样性, 2018, 26(6): 611–619.
|
张双双, 靳振江, 贾远航, 等. 2019. 岩溶地区不同土地利用方式土壤固碳细菌群落结构特征[J]. 环境科学, 2019, 40(1): 1–12.
DOI:10.3969/j.issn.1673-2049.2019.01.001 |
张彦军. 2018. 环境因子对土壤微生物呼吸及其温度敏感性变化特征的影响[J]. 环境科学, 2018, 39(6): 1–21.
|
Zhang S, Fan C, Wang Y, et al. 2018. Salt-tolerant and plant growth-promoting bacteria isolated from high-yield paddy soil[J]. Can J Microbiol: 1–33.
|
 2019, Vol. 39
2019, Vol. 39


