2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085;
3. 中国城市建设研究院有限公司, 北京 100120
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environment Sciences, Chinese Academy of Sciences, Beijing 100085;
3. China Urban Construction Design and Research Institute Corporation Limited, Beijing 100120
人类活动对全球氮循环的干扰和影响, 远超过其它元素循环, 极大地加速了地球表层环境的变化.目前由人类活动引发的全球固氮量已经远远超过自然作用的总和, 严重破坏了原有的氮循环平衡, 导致一系列严重的氮污染问题(Zhu et al., 2011).在微生物驱动下, 通过一系列氧化还原反应转化成不同的氮化合物, 包括硝化、反硝化、厌氧氨氧化以及硝酸盐异化还原等(Jetten et al., 2009).其中硝酸盐异化还原为铵(英文, DNRA), 不同于反硝化, DNRA是将NO3-还原为NH4+而不是N2, 产生的NH4+在硝化作用下重新生成NO2-和NO3-, 再次被反硝化、DNRA和厌氧氨氧化菌利用, 从而说明DNRA是氮循环重要的内在衔接过程(Zhou et al., 2014).由于检测方法的局限, DNRA的重要性一直被忽视.
关于DNRA的研究, 科学家们针对DNRA生态、生化机理方面进行研究, Rütting运用分子生物学及同位素标记的方法探讨了DNRA细菌的菌群结构和速率(Rütting et al., 2008).通过分子生物学的技术发现DNRA功能细菌主要是变型细菌(Rooks et al., 2012), 得到DNRA的细菌以兼氧厌氧型的细菌占主导地位, 厌氧和好氧型细菌占辅导地位(Mohan et al., 2007).目前, DNRA研究主要集中在海洋和河口湿地沉积物(An et al., 2002; Roberts et al., 2014; Bonaglia et al., 2017).在淡水生态系统中, DNRA的细菌分布、群落和活动性仍不为人们所熟知.
岸边带是水体与陆地群落之间的过渡地带, 能有效地控制住水生态系统的污染物, 从而防止污染物从陆地向河流迁移.研究发现(Hefting et al, 2006; Wang et al., 2010), 由于水位的频繁变化, 岸边带常受缺氧/好氧条件的波动影响, 具有较强的氧化还原驱动力, 成为氮循环的热点.特别是岸边带对NO3-起着“下沉”的作用(Groffman et al., 2002; Hefting et al, 2006; Wang et al., 2018), 而NO3-是最常见的地下水污染物, 也是DNRA过程的重要参与者.但目前对于DNRA过程在湿地岸边带的研究尚且不足.
因此, 本文选择中国南北方典型湿地8个点作为研究地点, 针对DNRA过程中的nrfA功能基因在Illumina MiSeq PE250平台进行高通量测序, 确定中国南北方典型湿地岸边带表层沉积物中的DNRA细菌的群落结构及其生物多样性, 然后应用多元统计分析方法, 将典型湿地影响其生物地理的潜在环境因子联系起来.旨在进一步了解硝酸盐异化还原为铵过程和DNRA细菌的生物多样性.
2 材料和方法(Materials and methods) 2.1 样品采集与保存本研究采集中国8个典型湿地岸边带的表层沉积物样品(深度0~10 cm), 样点按纬度从高到低排序:嘉兴(Jiaxing)、巢湖(Chaohu)、兰州黄河(Lanzhou)、白洋淀(Baiyangdian)、天津永定新河(Tianjin)、北京香山山泉(Beijing)、盘锦(Panjin)、乌梁素海(Wuliangsu).采样点具体信息见表 1.其中嘉兴巢湖为典型南方湿地样点, 兰州、白洋淀、天津、北京、盘锦以及乌梁素为典型北方湿地样点.每个采样点随机取6个表层沉积物样品, 去除植物残体和螺类动物残骸等杂质物后充分混匀.采集样品完毕, 用无菌自封袋密封保存后, 再用冰盒储存带回实验室.样品到达立即测其各项理化性质, 放至4 ℃冰箱储存, 剩下部分经冷冻干燥后保存在-80 ℃超低温冰箱用于DNA提取和后续的分子生物实验.
| 表 1 典型湿地沉积物采样点信息 Table 1 Information of sites in wetland sediments |
按照标准检测方法《土壤农化分析》(鲍士旦, 2000)进行相应理化因子的测定.其中含水率(MC)以样品在105 ℃下的失水量计算; 总有机质(TOM)是以样品在550 ℃的烧失量计算(LOI550)(张文河等, 2007); 向土样中按照土壤/水为1:5的质量比加入超纯水浸提1 h后测定其pH值; 用KCl溶液(2 mol·L-1)对土壤样品进行浸提, 利用连续流动分析仪(SAN plus, Skalar Analytical B.V., Breda, the Netherlands)测定土壤氨氮(NH4+-N)、亚硝酸盐氮(NO2--N)和硝酸盐氮(NO3--N)的浓度; 将风干的土壤样品过100目筛(0.149 mm)后利用元素分析仪对土壤总氮(TN)、总碳(TC)和总硫(TS)同时进行测定.样品所有理化性质都进行3次重复测定取平均值(表 2).
| 表 2 湿地沉积物理化性质 Table 2 Chemical characteristics of wetland sediments |
利用FastDNA Spin Kit for Soil DNA提取试剂盒(MP Biomedicals, USA), 参照说明书, 称取约0.33 g冷冻干燥处理后的土壤样品, 提取样品中的总DNA, 用1%琼脂糖凝胶对提取的DNA进行检测, 并用Nano Drop 2000 UV-Vis Spectrophotometer (Thermo Fisher Scientific, USA)测定其DNA浓度, 在260 nm和280 nm处的吸光度比值均为1.8左右, 表明获得了高质量的DNA.使用DNRA菌的nrfA功能基因的特异性引物nrfA-F2aw (5′-CARTGYCAYGTBGARTA-3′)和nrfA-R1 (5′-TWNGGCATRTGRCARTC-3′)对样品DNA片段进行PCR扩增(Welsh et al., 2014).扩增体系为50 μL:10×buffer 5 μL, dNTP (2.5 mmol·L-1) 4 μL, 浓度为10 mmol·L-1的前、后引物各1 μL, BSA 0.5 μL, Taq酶(2.5 U) 0.25 μL, 稀释10倍的DNA模板4 μL, 用ddH2O补足50 μL (Zhu et al., 2015).扩增条件为:95 ℃预变性10 min; 95 ℃变性30 s, 52 ℃退火30 s, 72 ℃延伸20 s, 40个循环; 72 ℃延伸10 min.取5 μL PCR扩增产物用1%凝胶进行电泳验证, 通过Illumina MiSeq PE250平台进行测序(奥维森生物信息科技有限公司, 北京).
2.4 实时荧光定量PCR使用实时荧光定量PCR采用SYBR Green Ⅰ染色法, 所用试剂为SYBR Premix Ex Taq TM试剂盒(TaKaRa, 大连).实验选用特异性引物nrfA-F2aw和nrfA-R1针对功能基因nrfA进行丰度的定量分析.qPCR的反应为20 μL体系:SYBR Premix Ex Taq酶10 μL, ROX 0.4 μL, 牛血清蛋白(BSA) 0.4 μL, 正反向引物(10 mmol·L-1)各1 μL, 稀释10倍的DNA模板2 μL, 无核酸酶纯水补足至20 μL.实验使用ABI 7500 Real-Time PCR System扩增仪(Applied Biosystems, CA, USA)进行定量.扩增测序如下(Mohan et al., 2004; Welsh et al., 2014; Song et al., 2014):95 ℃预变性5 min, 94 ℃变性30 s, 53 ℃退火45 s, 72 ℃延伸30 s, 共40循环.按10倍浓度梯度的稀释质粒, 设置7个梯度, 3个平行.根据梯度曲线扩增得的Ct值和拷贝数绘制标准曲线; 每个样品设有3个平行样, 用7500 SDS System配套软件进行分析计算数据结果, 得出DNRA细菌丰度值.其中, 阴性对照组定量结果须为未检测到(ND), 标准样品扩增效率介于90%~110%, 标准曲线相关性大于0.98, 扩增溶解曲线为单一峰.
2.5 序列分析高通量测序时用带有Barcode信息的引物对样品nrfA进行PCR扩增以区分样品.依托Illumina MiSeq PE250平台完成nrfA高通量测序, 由奥维森公司构建高通量基因测序文库.测序公司返回Fasta格式序列结果, 后续进行序列处理, 步骤如下:首先通过Uchime、Qiime、Mothur、Excel、Notapad和Bioedit等软件检测并筛除低质量的序列, 且得到非冗余的核酸序列(Schloss et al., 2009).通过Bioedit软件将非冗余的核酸序列翻译成氨基酸序列, 然后通过Uclust软件以90%的相似度将氨基酸序列进行聚类(Edgar, 2010; Wang et al., 2018), 得到OTUs的分布结果.基于OTUs水平进行生态学分析:将每个OTUs代表的核酸序列在Genbank数据库上进行BLAST(Basic Local Alignment Search Tool)比对; 通过编写的Perl脚本计算多样性等(庄林杰等, 2017).
2.6 统计分析使用去趋势对应分析(DCA)来确定数据中的单峰性或线性度.冗余分析(RDA)来确定这些环境变量解释的方差和DNRA群落结构的分布.根据Bray-Curtis差异度指数进行群落的分类和排序, 分类选择UPGMA算法(Mothur计算), 排序选用主坐标分析(Principle Coordinate Analysis, PCoA).用SPSS21.0软件(Statistical Product and Service Solutions)进行相关性分析(均采用Spearman相关系数), 本研究的显著性水平为α≤ 0.05.利用微软Office、Origin9.0及Heatmap软件作图.
3 结果(Results) 3.1 DNRA细菌的高通量测序结果从典型湿地沉积物样品中提取DNA后, 用PCR扩增nrfA基因, 并在Illumina MiSeq PE250平台上进行测序.由生物信息学软件对序列进行严格控制, 随机抽平各样点序列为60000条.结果表明, 质量控制后的序列均为DNRA细菌的序列.经过质量控制, 得到高质量的序列数量和90%聚类的相似度, 得到OTU数量和覆盖率分布(表 3).从表中的统计结果可以看出, OTU数在132~263之间, 嘉兴OTU数最多, 乌梁素海的OTU数最少, 南方样品的OTU数大于北方, 覆盖率均在97%以上.以500条序列为梯度, 进行连续累加来计算OTUs的数量, 得出OTUs随序列数增加的变化趋势.随着序列数的增大, 样本中的OTUs数量增加趋势逐渐趋于平缓,表明测序数据充足, 结果与覆盖率指数相符, 则说明本文用的高通量数据可用.
| 表 3 高通量测序样品描述 Table 3 Summary of high-throughput sequencing results |
通过实时荧光定量PCR, 得到8个典型湿地沉积物样点的丰度, 结果如图 1所示, DNRA细菌的丰度最高在巢湖, 为(8.44±0.48)×108 copies·g-1, 最低在白洋淀, 为(6.69±0.28)×107 copies·g-1.湿地沉积物DNRA细菌的丰度可以划分为两个水平级, 在南方丰度相对较高, 范围在(7.31±0.26)×108~(8.44±0.48)×108 copies·g-1, 而北方样点的DNRA菌的丰度较低, 范围在(6.69±0.28)×107~(1.92±0.61)×108 copies·g-1.南北方湿地样点的DNRA细菌的丰度值存在着明显的差异.
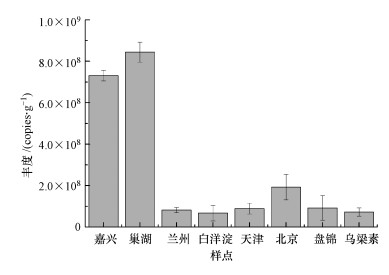 |
| 图 1 典型湿地沉积物DNRA细菌的丰度 Fig. 1 Abundance of DNRA bacteria in wetland sediments |
典型湿地沉积物中DNRA细菌的所有序列通过质控, 选取涵盖每个注释种属99%序列数的OUTs, 对样品和OUTs进行双向二维聚类, 绘制Heatmap图, 通过颜色的梯度和相似程度来反映数据的相似性和差异性, 可以清楚地获知各OUT下的DNRA来源和每一个样品下的DNRA的群落分布.从颜色深浅代表的reads数目来看, 由图 2可以看出, 各样点的菌群结构差异度较大, 而通过聚类分析可得, 各样本聚类时体现出一定的空间效应, 纬度相近的样品先聚类, 其中样点嘉兴、巢湖、白洋淀聚为一类, 兰州、北京、盘锦聚为一类, 天津和乌梁素分别为一类.说明空间的差异度对DNRA的菌群结构影响较大.根据NCBI数据库中已探明的纯培养DNRA细菌的nrfA基因序列作为参比序列, 利用BLAST(basic local alignment search tool)进行序列比对和注释.按照门分类, 各样点的OUTs注释到了变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、绿弯菌门(Chloroflexi) 等8个门, 共8个典型湿地样品中序列隶属于Proteobacteria 门的占61.31%, 隶属于其他门的序列不多, 分别不足10%.按照门水平的分类, 各样点差异度不大, 说明DNRA菌属在典型湿地中, 隶属于Proteobacteria 门的DNRA细菌占据主导地位.
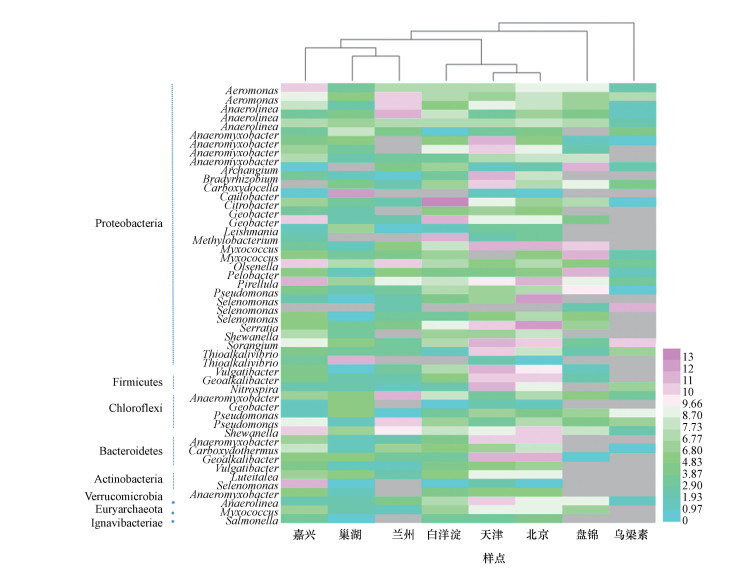 |
| 图 2 典型湿地表层土壤DNRA细菌的热图 Fig. 2 The heatmap of DNRA bacteria in wetland sediments |
在Uclust软件以90%相似度下进行聚类, 共得到274个OTUs, 挑出每个OTUs的代表氨基酸序列.按通过序列比对分析做出相对丰度图, 如图 3所示, 按照属水平的分类, 各样点的OUTs注释到了Anaeromyxobacter等23个属, 除了兰州黄河湿地样点中Vulgatibacter菌相对丰度最高, 其他样点均是Anaeromyxobacter菌相对丰度占领主要地位.其中相对丰度最高的Anaeromyxobacter、Anaerolinea和Dokdonella 分别占24.71%、9.70%和7.94%.除了厌氧绳菌(Anaerolinea)属于绿弯菌门(Chloroflexi), Anaeromyxobacter、Dokdonella和Vulgatibacter均属于变形菌门(Proteobacteria).由图可知, 按照属水平划分, 各样点之间菌属的相对分布各有不同, 不同湿地沉积物的DNRA菌落结构差别较大.
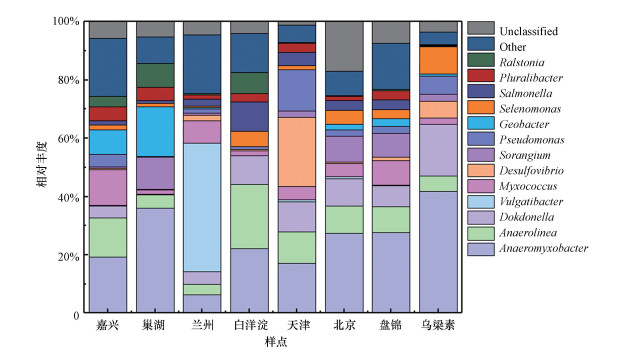 |
| 图 3 典型湿地DNRA细菌的相对丰度图 Fig. 3 Relative abundance of DNRA bacteria in wetland sediments |
本研究中涉及的α-多样性指数包括丰富度指数(Chao1指数)、优势度指数(Simpson指数)和多样性指数(Shannon指数).Chao1指数是指群落中含OTUs数目的指数, 值越高说明群落内物种的数目越多.Shannon指数主要反映群落种数和各种间个体分配的均匀性; Simpson指数表示在一个群落中随机取样的2个个体属于不同种的概率.表 4所示的各样点α-多样性指数由软件Mothur计算所得.由表 4可知, Chao指数最高的是巢湖样点(15234.20), 最低的是天津永定新河沉积物(4060.58);Shannon指数最高的是盘锦(7.4634), 最低的是天津永定新河沉积物(5.6318);Simpson指数最高的兰州黄河沉积物(0.0319), 最低的是嘉兴湿地沉积物(0.0024).通过α-多样性指数分析可得巢湖和嘉兴湿地样点的物种多样性相对较高, 而天津、乌梁素等样点的物种多样性相对较低.Chao指数南方样点远大于北方样点, 而南北方样点的Shannon指数和Simpson指数差异不大, 由此在一定程度上可以反应南方典型湿地沉积物中的DNRA细菌的物种多样性大于北方.
| 表 4 典型湿地表层土壤DNRA细菌的α-多样性指数 Table 4 α-diversity index of DNRA bacteria in wetland sediments |
在OTUs水平计算典型湿地表层沉积物样品的Bray-Curtis差异度矩阵, 利用Qiime软件做主坐标轴图(Principal Coordinate Axis Analysis, PCoA)(图 4).如图 4所示, 两轴对排序结果的解释度分别为35.81%和17.22%, 数值较低, 说明样本之间的遗传亲缘度不高, 即使是同一类别的湿地表层沉积物, DNRA细菌群落依然显示出较大差异, 这说明土地类型并不是影响湿地DNRA细菌群落结构的重要因素, 两轴较低的解释度也说明可能还存在其他影响因素.图中结果显示, 无论从第一轴还是第二轴, 图中均呈现出较为一致的规律:南方典型湿地样点嘉兴和巢湖聚集在一起, 说明南方两个样点的群落结构较为相似; 而北方典型湿地样点乌梁素、白洋淀、天津、兰州、北京及盘锦聚集在一起, 说明北方6个湿地样点的群落结构比较类似.
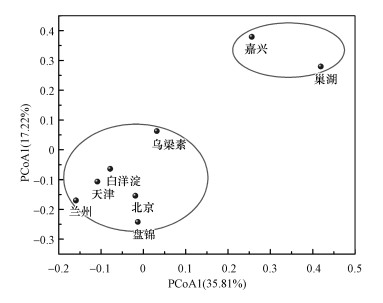 |
| 图 4 典型湿地表层土壤DNRA菌的PCoA图 Fig. 4 Principal coordinates analysis of DNRA bacteria in wetland sediments |
通过各样点DNRA菌的nrfA功能基因的丰度及菌群相对丰度, 结合理化性质进行SPSS相关性分析(表 5)和冗余分析(RDA)(图 5)来探究环境影响因子对中国典型湿地沉积物中DNRA细菌的群落结构的影响.
| 表 5 典型湿地表层沉积物DNRA菌群与环境因子相关性分析 Table 5 Correlation analysis between community structure of DNRA bacteria and chemical characters of soil (n=8) |
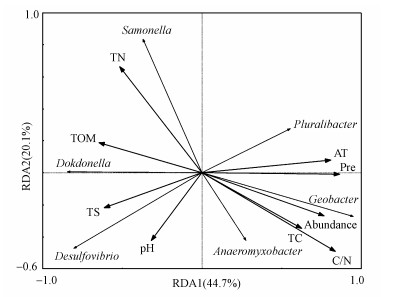 |
| 图 5 典型湿地表层沉积物RDA图 Fig. 5 RDA analysis of DNRA bactreial community and environmental factors |
先通过CANOCO4.5软件进行去趋势对应分析(DCA), 分析结果为0.9, 适合做RDA.将高通量测序得出的DNRA菌群与重要的理化因子作矩阵, 研究其与环境因子之间的关系并得到图 5.由RDA和SPSS分析可得, DNRA菌的丰度与TN显著负相关(r=-0.929, p=0.001), 与碳氮比(C/N)(r=0.762, p=0.028)显著正相关, 和年平均降水量(r=0.905, p=0.002)显著正相关.典型湿地DNRA细菌的相对丰度较高的6个种属与环境因子之间的相关性:Anaeromyxobacter属与TS显著负相关(r=-0.719, p=0.045) ; Dokdonella属与年平均降水量(r=-0.738, p=0.037)和年平均温度(r=-0.833, p=0.010)显著负相关; Desulfovibrio属与年平均温度(r=-0.714, p=0.047)显著负相关, Geobacter属与TN显著负相关(r=-0.905, p=0.002) 与碳氮比(C/N)(r=0.833, p=0.010)和年平均温度(r=0.762, p=0.028)显著正相关; Salmonella属与C/N(r=-0.786, p=0.021)显著负相关; Pluralibacter属与年平均温度(r=0.881, p=0.004)显著正相关.其它种属和地理环境因子之间没有显示出显著的相关性.
4 讨论(Discussion)本文运用高通量测序技术, 针对中国8个典型湿地岸边带表层沉积物中的DNRA过程进行实验研究.分析了DNRA细菌在湿地沉积物系统中的分布规律、群落结构、多样性、定量丰度等, 并结合地理理化因子进行了分析.
结合DNRA菌的α-多样性、β-多样性和DNRA菌群落结构的组成分布可以知道, 在8个中国典型湿地岸边带沉积物取样点中, α-多样性指数均是呈现出南方样点(巢湖、嘉兴)的物种多样性大于北方样点(兰州、白洋淀、天津、北京、盘锦、乌梁素), 其中南方样点的丰富度指数远大于北方, 可能是因为南方的降水量及年平均温度高于北方而引起的(Yang et al., 2004; Robertson et al., 2010).由β-多样性分析发现, 南北湿地样点的DNRA细菌的群结构按照南北地区差异聚类为两大类, 可能是环境温度对DNRA细菌的菌群结构带来的潜在影响.由于各采样点纬度的差异导致温度差异, 加上各地地理因子和环境因子差异等的综合因素, 造成了各样点下DNRA菌群落组成的不同.
有研究表明, 温度的变化决定了该样点微生物的种群结构组成的变化(Ogilivie et al., 1997), 而温度对DNRA细菌的群落结构、丰度以及活性也有着重要影响(Tomaszek et al., 2007; Dunn et al., 2013; Wang et al., 2018).从DNRA细菌实时定量分析结果发现:中国南北典型湿地样点的DNRA菌群丰度的差异性明显, 而温度与DNRA的速率呈正相关(Bradbury et al., 2007).当温度低于10 ℃时, DNRA几乎完全被抑制(Ribas et al., 2015).17~22 ℃是DNRA的最佳温度(Kelly-Gerreyn et al., 2007).而本研究中南方的典型湿地样点的年平均温度均高于17 ℃, DNRA细菌的丰度明显高于年平均温度均低于13 ℃的北方湿地样点, 从而进一步确认了温度对DNRA细菌的影响.
传统观点一直认为DNRA过程只发生于缺氧和厌氧环境, 而且由于检测方法的局限, DNRA的重要性一直被忽视.近5年最新研究发现, 在好氧条件下也会发生DNRA过程, 其反应速率甚至要高出缺氧状态下一个数量级水平(Steenkamp et al., 1981; Sher et al, 2008; Lu et al., 2013).本文基于nrfA功能基因的高通量研究方法, 样品中所检测到的DNRA细菌最主要隶属于厌氧粘细菌(Anaeromyxobacter)属.厌氧粘细菌是存在于多种土壤和沉积物环境中的细菌(Thomas et al., 2007).这就解释了Anaeromyxobacter在整体上以及在各个地点上处于主导地位的结果.通过相对丰度图和热图可以看出各个样点的DNRA细菌的菌群结构差异度较大, 是由于环境因子的差异导致DNRA细菌的菌群的差异(Em et al., 2015).结合SPSS和RDA的相关性分析, DNRA菌的丰度与TN显著负相关(r=-0.929, p=0.001), 与C/N(r=0.762, p=0.028)显著正相关, 已有研究表明, 不同生态系统土壤中的DNRA速率与TN、C/NO3-和土壤C/N比显著相关(Risgaard-Petersen et al., 2003; Chen et al., 2015).在低溶解氧和低氧化还原电位的环境下, 高C浓度和高C/N比可导致DNRA高于反硝化速率, 占据着更重要的地位(Bonin et al., 1996;Rütting et al., 2008; Jahangir et al., 2017), 而本研究中南方典型湿地样点C/N值较高, 均在22以上, 这是促进DNRA过程的一个重要影响因素.从而南方湿地样点DNRA细菌的丰度和物种多样性更高得到了进一步的解释.在高硝酸盐负荷影响下, 反硝化作用主要受硝酸盐限制, 而有机物的富集和还原条件使硝酸盐还原向DNRA方向移动(Nizzoli et al., 2010).中国南方典型是湿地样点中的硝酸盐浓度大于北方湿地样点, 这对于南方湿地DNRA细菌的丰度相对较高也有着一定的影响.一般研究认为, 硫化物可促进DNRA过程的发生, DNRA过程可以通过无机化能自养硫细菌氧化达到去除硝酸盐的目的(Steenkamp et al., 1981; Sher et al., 2008; Lu et al., 2013).而本文通过相关性分析发现总硫对中国典型湿地样点的DNRA细菌的影响较小, 可能是由于各典型湿地样点中硫化物含量较低且样点间硫化物浓度差异度不大, 导致硫化菌对典型湿地样点的作用不明显.
综上, 本文运用高通量测序技术对DNRA功能基因nrfA扩增, 一定程度上揭示了不同地理分布的典型湿地沉积物中DNRA细菌的丰度、群落组成、多样性、及其与部分理化因子之间的关系.但DNRA细菌各种属和关键理化因子间的生物调节机制, DNRA过程的关键驱动因素以及与其他氮循环过程的耦合机理等, 都有待进一步研究.
An S, Gardner W. 2002. Dissimilatory nitrate reduction to ammonium (DNRA) as a nitrogen link, versus denitrification as a sink in a shallow estuary (Laguna Madre/Baffin Bay, Texas)[J]. Marine Ecology Progress, 237(1499): 41–50.
|
鲍士旦. 2000. 土壤农化分析(第3版)[M]. 北京: 中国农业出版社.
|
Bonaglia S, Hylén A, Rattray J E, et al. 2017. The fate of fixed nitrogen in marine sediments with low organic loading: an in situ study[J]. Biogeosciences, 14(2): 1–31.
|
Bradbury D C, Firestone M K. 2007. Environmental control of microbial N transformations in redwood forests [R]. In (eds Standiford R B, Giusti G A, Valachovic Y, et al.), Proceedings of the Redwood Region Forest Science Symposium: What Does the Future Hold? Gen. Tech. Rep. PSW-GTR-194. Pacific Southwest Research Station, Forest Service, U.S. Department of Agriculture, Albany, CA, USA, 203-204
|
Chen Z, Wang C, Gschwendtner S, et al. 2015. Relationships between denitrification gene expression, dissimilatory nitrate reduction to ammonium and nitrous oxide and dinitrogen production in montane grassland soils[J]. Soil Biology and Biochemistry, 87: 67–77.
DOI:10.1016/j.soilbio.2015.03.030
|
Dunn R J K, Robertson D, Teasdale P R, et al. 2013. Benthic metabolism and nitrogen dynamics in an urbanised tidal creek: Domination of DNRA over denitrification as a nitrate reduction pathway[J]. Estuarine, Coastal and Shelf Science, 131: 271–281.
DOI:10.1016/j.ecss.2013.06.027
|
Edgar R C. 2010. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 26(19): 2460.
DOI:10.1093/bioinformatics/btq461
|
Em V D B, Van D U, Abbas B, et al. 2015. Enrichment of DNRA bacteria in a continuous culture[J]. Isme Journal Multidisciplinary Journal of Microbial Ecology, 9(12): 2749.
|
Groffman P M, Boulware N J, Zipperer W C, et al. 2002. Soil nitrogen cycle processes in urban riparian zones[J]. Environmental Science & Technology, 36(21): 4547–4552.
|
Jahangir M M R, Fenton O, Müller C, et al. 2017. In situ, denitrification and DNRA rates in groundwater beneath an integrated constructed wetland[J]. Water Research, 111: 254–264.
DOI:10.1016/j.watres.2017.01.015
|
Jetten M S, Lv N, Strous M, et al. 2009. Biochemistry and molecular biology of anammox bacteria[J]. Crit Rev Biochem Mol Biol, 44(2/3): 65–84.
|
Hefting M M, Bobbink R, Janssens M P. 2006. Spatial variation in denitrification and N2O emission in relation to nitrate removal efficiency in a N-stressed riparian buffer zone[J]. Ecosystems, 9(4): 550–563.
DOI:10.1007/s10021-006-0160-8
|
Hefting M, Clément J C, Dowrick D, et al. 2004. Water table elevation controls on soil nitrogen cycling in riparian wetlands along a European climatic gradient[J]. Biogeochemistry, 67(1): 113–134.
|
Kelly-Gerreyn B A, Trimmer M, Hydes D J. 2001. A diagenetic model discriminating denitrification and dissimilatory nitrate reduction to ammonium in a temperate estuarine sediment[J]. Marine Ecology Progress, 220(1): 33–46.
|
Lu W W, Zhang H L, Shi W M. 2013. Dissimilatory nitrate reduction to ammonium in an anaerobic agricultural soil as affected by glucose and free sulfide[J]. European Journal of Soil Biology, 58(58): 98–104.
|
Mohan S B, Cole J A. 2007. Chapter 7-the dissimilatory reduction of nitrate to ammonia by anaerobic bacteria[J]. Biology of the Nitrogen Cycle: 93–106.
|
Nizzoli D, Carraro E, Nigro V, et al. 2010. Effect of organic enrichment and thermal regime on denitrification and dissimilatory nitrate reduction to ammonium (DNRA) in hypolimnetic sediments of two lowland lakes[J]. Water Research, 44(9): 0–2724.
|
Ogilvie B G, Rutter M, Nedwell D B. 1997. Selection by temperature of nitrate-reducing bacteria from estuarine sediments: species composition and competition for nitrate[J]. FEMS Microbiology Ecology, 23(1): 11–22.
|
Ribas D, Calderer M, Martí V, et al. 2015. Effect of different seasonal conditions on the potential of wetland soils for groundwater denitrification[J]. Desalination & Water Treatment, 53(4): 994–1000.
|
Risgaard-Petersen N, Nielsen L P, Rysgaard S, et al. 2003. Erratum: Application of the isotope pairing technique in sediments where anammox and denitrification co-exist[J]. Limnology & Oceanography Methods, 1(1): 63–73.
|
Roberts K L, Kessler A J, Grace M R, et al. 2014. Increased rates of dissimilatory nitrate reduction to ammonium (DNRA) under oxic conditions in a periodically hypoxic estuary[J]. Geochimica Et Cosmochimica Acta, 133(6): 313–324.
|
Robertson T R, Tissue Z D T. 2010. Precipitation magnitude and timing differentially affect species richness and plant density in the sotol grassland of the Chihuahuan Desert[J]. Oecologia, 162(1): 185–197.
DOI:10.1007/s00442-009-1449-z
|
Rooks C, Schmid M C, Mehsana W, et al. 2012. The depth-specific significance and relative abundance of anaerobic ammonium-oxidizing bacteria in estuarine sediments (Medway Estuary, UK)[J]. Fems Microbiology Ecology, 80(1): 19–29.
DOI:10.1111/j.1574-6941.2011.01266.x
|
Rütting T, Huygens D, Müller C, et al. 2008. Functional role of DNRA and nitrite reduction in a pristine south Chilean Nothofagus forest[J]. Biogeochemistry, 90(3): 243–258.
DOI:10.1007/s10533-008-9250-3
|
Schloss P D, John Q. 2009. A high-throughput DNA sequence aligner for microbial ecology studies[J]. PLoS ONE, 4(12): e8230.
DOI:10.1371/journal.pone.0008230
|
Wang S Y, Wang W, Liu L, et al. 2018. Microbial nitrogen cycle hotspots in the plant-bed/ditch system of a constructed wetland with N2O mitigation[J]. Environmental Science & Technology, 52(11): 6226–6236.
|
Sher Y, Schneider K, Schwermer C U, et al. 2008. Sulfide-induced nitrate reduction in the sludge of an anaerobic digester of a zero-discharge recirculating mariculture system[J]. Water Research, 42(16): 4386–4392.
DOI:10.1016/j.watres.2008.07.031
|
Song B, Lisa J A, Tobias C R. 2014. Linking DNRA community structure and activity in a shallow lagoonal estuarine system[J]. Frontiers in Microbiology, 5: 460.
|
Steenkamp D J, Peck H D. 1981. Proton translocation associated with nitrite respiration in Desulfovibrio desulfuricans[J]. The Journal of Biological Chemistry, 256(11): 5450–5458.
|
Thomas S H, Wagner R D, Arakaki A K, et al. 2008. The mosaic genome of anaeromyxobacter dehalogenans strain 2CP-C suggests an aerobic common ancestor to the delta-proteobacteria[J]. PLOS ONE, 3(5): 2103.
DOI:10.1371/journal.pone.0002103
|
Tomaszek J A, Rokosz G R. 2007. Rates of dissimilatory nitrate reduction to ammonium in two Polish reservoirs: impacts of temperature, organic matter content, and nitrate concentration[J]. Environmental Technology, 28(7): 771–778.
DOI:10.1080/09593332808618834
|
Wang W, Wang D, Yin C. 2010. A field study on the hydrochemistry of land/inland water ecotones with reed domination[J]. Clean-Soil Air Water, 30(2/3): 117–127.
|
Wang W, Liu W, Wu D, et al. 2018. Differentiation of nitrogen and microbial community in the littoral and limnetic sediments of a large shallow eutrophic lake (Chaohu Lake, China)[J]. Journal of Soils and Sediments: 1–12.
|
Welsh A, Chee-Sanford J C, Connor L M, et al. 2014. Refined nrfA phylogeny improves PCR-based nrfA gene detection[J]. Applied & Environmental Microbiology, 80(7): 2110–2119.
|
Yang Y, Rao S, Hu H, et al. 2004. Plant species richness of alpine grasslands in relation to environmental factors and biomass on the Tibetan Plateau[J]. Chinese Biodiversity, 12(1): 200–205.
|
张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理(汉文版), 2007, 30(3): 137–141.
|
Zhou J M, Song Z Y, Yan D J, et al. 2014. Performance of a haloalkaliphilic bioreactor under different, NO3-/SO42- math Container Loading Mathjax, ratios[J]. Bioresource Technology, 153(1): 216–222.
|
Zhu G, Wang S, Wang Y, et al. 2011. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. Isme Journal, 5(12): 1905–1912.
DOI:10.1038/ismej.2011.63
|
Zhu G, Xia C, Wang S, et al. 2015. Occurrence, activity and contribution of anammox in some freshwater extreme environments[J]. Environmental Microbiology Reports, 7(6): 961–969.
DOI:10.1111/1758-2229.12341
|
庄林杰, 夏超, 田晴, 等. 2017. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 2017, 37(1): 261–271.
|
 2019, Vol. 39
2019, Vol. 39


