2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environment Sciences, Chinese Academy of Sciences, Beijing 100085
氮以+5价至-3价之间的多种物质形式参与生物地球化学循环(Stein et al., 2016).由微生物驱动的氮循环过程涉及许多氧化还原反应, 其中, 反硝化过程将NO3-还原成N2或N2O, 硝酸盐异化还原成铵(Dissimilatory nitrate reduction to ammonium, DNRA)的过程以NO3-/NO2-为电子受体, 氧化NADH产生NH4+, 该过程中产生的NH4+一部分与NO2-进行厌氧氨氧化反应, 另一部分在硝化反应功能微生物作用下重新生成NO2-和NO3-, 新生成的NO2-和NO3-又再次参与反硝化、DNRA和厌氧氨氧化反应, 生成NO2-、NH4+和N2.研究表明, DNRA过程是微生物氮循环的关键过程之一(Steenkamp et al., 1981).
水稻是重要的粮食作物, 随着稻田氮肥施用量的快速增加, 在水稻产量增加的同时也导致了严重的氮损失, 且主要通过NH3挥发、N2O排放和NO3-淋失等途径而损失掉(Xing et al., 2000), 并对水生态安全、全球气候等造成了严重威胁.稻田中, 反硝化过程将NO3-还原成N2不仅造成氮损失, 还会产生具有温室效应的中间产物N2O(Zhu et al., 2011).DNRA过程可将土壤中的NO3-/NO2-还原成NH4+-N而被作物吸收利用, 有利于稻田中氮的保留及环境保护.
参与反硝化作用的细菌通常是呼吸细菌, 该细菌利用NO3-作为呼吸链中的末端电子受体(Zumft, 1997);而参与DNRA过程的细菌是发酵型细菌, 主要利用NO3-作为电子吸收体(Cole et al., 1980;Cole, 1990).早在1938年Woods对Clostridium welchii(韦氏梭菌)进行纯培养时发现了DNRA过程(Woods et al., 1938).据报道可知, 能够进行DNRA的细菌包括专性厌氧菌(如梭菌)、兼性厌氧菌(如肠杆菌)和好氧菌(如芽孢杆菌)(Tiedje, 1988).土壤中反硝化作用和DNRA之间的硝酸盐分配不仅取决于菌群, 还取决于环境条件.尽管对该过程的微生物学和生态学还没有很好的了解, 但强烈的还原状态和丰富的含碳量一直被认为是对DNRA有利的环境条件(Buresh et al., 1978;Tiedje et al., 1983;Tiedje, 1988).在无外加碳源的条件下进行的缺氧培养实验中, 通常只有少量添加的NO3-(通常少于5%)被转化为土壤中的NH4+或有机氮.例如, Buresh和Patrick报道, 在风干且厌氧性预培养的土壤中, 0.9%~1.8%的NO3-以NH4+的形式保留(Buresh et al., 1978).其他研究人员也报道了类似的数据(Caskey et al., 1979;DeCatanzaro et al., 1987;Yin et al., 1998).
通常认为, 有利于DNRA的天然土壤不是很常见, 反硝化通常是细菌NO3-异化还原的唯一途径.因此, DNRA似乎没有什么农业意义.但有证据表明, 在无外加碳源的土壤中, DNRA可能大量存在(Chen et al., 1995a;1995b).在不同的陆地生态系统(如森林、湿地、草地和农田)土壤中, DNRA在总NO3-的消耗方面占比范围广泛(0~119.8%)(Rütting et al., 2011).据报道, DNRA在农业稻田土壤中占总NO3-消耗的14.9%(Yin et al., 2002), 占15NO3-添加量的3.88%~25.40%(Lu et al., 2012).在特定条件下, DNRA过程可能在稻田土壤中发挥着重要作用, 这与DNRA似乎没有什么农业意义的结论相矛盾, 因此, 需要进一步了解稻田土壤中DNRA的影响因素.目前, 人们对DNRA细菌在稻田垂向土壤中的研究尚且不足.鉴于此, 本研究选择位于广东韶关市和吉林安图县的两块稻田作为研究地点, 通过扩增、测序等生物学技术, 深入研究两种稻田土壤中DNRA细菌的丰度、微生物群落组成及其各自的关键菌属, 并探究稻田土壤中DNRA细菌群落结构的垂向分布规律及其与环境因子的关系.
2 材料和方法(Materials and methods) 2.1 样品采集与保存本研究选择位于广东韶关市(24°33′37"N, 113°31′0"E)和吉林安图县(42°56′50"N, 128°28′48"E)的两块稻田为采样地点, 采样时间为2018年7月, 采样深度为1 m, 每10 cm采取1个土壤层样品, 共计20个土壤样品.将样品按其对应采样深度进行排序, 样品标号列于表 1, 其中, S1~S10为韶关稻田土壤样品, A1~A10为安图稻田土壤样品.在样品对应深度的土壤层随机采取3个土壤样品, 去除植物残体等杂质物后充分混匀, 混匀后的3个土壤样品一起作为一个土壤样品, 该样品代表对应土壤层土壤样品的平均水平(Mroz et al., 1991;Zhang et al., 2012;Zhu et al., 2015;Su et al., 2017).采取的土壤样品用无菌自封袋密封保存后放入冰盒储存并运回实验室.样品到达实验室后一部分放至4 ℃冰箱储存, 并立即进行各项理化指标的测定;一部分保存在-80 ℃超低温冰箱, 经冷冻干燥后用于DNA提取和后续的分子生物实验.
| 表 1 稻田土壤样品编号信息 Table 1 Soil sample labelling information |
用2 mol·L-1 KCl溶液将土样浸提1 h, 然后用0.45 μm微孔滤膜过滤, 得到其上清液, 用连续流动分析仪(SAN plus, Skalar Analytical B.V., Breda, the Netherlands)测定其氨氮(NH4+-N)、亚硝酸盐氮(NO2--N)和硝酸盐氮(NO3--N)的浓度;超纯水与土样按水土质量比为5:1充分混匀后静置30 min, 采用电极法测定样品pH;根据样品在105 ℃下的失水量来计算样品的含水率(MC), 然后根据样品在550 ℃下的烧失量来计算样品中的总有机质(TOM)(鲍士旦, 2000;张文河等, 2007);冷冻干燥的土样经研磨过筛后, 利用元素分析仪(Elementar Vario EL Ⅲ analyzer, 德国Elementar公司)对总氮(TN)、总碳(TC)和总硫(TS)的含量进行测定.所有理化指标的测定都有3个重复, 结果取平均值.
2.3 DNA提取、聚合酶链式反应(PCR)、荧光定量PCR扩增及高通量测序称取约0.33 g冷冻干燥的土壤样品, 按照FastDNA Spin Kit for Soil DNA提取试剂盒(安诺伦生物科技有限公司, 美国)的说明书进行样品总DNA的提取, 然后对提取的DNA进行检测, 判断其扩增条带的单一性和条带长度的正确性, 并测定其DNA浓度.然后对样品DNA进行PCR扩增, 实验中使用的特异性引物是nrfA-F2aw (5′-CARTGYCAYGTBGARTA-3′)和nrfA-R1(5′-TWNGGCATRTGRCARTC-3′).扩增体系(50 μL)为:10×buffer 5 μL, 2.5 μmol·L-1 dNTP 4 μL, ExTaq DNA聚合酶1 μL, 10 μmol·L-1 nrfA-F2aw和10 μmol·L-1 nrfA-R1各2 μL, DNA模板1 μL, 加ddH2O补足至50 μL(Welsh et al., 2014;Zhu et al., 2015).扩增条件为:95 ℃预变性5 min;94 ℃变性0.5 min, 52 ℃退火0.5 min, 72 ℃延伸20 s, 40个循环;72 ℃延伸10 min.
定量PCR反应体系为25 μL, 体系如下:SYBR qPCR Mix 12.5 μL, 10 μmol·L-1 nrfA-F2aw和10 μmol·L-1 nrfA-R1各1 μL, 50×Rox 0.5 μL, DNA模板5 μL, 加ddH2O补足至25 μL.使用ABI 7500实时定量扩增PCR仪(应用生物系统, 加利福尼亚州, 美国)进行荧光定量PCR扩增反应.定量PCR的程序:95 ℃预变性5 min;94 ℃变性30 s, 52 ℃退火30 s, 72 ℃延伸20 s, 40个循环.其中, 反应条件只适宜小样本量的操作, 反应设置阴性对照(以dd H2O代替DNA), 所有样品反应设置3个重复, 利用7500 SDS System配套软件进行分析计算, 通过标准曲线求得DNRA细菌的丰度值.阴性对照组定量结果须为未检测到(ND), 标准曲线扩增效率要求介于85%~110%之间, 且标准曲线可决系数大于0.995, 标准曲线和样品的溶解曲线为单一峰.
本研究中根据DNRA细菌丰度的定量结果(图 2), 选取每个稻田中6个代表土样, 共计12个样品(韶关稻田样品:S1、S2、S3、S4、S7、S10;安图稻田样品:A1、A2、A3、A5、A8、A10), 在Illumina MiSeq PE250平台对样品DNRA过程的功能基因nrfA进行高通量测序(百迈客生物科技有限公司, 北京).
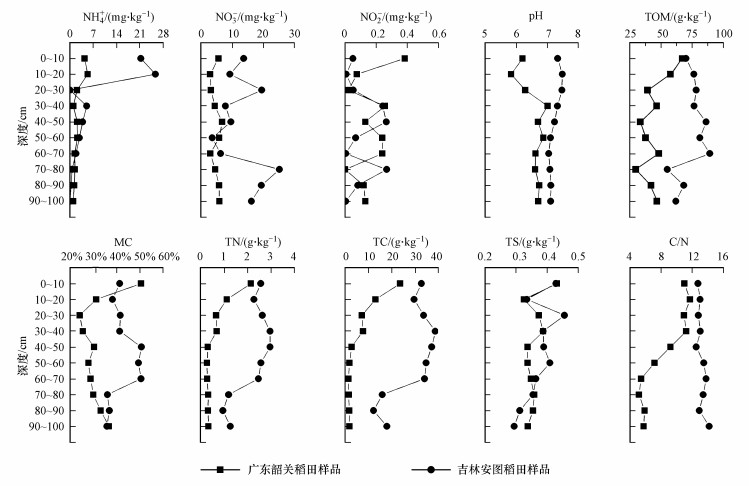 |
| 图 1 稻田土壤样品理化指标 Fig. 1 Physicochemical characteristics of the paddy soil samples |
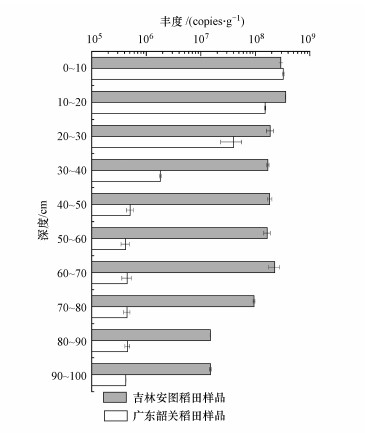 |
| 图 2 稻田土壤样品DNRA细菌的丰度 Fig. 2 Abundance of DNRA bacteria in soil samples of paddy soils |
每个土壤样品测序返回后的序列使用Qiime软件(Caporaso et al., 2010)过滤掉低质量的序列和短序列, 得到高质量的序列;之后利用Uchime软件(Edgar et al., 2011)去除PCR过程中不完整扩增导致的嵌合体序列;使用Mothur软件和Bioedit软件得到非冗余的核酸序列并进行翻译, 最终得到氨基酸序列, 并根据90%的相似度将序列进行聚类分析得到OTU的分布结果(Schloss et al., 2009;Edgar, 2010).选取占稻田样品序列总数60%的OTU作为代表OTU(Lundberg et al., 2012), 把OTU的代表氨基酸序列与Non-redundant protein sequrnces(nr)数据库中nrfA基因的氨基酸序列进行BLAST(Basic Local Alignment Search Tool)比对注释, 序列比对使用网页版BLAST, 将Uncultured/environmental sample排除掉, 采用的算法为blastp(protein-protein BLAST)(Kent, 2002;庄林杰等, 2017).
土壤样品群落结构分析:根据Bray-Cruits差异度指数, 用UPGMA算法进行群落分类, 用主坐标分析的方法(Principle Coordinate Analysis, PCoA)进行群落排序, 做出PcoA图, 从而进行样品间DNRA群落结构的差异性分析;通过Perl脚本及Mothur软件对土壤样品进行α-多样性和β-多样性分析(庄林杰等, 2017);基于R语言的psych软件包, 通过Spearman相关系数, 采用pairwise分析, 对样品OTU数进行相关性分析, 将r>0.8和p < 0.05的物种间的相互作用关系用网络图的形式展示, 分析DNRA群落结构的关键菌属;利用去趋势对应分析(DCA)和冗余分析(RDA)探究环境因素与DNRA群落结构的关系.通过SPSS 21.0软件进行相关性分析, 均采用Spearman相关系数;利用Origin9.0作图.
3 结果(Results) 3.1 稻田垂向(0~1 m)土壤样品理化性质由图 1可知, 韶关稻田和安图稻田土壤的NH4+含量分别为0.99~5.37 mg·kg-1和0.02~25.69 mg·kg-1, 最高值均出现在10~20 cm土层;土壤的NO3-含量分别为3.66~25.28 mg·kg-1和2.99~6.87 mg·kg-1, NO2-含量分别为0~0.38 mg·kg-1和0.01~0.27 mg·kg-1;含水率分别为24.20%~36.77%和35.92%~50.54%.韶关稻田土壤TC、TN和TS的最大值均出现在0~10 cm土层, pH为5.84~7.02, 土壤呈弱酸性;安图稻田土壤TC、TN的最大值均出现在30~40 cm土层, TS的最大值出现在20~30 cm土层, pH为7.05~7.49, 土壤呈弱碱性.安图稻田样品C/N在12.56~14.22之间, 韶关稻田样品C/N在5.17~11.75之间.
3.2 DNRA细菌的nrfA基因的丰度样品DNRA细菌nrfA基因丰度如图 2所示.由图可知, 安图稻田样品DNRA细菌的丰度值整体显著高于韶关稻田样品(p=0.002, n1=n2=10), 其中, 韶关稻田样品丰度最高值为(3.25±0.12)×108 copies·g-1, 出现在0~10 cm土层, 安图稻田样品丰度最高值为(3.60±0.05)×108 copies·g-1, 出现在10~20 cm土层;两个稻田样品DNRA细菌丰度最低值均出现在深层土壤层(90~100 cm), 韶关稻田和安图稻田土壤样品DNRA细菌丰度最低值分别为(4.22±0.04)×105 copies·g-1和(1.51±0.06)×107 copies·g-1.韶关稻田不同层次土壤样品之间的DNRA细菌丰度值存在明显差异, 且这种差异随土壤深度增加而降低;而安图稻田在0~70 cm深度之间土壤样品的DNRA细菌丰度值无明显差异, 从70 cm处丰度开始递减.
将DNRA细菌的丰度与土壤理化因子使用SPSS软件进行相关性分析, 结果列于表 2.由表可知, 与DNRA细菌丰度显著正相关的理化因子为:NH4+(r=0.484, p=0.031)、pH(r=0.476, p=0.034)、TS(r=0.486, p=0.030)、TOM(r=0.786, p=0.000)、MC(r=-0.734, p=0.000)、TN(r=0.843, p=0.000)、TC(r=0.838, p=0.000)、C/N(r=0.642, p=0.002);与DNRA细菌丰度显著负相关的因子为NO3-(r=-0.474, p=0.035);NO2-与DNRA细菌丰度没有相关性.
| 表 2 稻田样品中DNRA细菌丰度与理化因子的相关性分析(n=20) Table 2 Correlation analysis between abundance of DNRA bacteria and physicochemical characteristics of paddy soils |
将土壤样品得到的OTU数量和覆盖率指数等信息列于表 3.由表可知, 质控处理后每个稻田土壤样品得到60000条高质量序列, 样品的OTU数量在274~4630之间, 其中, OTU数量最多的是样品A1, OTU数量最少的是样品S10, 安图稻田样品的OTU数量显著高于韶关稻田样品的OTU数量(p=0.027, n1=n2=6), 测序结果覆盖率均在97%以上, 表示样品取样充足, 测序数据可靠.
| 表 3 高通量测序样品描述 Table 3 Summary of high-throughput sequencing results |
对稻田样品进行α-多样性分析, 将各样品的丰富度指数(Chao1指数)、优势度指数(Simpson指数)和多样性指数(Shannon指数)列于表 4.由表可知, Chao1指数和Shannon指数最高的样品均是A2, 分别为6134.308、6.849;Chao1指数和Shannon指数最低的样品均是S10, 分别为353.000和3.907;Simpson指数最高的是样品S10(0.029), 最低的是样品S2和A2, 均为0.004.由此可得两个稻田样品中, 安图稻田亚表层(10~20 cm)样品A2的物种多样性最高, 韶关底层样品S10的物种多样性最低.把两个稻田相同深度的土壤样品之间进行两两对比, 发现Chao1指数和Shannon指数均是安图稻田样品大于韶关稻田样品, 而Simpson指数则为安图稻田样品均小于韶关稻田样品, 由此可得安图稻田样品的DNRA细菌的物种多样性大于韶关稻田样品.比较同一稻田各样品的α-多样性指数, 发现深度相差较大的土壤层间的多样性指数差异较大, 而相邻土壤层样品间多样性指数差异较小, 10~20 cm土壤层样品的Chao1指数和Shannon指数均大于0~10 cm土壤层样品, Simpson指数均小于0~10 cm土壤层样品, 可得两个稻田中均是亚表层10~20 cm土壤样品的物种多样性大于表层0~10 cm土壤样品.
| 表 4 土壤样品DNRA细菌的α-多样性指数 Table 4 The α-diversity index of DNRA bacteria in soil samples of paddy soils |
对稻田样品进行β-多样性分析, 做出PCoA图, 如图 3所示, 关于样点间群落差异受到环境的影响, 各排序轴的解释度较低, 两轴对排序结果的解释度分别是32.1%和14.8%.图中韶关稻田样品S1和S2聚在一起, S3、S4、S7聚在一起, 而深度相差较大的S10与其它样品之间距离较远, 表明样品S1和S2之间, S3、S4和S7之间DNRA细菌的群落结构更相似, 说明随着土壤深度的增加韶关稻田样品之间群落结构出现差异, 且临近土壤层之间群落结构更相似.安图稻田所有土壤样品A1、A2、A3、A5、A8、A10聚在一起, 距离很近, 而与韶关稻田样品之间距离都很远, 表明安图稻田样品之间的群落结构相似, 与韶关稻田样品之间菌群结构差异度很大.使用PERMANOVA统计分析对PCoA图聚类结果进行检验, 结果显示, 与PCoA结果一致, 韶关稻田和安图稻田两个采样点的土壤样品细菌群落结构具有显著差异(p < 0.05).两个稻田样品间的群落结构体现出明显的空间异质性.
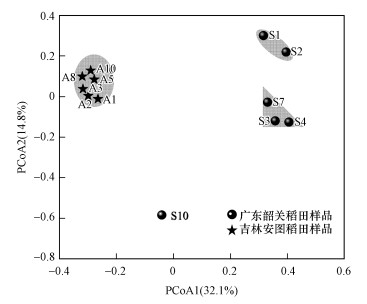 |
| 图 3 稻田土壤样品DNRA细菌的PCoA图 Fig. 3 Principal coordinates analysis of DNRA bacteria in paddy soil samples |
选取占稻田样品序列总数60%的前288个OTU作为代表OTU(Lundberg et al., 2012).通过序列比对和注释, 按照门分类, 样品的OTUs注释到了变形菌门Proteobacteria(40.00%)、热脱硫杆菌门Thermodesulfobacteria(19.49%)、硝化螺旋菌门Nitrospirae(10.90%)等12个门, 样品共注释到了Anaeromyxobacter等36个属, 其中, 相对丰度前三的Anaeromyxobacter、Caldimicrobium和Nitrospira分别占28.66%、19.49%和10.90%, Chthoniobacter占9.15%, Caldilinea占5.06%, 其他菌属的占比不足5%.在属水平上做出相对丰度图, 由图 4可知, 安图稻田样品之间群落结构差异较小, 韶关稻田样品之间群落结构差异较大, 12个土壤样品(除去S1、S7)的群落结构体现出一定的规律性, 均是属于变形菌门Proteobacteria的Anaeromyxobacter菌属相对丰度最高, 属于热脱硫杆菌门Thermodesulfobacteria的Caldimicrobium菌属次之, 其中, Anaeromyxobacter菌属在样品S3中占比最高(49.7%), 在样品S7中占比最低(12.1%), 样品S1和样品S7均以Caldimicrobium菌属为主, Anaeromyxobacter菌属次之, 变形菌门Proteobacteria和热脱硫杆菌门Thermodesulfobacteria是两个稻田DNRA细菌中优势最明显的两个门, 二者相对丰度之和占DNRA细菌的59.5%.Chthoniobacter菌属呈现出随土壤深度增加相对丰度增加的趋势, 并且其在安图稻田中的相对丰度(6.7%~21.9%)高于在韶关稻田中的相对丰度(0.8%~8.5%).
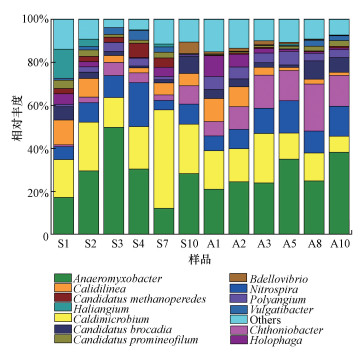 |
| 图 4 稻田土壤样品DNRA细菌的相对丰度 Fig. 4 Relative abundance of DNRA bacteria in the paddy soil samples |
稻田样品在属水平上的网络图如图 5所示, 由图可知, 韶关稻田样品和安图稻田样品的物种大体都分为4个模块, 其它模块占比较少, 不足8%.图 5a的韶关稻田网络图中共有133个节点(物种), 1312条边组成;图 5b的安图稻田网络图中共有138个节点(物种), 1477条边组成.综合考虑degree、closeness centrality、betweenness centrality等网络拓扑结构得出:韶关稻田中占比最大的模块ModuleⅠ(19%)及其它几个重要模块ModuleⅡ(13%)、ModuleⅢ(12%)、ModuleⅣ(12%)中的关键菌属分别是Anaeromyxobacte、Caldimicrobium、Chthoniobacter、Haliangium;安图稻田中占比最大的ModuleⅠ(32%)内的关键菌属是Caldimicrobium, 模块ModuleⅡ(17%)和ModuleⅢ(12%)内的关键菌属均是Anaeromyxobacter, ModuleⅣ(12%)内的关键菌属是Nitrospira.
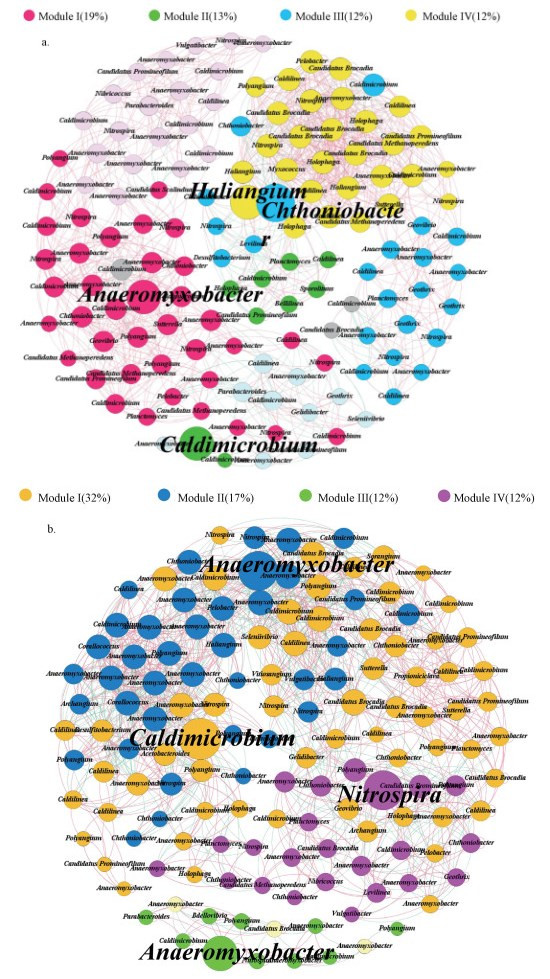 |
| 图 5 稻田土壤样品DNRA细菌分子生态网络分析 (a.广东韶关稻田,b.吉林安图稻田) Fig. 5 Molecular ecological network analysis of DNRA bacteria in soil samples of paddy soils |
综上, 韶关稻田关键菌属主要是Anaeromyxobacter、Caldimicrobium、Chthoniobacter、Haliangium, 而安图稻田关键菌属主要是Anaeromyxobacter、Caldimicrobium、Nitrospira, 处于不同地理环境的两个稻田中关键物种有所不同.关键物种的消亡会给群落结构及其功能带来巨大的改变, 其在微生物群落中有着独特的地位(Banerjee et al., 2011).
3.6 DNRA细菌的群落结构与土壤理化因子的相关性分析使用SPSS软件进行关键菌属的丰度与土壤理化因子之间的相关性分析(表 5)和冗余分析(RDA)(图 6), 探究DNRA细菌与理化因子的关系.
| 表 5 稻田土壤中DNRA关键菌属与环境因子相关性分析(n=12) Table 5 Correlation analysis between DNRA bacteria and physicochemical characteristics of paddy soils |
 |
| 图 6 DNRA细菌与理化因子的RDA分析(n=12) Fig. 6 RDA analysis between DNRA bacteria and physicochemical characteristics |
由相关性分析得出:Caldimicrobium属与C/N呈显著负相关(r=-0.727, p=0.007);Chthoniobacter属与NH4+呈显著负相关(r=-0.601, p=0.039), 与pH呈显著正相关(r=0.615, p =0.033).Polyangium属与pH呈显著正相关(r=0.622, p=0.031), 与TN呈显著负相关(r=-0.580, p=0.048), 与TC呈显著负相关(r=-0.580, p=0.048);Caldimicrobium属与TOM呈显著负相关(r=-0.727, p=0.007).由RDA分析得出理化因子的解释度并进行解释度排序, 得出影响DNRA群落结构的关键环境限制因子为TC.
4 讨论(Discussion)南方如韶关等地, 以红壤土为主, 土壤淋溶严重, 相对贫瘠, 有机质含量较低;安图稻田以黑土为主, 土壤肥沃, 有机质含量高, 因地处寒温带, 土面蒸发小, 土壤持水能力强; 而韶关稻田地处热带亚热带, 土面蒸发及植物蒸腾作用强, 土壤持水能力弱, 灌溉量大且频率高.土壤本身发育和耕种灌溉等活动强度的不同, 以及气候的叠加影响造成土壤性质及微生物等过程差异, 致使两个稻田样品间DNRA细菌群落结构呈现出明显的空间异质性.
水稻同化作用固定的有机碳随植物的死亡、茎叶的凋落腐烂而储存于土壤表层有机碳中, 以及稻田的施肥和含NO3-水的灌溉, 使稻田表层土壤中含有丰富的无机氮源和有机碳源(Patra et al., 2006;Guo et al., 2013).研究发现, 在C/N低、碳源不足、NO3-含量高的条件下可以促进异养反硝化的进行;而在氮源缺乏、碳源充足、高C/N的条件下DNRA过程容易发生, 高C/N环境条件对DNRA过程是有利的(傅利剑等, 2005).土壤表层较高的有机质含量、丰富的碳源是DNRA细菌更多的存在于稻田表层(0 ~20 cm)土壤中的环境条件.安图稻田土壤样品C/N在12.56~14.22之间, 韶关稻田土壤样品C/N在5.17~11.75之间, 较高的C/N是安图稻田土壤中DNRA细菌丰度、菌群多样性较韶关稻田土壤偏高的一个重要因素.
由相关性分析和RDA分析可知, 稻田土壤样品含水率与样品DNRA细菌的丰度呈显著正相关, 且属于主要的影响因子.暴雨过后施撒肥料可以增加有机质矿化而消耗土壤中的O2, 进而抑制土壤微生物的硝化作用, 不利于产NO3-(Ball et al., 2004);水分的增加可降低土壤中的氧含量, 与高溶解氧和厌氧条件下相比较, DNRA过程在微氧环境中更活跃(刘佳, 2007);土壤中一定的含水率对细胞内部物质的传递及细菌的生长等生命活动都是有益的, 可以加快细胞在土壤中的移动速度, 提高细胞中水分含量, 为细胞内物质传递提供良好的介质(周利等, 2019).DNRA过程对温度的要求较高, 韦宗敏(2012)在对珠江口沉积物的研究中发现DNRA的最佳温度是30 ℃;并且温度的变化也会影响DNRA细菌的群落结构及活性(Ogilvie et al., 1997;Tomaszek et al., 2007).两个稻田样品采样时间均是在夏季7月, 环境温度差异不大, 温度不是影响其群落结构差异的关键性因素;环境因子pH和DNRA过程相互影响, DNRA多发生于偏碱性的环境中, 并且DNRA过程中产碱(陶怡乐等, 2016), 实验测得安图稻田的pH为7.05~7.49, 土壤偏碱性, 而韶关稻田的pH为5.84~7.02, 土壤偏酸性, 这也是安图稻田样品中DNRA细菌丰度、菌群多样性大于韶关稻田样品的一个重要环境因子.
稻田样品中DNRA细菌占比最高的是厌氧粘细菌(Anaeromyxobacter), Anaeromyxobacter在每个土壤样品中相对丰度都特别高, 是一种多功能的变形细菌, 分布在不同类型的土壤和沉积物环境中(Thomas et al., 2008).Anaeromyxobacter缺乏标志性脱氮基因nirS和nirK(编码NO2-→NO还原酶), 通过把NO3-还原成NH4+(DNRA过程)或者把N2O还原成N2的过程获得生长所需的能量, Anaeromyxobacter也通过将Fe(Ⅲ)还原为Fe(Ⅱ)而生长, 过程中与NO2-发生化学反应生成N2O, 即Anaeromyxobacter耦合土壤中异化铁还原过程, 可把NO2-通过化学反应生成N2O, 进而产生N2(即生物化学脱氮).考虑到土壤和沉积物中铁的普遍存在及异化铁还原过程和NO3-还原剂的广泛分布, Anaeromyxobacter可能对稻田中的N损失和N2O通量有很大贡献, 不利于土壤中氮素的保存(Onley et al., 2018).
沉积物中H2S、S2-和S单质等硫化物可作为电子供体为DNRA反应提供电子, 对DNRA的发生有促进作用(Sher et al., 2008;Lu et al., 2013).以NO3-为电子受体, 自养硫细菌氧化H2S、S2-及S单质以达到去除硝酸盐的目的, 是一种特殊的自养DNRA方式(Sayama et al., 1983).样品中丰度占比第二的关键菌属是Caldimicrobium, Caldimicrobium属于热脱硫杆菌门(Thermodesulfobacteria), 而Thermodesulfobacteria是一个小类群的嗜热硫酸盐还原细菌, 属于厌氧微生物.研究表明, 硫酸盐还原菌在无氧或极少氧条件下将硫酸盐还原成H2S而获得生存的能量, 过程中的产物H2S为自养类型的DNRA反应提供了反应底物.由RDA分析发现, 稻田土壤层样品中DNRA细菌丰度和TS显著正相关, 推测稻田中进行着自养类型的DNRA过程, 且较高的硫化物含量可能使硫细菌在稻田垂向土壤中发挥了较大的DNRA作用.
稻田土壤中丰度占比第三的关键菌属硝化螺菌属(Nitrospira)主要参与亚硝酸盐氧化反应, 常被定义为NOB(Nitrite Oxidizing Bacteria).Nitrospira在土壤中大量存在并进行着硝化作用的同时也具有DNRA功能, 在林地、草地和农田土壤生态系统中主要的NOB群落分为Nitrospira(硝化螺菌属)和Nitrobacter(硝化杆菌属)(Wang et al., 2015), 并且Nitrospira种群的多样性高于Nitrobacter(Freitag et al., 2005).Nitrospira菌属中多个物种包含有nrfA和反硝化功能基因, 如菌株Nitrospira ND1的基因组序列中包含了亚硝酸盐还原酶nrfA的编码(Ushiki et al., 2017).
5 结论(Conclusions)本研究在一定程度上揭示了稻田土壤中DNRA细菌群落结构的垂向分布规律及其与环境因子的关系, 在韶关和安图稻田土壤中, DNRA细菌更多的存在于表层土壤中, 且表层样品群落结构的多样性大于深层样品, Anaeromyxobacter、Caldimicrobium、Nitrospira和Chthoniobacter是两个稻田中DNRA细菌群落组成中的关键菌属.偏碱性、有机质丰富、氮源缺乏、碳源丰富且C/N较高的环境及适宜含水率是稻田垂向生态系统中DNRA细菌适宜的生存环境, 应该结合土壤微生物群落对环境因子的响应机制来探索稻田的氮素保存, 降低稻田施肥量从而减少环境污染.
Ball B C, McTaggart I P, Scott A. 2004. Mitigation of greenhouse gas emissions from soil under silage production by use of organic manures or slow-release fertilizer[J]. Soil Use and Management, 20(3): 287-295. DOI:10.1079/SUM2004257 |
Banerjee S, Schlaeppi K, van der Heijden M G A. 2018. Keystone taxa as drivers of microbiome structure and functioning[J]. Nature Reviews Microbiology, 16(9): 567. DOI:10.1038/s41579-018-0024-1 |
鲍士旦. 2000. 土壤农化分析(第3版)[M]. 北京: 中国农业出版社.
|
Buresh R J, Patrick W H. 1978. Nitrate reduction to ammonium in anaerobic soil[J]. Soil Science Society of America Journal, 42(6): 913-918. DOI:10.2136/sssaj1978.03615995004200060017x |
Caporaso J G, Kuczynski J, Stombaugh J. 2010. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
Caskey W H, Tiedje J M. 1979. Evidence for clostridia as agents of dissimilatory reduction of nitrate to ammonium in soils[J]. Soil Science Society of America Journal, 43(5): 931-936. DOI:10.2136/sssaj1979.03615995004300050023x |
Chen D L, Chalk P M, Freney J R. 1995a. Distribution of reduced products of 15N-labelled nitrate in anaerobic soils[J]. Soil Biology & Biochemistry, 27(12): 1539-1545. |
Chen D L, Chalk P M, Freney J R, et al. 1995b. Estimation of nitrification rates in flooded soils[J]. Microbial Ecology, 30(3): 269. |
Cole J A, Brown C M. 1980. Nitrite reduction to ammonia by fermentative bacteria:A short circuit in the biological nitrogen cycle[J]. Fems Microbiology Letters, 7(2): 65-72. DOI:10.1111/j.1574-6941.1980.tb01578.x |
Cole J A. 1990. Physiology, Biochemistry and Genetics of Nitrate Dissimilation to Ammonia//Denitrification in Soil and Sediment[M]. Boston, MA: Springer, 57-76.
|
DeCatanzaro J B, Beauchamp E G, Drury C F. 1987. Denitrification vs dissimilatory nitrate reduction in soil with alfalfa, straw, glucose and sulfide treatments[J]. Soil Biology and Biochemistry, 19(5): 583-587. DOI:10.1016/0038-0717(87)90102-7 |
Edgar R C. 2010. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
Edgar R C, Haas B J, Clemente J C, et al. 2011. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 27(16): 2194-200. DOI:10.1093/bioinformatics/btr381 |
Freitag T E, Chang L, Clegg C D, et al. 2005. Influence of inorganic nitrogen management regime on the diversity of nitrite-oxidizing bacteria in agricultural grassland soils[J]. Applied&Environmental Microbiology, 71(12): 8323-8334. |
傅利剑, 郭丹钊, 史春龙, 等. 2005. 碳源及碳氮比对异养反硝化微生物异养反硝化作用的影响[J]. 生态与农村环境学报, 21(2): 42-45. DOI:10.3969/j.issn.1673-4831.2005.02.009 |
Guo G X, Deng H, Qiao M, et al. 2013. Effect of long-term wastewater irrigation on potential denitrification and denitrifying communities in soils at the watershed scale[J]. Environmental Science & Technology, 47(7): 3105-3113. |
Kelso B, Smith R V, Laughlin R J, et al. 1997. Dissimilatory nitrate reduction in anaerobic sediments leading to river nitrite accumulation[J]. FEMS Microbiology Ecology, 63(12): 4679-4685. |
Kent W J. 2002. BLAT-the BLAST-like alignment tool[J]. Genome Research, 12(4): 656-664. DOI:10.1101/gr.229202 |
刘佳.2007.微生物好氧硝酸盐还原产铵研究[D].成都: 四川大学
|
Lu W W, Zhang H L, Shi W M. 2013. Dissimilatory nitrate reduction to ammonium in an anaerobic agricultural soil as affected by glucose and free sulfide[J]. European Journal of Soil Biology, 58: 98-104. DOI:10.1016/j.ejsobi.2013.07.003 |
Lu W W, Riya S, Sheng Z, et al. 2012. In situ dissimilatory nitrate reduction to ammonium in a paddy soil fertilized with liquid cattle waste[J]. Pedosphere, 22(3): 314-321. DOI:10.1016/S1002-0160(12)60018-6 |
Lundberg D S, Lebeis S L, Paredes S H, et al. 2012. Defining the core Arabidopsis thaliana root microbiome[J]. Nature, 488(7409): 86. DOI:10.1038/nature11237 |
Mroz G D, Reed D D. 1991. Forest soil sampling efficiency:Matching laboratory analyses and field sampling procedures[J]. Soil Science Society of America Journal, 55(5): 1413-1416. DOI:10.2136/sssaj1991.03615995005500050035x |
Ogilvie B G, Rutter M, Nedwell D B. 1997. Selection by temperature of nitrate-reducing bacteria from estuarine sediments:species composition and competition for nitrate[J]. FEMS Microbiology Ecology, 23(1): 11-22. DOI:10.1111/j.1574-6941.1997.tb00386.x |
Onley J R, Ahsan S, Sanford R A, et al. 2018. Denitrification by Anaeromyxobacter dehalogenans, a common soil bacterium lacking the nitrite reductase genes nirS and nirK[J]. Applied Environmental Microbiology, 84(4): e01985-17. |
Patra A K, Abbadie L, Clays-Josserand A, et al. 2006. Effects of management regime and plant species on the enzyme activity and genetic structure of N-fixing, denitrifying and nitrifying bacterial communities in grassland soils[J]. Environmental Microbiology, 8(6): 1005-1016. DOI:10.1111/j.1462-2920.2006.00992.x |
Rütting T, Boeckx P, Müller C, et al. 2011. Assessment of the importance of dissimilatory nitrate reduction to ammonium for the terrestrial nitrogen cycle[J]. Biogeosciences, 8(7): 1779-1791. DOI:10.5194/bg-8-1779-2011 |
Sayama M, Risgaard-Petersen N, Nielsen L P, et al. 2005. Impact of bacterial NO3- transport on sediment biogeochemistry[J]. Applied and Environmental Microbiology, 71(11): 7575-7577. DOI:10.1128/AEM.71.11.7575-7577.2005 |
Schloss P D, John Q. 2009. A high-throughput DNA sequence aligner for microbial ecology studies[J]. PLoS ONE, 4(12): e8230. DOI:10.1371/journal.pone.0008230 |
Sher Y, Schneider K, Schwermer C U, et al. 2008. Sulfide-induced nitrate reduction in the sludge of an anaerobic digester of a zero-discharge recirculating mariculture system[J]. Water Research, 42(16): 4386-4392. DOI:10.1016/j.watres.2008.07.031 |
Steenkamp D J, Peck H D. 1981. Proton translocation associated with nitrite respiration in Desulfovibrio desulfuricans[J]. Journal of Biological Chemistry, 256(11): 5450-5458. |
Stein L Y, Klotz M G. 2016. The nitrogen cycle[J]. Current Biology, 26(3): R94-R98. DOI:10.1016/j.cub.2015.12.021 |
Su Y, Wang W, Wu D, et al. 2017. Stimulating ammonia oxidizing bacteria (AOB) activity drives the ammonium oxidation rate in a constructed wetland (CW)[J]. Science of the Total Environment, 624: 87. |
陶怡乐, 温东辉. 2016. 细菌硝酸盐异化还原成铵过程及其在河口生态系统中的潜在地位与影响[J]. 微生物学通报, 43(1): 172-181. |
Thomas S H, Wagner R D, Arakaki A K, et al. 2008. The mosaic genome of Anaeromyxobacter dehalogenans strain 2CP-C suggests an aerobic common ancestor to the delta-proteobacteria[J]. PloS One, 3(5): e2103. DOI:10.1371/journal.pone.0002103 |
Tiedje J M. 1988. Ecology of denitrification and dissimilatory nitrate reduction to ammonium[J]. Biology of Anaerobic Microorganisms, 717: 179-244. |
Tiedje J M, Sexstone A J, Myrold D D, et al. 1983. Denitrification:ecological niches, competition and survival[J]. Antonie van Leeuwenhoek, 48(6): 569-583. DOI:10.1007/BF00399542 |
Tomaszek J A, Rokosz G R. 2007. Rates of dissimilatory nitrate reduction to ammonium in two Polish reservoirs:impacts of temperature, organic matter content, and nitrate concentration[J]. Environmental Technology, 28(7): 771-778. DOI:10.1080/09593332808618834 |
Ushiki N, Fujitani H, Shimada Y, et al. 2017. Genomic analysis of two phylogenetically distinct nitrospira species reveals their genomic plasticity and functional diversity[J]. Frontiers in Microbiology, 8: 1-7. |
Wang B, Zhao J, Guo Z, et al. 2015. Differential contributions of ammonia oxidizers and nitrite oxidizers to nitrification in four paddy soils[J]. The ISME Journal, 9(5): 1062. DOI:10.1038/ismej.2014.194 |
韦宗敏.2012.微好氧环境中硝酸盐异化还原成铵的影响研究[D].广州: 华南理工大学 http://cdmd.cnki.com.cn/Article/CDMD-10561-1012450921.htm
|
Welsh A, Chee-Sanford J C, Connor L M, et al. 2014. Refined nrfA phylogeny improves PCR-based nrfA gene detection[J]. Applied & Environmental Microbiology, 80(7): 2110-2119. |
Woods D D. 1938. The reduction of nitrate to ammonia by Clostridium welchii[J]. Biochemical Journal, 32(11): 2000. DOI:10.1042/bj0322000 |
Xing G X, Zhu Z L. 2000. An assessment of N loss from agricultural fields to the environment in China[J]. Nutrient Cycling in Agroecosystems, 57(1): 67-73. DOI:10.1023/A:1009717603427 |
Yin S X, Chen D, Chen L M, et al. 2002. Dissimilatory nitrate reduction to ammonium and responsible microorganisms in two Chinese and Australian paddy soils[J]. Soil Biology and Biochemistry, 34(8): 1131-1137. DOI:10.1016/S0038-0717(02)00049-4 |
Yin S X, Shen Q, Tang Y, et al. 1998. Reduction of nitrate to ammonium in selected paddy soils of China[J]. Pedosphere, 8(3): 221-228. |
张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理(汉文版), 30(3): 137-141. |
Zhang L M, Hu H W, Shen J P, et al. 2012. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. Isme Journal Multidisciplinary Journal of Microbial Ecology, 6(5): 1032-1045. |
周利, 王晓侠, 周立光, 等. 2019. 旱地作物根际和非根际土壤硝酸盐异化还原成铵细菌群落组成的研究[J]. 环境科学学报, 39(2): 527-536. |
Zhu G, Wang S, Wang Y, et al. 2011. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. The ISME Journal, 5(12): 1905. DOI:10.1038/ismej.2011.63 |
Zhu G, Xia C, Wang S, et al. 2015. Occurrence, activity and contribution of anammox in some freshwater extreme environments[J]. Environmental Microbiology Reports, 7(6): 961-969. DOI:10.1111/1758-2229.12341 |
Zumft W G. 1997. Cell biology and molecular basis of denitrification[J]. Microbiol Mol Biol Rev, 61(4): 533-616. DOI:10.1128/.61.4.533-616.1997 |
庄林杰, 夏超, 田晴, 等. 2017. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 37(1): 261-271. |
 2020, Vol. 40
2020, Vol. 40


