2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environment Sciences, Chinese Academy of Sciences, Beijing 100085
人类活动严重地破坏了原有的氮循环平衡, 导致一系列严重的氮污染问题(Zhu et al., 2011).湖泊、河流等生态系统中的氮污染问题也已经成为人们关注的焦点(Tobias et al., 2003).氮循环在微生物驱动下, 经过一系列氧化还原反应转化成不同的含氮化合物(Jetten et al., 2009).好氧硝化是氮循环中的关键步骤, 通常被认为是一个两步过程.在两步硝化过程中, 氨首先被氨氧化微生物(AOM)包括氨氧化细菌(AOB)和氨氧化古菌(AOA)氧化成亚硝酸盐(Konneke et al., 2005), 然后亚硝酸盐被亚硝酸盐氧化细菌(NOB)进一步氧化成硝酸盐(Teske et al., 1994).然而, 最近发现了完全氨氧化菌(comammox)(van Kessel et al., 2015; Daims et al., 2015; Pinto et al., 2016)能够将氨完全氧化成硝酸盐的微生物.comammox的发现使人们对氮循环, 尤其是硝化过程有了新的认识.
1890年, 俄罗斯科学家Winogradsky提出, 硝化作用需要由两组不同的微生物分两步完成(Winogradsky, 1980).2006年, Costa等根据最理想代谢过程中的动力学理论(Heinrich et al., 1996; Pfeiffer et al., 2004)提出了不同观点, 认为氨全程被一种微生物氧化成硝酸盐是可行的, 而且这一过程在热力学上更容易实现(Costa et al., 2006).2015年, 3个科学团队(van Kessel et al., 2015; Daims et al., 2015; Pinto et al., 2016)分别发现了comammox以确凿的证据证明了Costa提出的理论.硝化作用是自然界中连接氧化态和还原态无机氮库的唯一生物学过程, 硝化微生物在维持全球氮素平衡中起着关键作用.comammox细菌作为自养型硝化微生物, 参与硝化过程输出的硝态氮可为反硝化作用提供底物, 因此了解完全氨氧化过程对环境氮循环有重要意义.
有关comammox的研究, 研究者们在公共数据库中挖掘发现在大气颗粒中(Gao et al., 2016)、污水处理厂(Chao et al., 2016; Gonzalez-Martinez et al., 2016; Annavajhala et al., 2018)、地下水(Palmo et al., 2016; Fowler, et al., 2018)、淡水循环养殖系统(Bartelme et al., 2017)和自来水生物膜(Pinto et al., 2016; Wang, 2017)等都有comammox存在.但是关于湖泊comammox的相关研究鲜有报道.巢湖是我国第五大淡水湖, 具有防洪、供水、灌溉、交通、渔业、旅游等多种功能, 在区域社会经济发展中发挥着极其重要的作用(Chen et al., 2011).近年来随着地表水水质的不断下降, 水源地水质也受到不同程度的污染, 将comammox细菌作为功能微生物, 或许可以研发一种新型的微污染水体脱氮工艺.对巢湖地区进行相关研究, 或将为改善其水质提供新思路.
因此, 本文选择巢湖岸边带和湖心区冬夏季的表层沉积物为研究对象, 采用高通量、定量PCR技术, 确定其表层沉积物中的comammox细菌的丰度、群落结构及其生物多样性, 并应用统计分析方法, 将与影响其分布的潜在环境因子联系起来以及评估comammox细菌、AOB和AOA的生态位分化.
2 材料和方法(Materials and methods) 2.1 样品采集与保存本研究采集巢湖岸边带和湖心区冬夏两季表层沉积物(0~10 cm)样品12个.其中夏季岸边带样品3个(SA3、SA5和SA6);冬季岸边带样品3个(WA3、WA5和WA6);夏季湖心区3个(SH4、SH9和SH10);冬季湖心区3个(WH4、WH9和WH10).每个采样点随机取3个表层沉积物样品, 去除植物残体和螺类动物残骸等杂质物后充分混匀.采集样品完毕, 用无菌自封袋密封保存后, 再用冰盒储存带回实验室.样品到达立即测其各项理化性质, 放至4 ℃冰箱储存, 剩下部分经冷冻干燥后保存在-80 ℃超低温冰箱用于DNA提取和后续的分子生物实验.
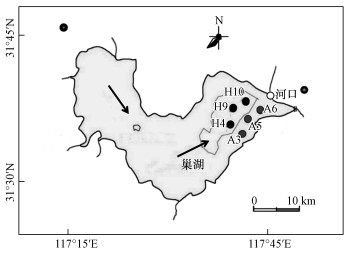 |
| 图 1 巢湖采样点分布图 Fig. 1 Location of Lake Chaohu and sampling sites |
按照标准检测方法《土壤农化分析》 (鲍士旦, 2000)进行相应理化因子的测定.总有机质(TOM)是以样品在550 ℃的烧失量计算(LOI550) (张文河等, 2007);向土样中按照土壤/水为1:5的质量比加入超纯水浸提1 h后测定其pH值;用KCl溶液(2 mol·L-1)对土壤样品进行浸提, 利用连续流动分析仪(SAN plus, Skalar Analytical B.V., Breda, the Netherlands)测定土壤氨氮(NH4+-N)、亚硝酸盐氮(NO2--N)和硝酸盐氮(NO3--N)的浓度;将风干的土壤样品过100目筛(0.149 mm)后利用元素分析仪对土壤总氮(TN)、总碳(TC)和总硫(TS)同时进行测定.样品所有理化性质都进行3次重复测定取平均值(表 1).文中与沉积物样品相关的单位都是冷冻干燥以后的重量.
| 表 1 巢湖冬夏季表层沉积物理化性质 Table 1 Physical properties of surface sediments in Chaohu Lake in winter and summer |
利用FastDNA Spin Kit for Soil DNA提取试剂盒(MP Biomedicals, USA), 参照说明书, 称取约0.33 g冷冻干燥处理后的土壤样品, 提取样品中的总DNA, 用1%琼脂糖凝胶对提取的DNA进行检测, 并用Nano Drop 2000 UV-Vis Spectrophotometer(Thermo Fisher Scientific, USA)测定其DNA浓度, 在260 nm和280 nm处的吸光度比值均为1.8左右, 表明获得了高质量的DNA.针对全程氨氧化微生物amoA功能基因, 采用特异性引物pmoA-189b-F (5′-GGNGACTGGGACTTYTGG-3′)和com-amoA-1-R (5′-CGAGATCATGGTGCTGTGAC-3′) (Luesken et al., 2011; Bartelme et al., 2017)进行扩增, 扩增条件为:95 ℃预变性10 min;95 ℃变性40 s, 53 ℃退火45 s, 72 ℃延伸30 s, 35个循环;72 ℃最后延伸10 min.扩增体系为25 μL:Go Taq® Green Master Mix 12.5 μL, 正反向引物各0.5 μL, 牛血清蛋白(BSA) 0.25 μL, 稀释10倍的DNA模板1 μL, 无核酸酶纯水补足至25 μL.取5 μL PCR扩增产物用1%凝胶进行电泳验证, 通过Illumina MiSeq平台进行测序(上海美吉生物医药科技有限公司).
2.4 实时荧光定量PCR使用实时荧光定量PCR采用SYBR Green I染色法, 所用试剂为SYBR Premix Ex Taq TM试剂盒(TaKaRa, 大连).实验使用ABI 7500 Real-Time PCR System扩增仪(Applied Biosystems, CA, USA)进行定量.实验选用特异性引物pmoA-189b-F和com-amoA-1-R针对comammox功能基因amoA进行丰度的定量分析.扩增测序如下:95 ℃预变性5 min, 94 ℃变性30 s, 53 ℃退火45 s, 72 ℃延伸30 s, 共40循环.针对AOB-amoA的定量分析采用引物amoA-1F9和amoA-2R5(Rotthauwe, 1997), 扩增条件为95 ℃预变性30 s, 94 ℃变性10 s, 52 ℃退火30 s, 72 ℃延伸45 s, 共35循环.针对AOA-amoA的定量分析采用引物Arch-amoAF和Arch-amoAR (Francis C A et al., 2005), 扩增条件为95 ℃预变性30 s, 94 ℃变性10 s, 58 ℃退火30 s, 72 ℃延伸45 s, 共35循环.qPCR的反应为20 μL体系:SYBR Premix Ex Taq酶10 μL, ROX 0.4 μL, 牛血清蛋白(BSA) 0.4 μL, 正反向引物(10 mmol·L-1)各1 μL, 稀释10倍的DNA模板2 μL, 无核酸酶纯水补足至20 μL.按10倍浓度梯度的稀释质粒, 设置7个梯度, 3个平行.根据梯度曲线扩增得的Ct值和拷贝数绘制标准曲线;每个样品设有3个平行样, 用7500 SDS System配套软件进行分析计算数据结果, 得出AOA、AOB和comammox细菌的丰度值.其中, 阴性对照组定量结果须为未检测到(ND), 标准样品扩增效率介于90%~110%, 标准曲线相关性大于0.98, 扩增溶解曲线为单一峰.
2.5 序列分析高通量测序时用带有Barcode信息的引物对样品amoA进行PCR扩增以区分样品.通过Illumina MiSeq平台(上海美吉生物医药科技有限公司)完成amoA高通量测序.测序公司返回Fasta格式序列结果, 后续进行序列处理, 步骤如下:首先通过Qiime、Mothur、Excel、Notapad和Bioedit等软件检测并筛除低质量的序列, 且得到非冗余的核酸序列(Schloss et al., 2009; Caporaso et al., 2010).通过Bioedit软件将非冗余的核酸序列翻译成氨基酸序列, 然后通过Uclust软件以90%的相似度将氨基酸序列进行聚类(Edgar, 2010; Wang et al., 2018), 得到可操作分类单元(operational taxonomic units, OTUs)的分布结果.基于OTUs水平进行生态学分析:将每个OTUs代表的核酸序列在Genbank数据库上进行BLAST (Basic Local Alignment Search Tool)比对;通过编写的Perl脚本计算多样性等(庄林杰等, 2017).
2.6 统计分析使用去趋势对应分析(DCA)来确定数据中的单峰性或线性度.冗余分析(RDA)来确定这些环境变量解释的方差和comammox细菌群落结构的分布.根据Bray-Curtis差异度指数进行群落的分类和排序, 分类选择UPGMA算法(Mothur计算), 排序选用主坐标分析(Principle Coordinate Analysis, PCoA).用SPSS21.0软件(Statistical Product and Service Solutions)进行相关性分析(均采用Spearman相关系数), 本研究的显著性水平为α≤ 0.05.利用微软Office、Origin9.0、Gephi等软件作图.
3 结果(Results) 3.1 基本理化性质巢湖冬季和夏季表层沉积物样品呈酸性(pH值为5.83~6.82).在时间尺度上, 夏季样品的TOM、NO3-、NO2-和TS浓度显著高于冬季样品(p = 0.008、0.005、0.003和0.012).夏季样品的TN和TC浓度显著低于冬季样本(p = 0.026和0.042).夏季样品NH4+浓度((71.50±13.46) mg·kg-1)低于冬季样本((83.44±39.6) mg·kg-1), 差异不显著(p> 0.05).夏季样品的C/N值(8.81±1.41)高于冬季样品(8.41±0.96), 差异不显著(p> 0.05).在空间尺度上, NH4+含量存在差异(p = 0.082, n=6), 岸边带区域为(61.12 ± 17.52) mg·kg-1, 湖盆中心区域为(93.82 ± 29.78) mg·kg-1.岸边带沉积物TC含量((11.89±1.14) mg·kg-1)显著低于湖盆中心((13.01 ±1.49)mg·kg-1) (p = 0.02; n=6).
3.2 comammox细菌、AOA和AOB的amoA基因的丰度通过实时荧光定量PCR技术, 分别对巢湖表层沉积物样品的AOA、AOB和comammox细菌进行定量分析, 结果如图 2所示, 基于amoA基因的comammox细菌的丰度在WA6点最高, 为(4.06±1.23)×107 copies·g-1;在SH9点最低, 为(5.20±0.72)×106 copies·g-1.AOA的丰度为(5.39±1.01)×105 ~(1.60±0.18)×107 copies·g-1;AOB的丰度为(6.16±1.57)×105~(4.30±0.19)×106 copies·g-1.统计学分析表明, comammox细菌的丰度显著高于AOA和AOB (p < 0.05).岸边带comammox细菌的丰度((2.15±1.14)×107 copies·g-1)高于湖心区((1.25± 1.09)×107 copies·g-1).冬季comammox细菌的丰度((1.92±1.13)×107 copies·g-1)高于夏季((1.07±0.76)×107 copies·g-1).
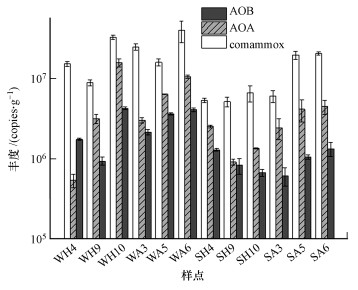 |
| 图 2 巢湖表层沉积物冬夏季comammox细菌、AOA和AOB的丰度 Fig. 2 The abundance of comammox, AOA and AOB in Chaohu Lake surface sediments in winter and summer |
将高通量测序获得的序列上传至NCBI, 登录号为SRR10336743-SRR10336754.对巢湖表层沉积物中comammox细菌的全部序列使用Qiime和Mothur等生物信息软件对序列进行严格质量控制, 选取涵盖每个注释种属99%序列数的OTUs.根据NCBI数据库中已探明的comammox细菌的amoA基因序列作为参比序列, 利用BLAST(basic local alignment search tool)进行序列比对和注释.对样品和OTUs进行双向二维聚类, 绘制Heatmap图, 通过颜色的梯度和相似程度来反映数据的相似性和差异性, 可以清楚地获知各OTU下的comammox来源和每一个样品下的comammox的群落分布.由图 3可以看出, 各样点的菌群结构差异度较大, 而通过聚类分析可得, 各样本聚类时体现出一定的时间效应, 相同季节的样品聚集在一起.说明时间的差异性对comammox细菌的群落结构影响较大.
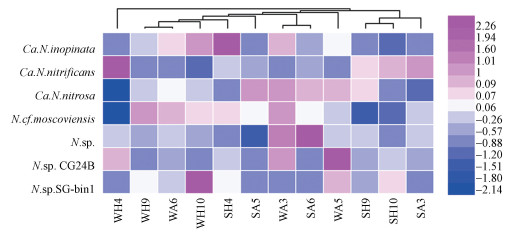 |
| 图 3 巢湖表层沉积物comammox细菌的热图 Fig. 3 The heatmap of comammox bacteria of surface sediments in Chaohu Lake |
在Uclust软件以90%相似度下进行聚类, 共得到154个OTUs, 挑出每个OTUs的代表氨基酸序列, 按序列比对分析做出相对丰度图, 如图 4a所示.按照种水平的分类, 各样点都注释到了3个comammox细菌即Candidatus Nitrospira nitrificans、Candidatus Nitrospira nitrosa和Candidatus Nitrospira inopinata(分别简写为Ca.N.nitrificans、Ca.N.nitrosa和Ca.N.inopinata;Nitrospira简写为N).其中在WH4样点Ca.N.nitrificans的相对丰度最高, 占比为78.72%, 在WA3样点Ca.N.nitrosa的相对丰度最高, 占比为49.80%, 在SH4样点Ca.N.inopinata的相对丰度最高, 占比为6.28%.在夏季样点中Ca.N.inopinata的相对丰度显著高于冬季样点(p < 0.05).对comammox细菌的Ca.N.nitrificans、Ca.N.nitrosa和Ca.N.inopinata进行细胞密度的计算如图 4b所示.统计分析显示, 岸边带中Ca.N.nitrificans的细胞密度(3.72±1.45)×106 cell·g-1高于湖心区(3.70±4.16)×106 cell·g-1.
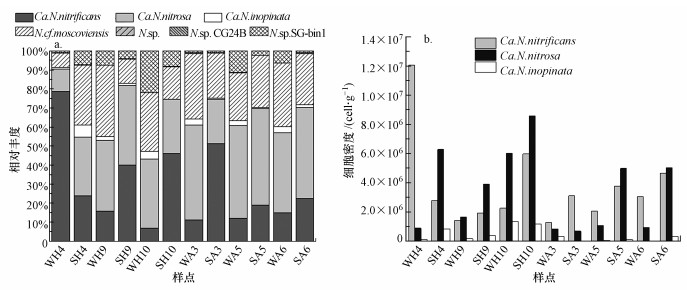 |
| 图 4 巢湖表层沉积物comammox菌的相对丰度图(a)和细胞密度(b) Fig. 4 Relative abundance(a) and cell density(b) of comammox bacteria of surface sediments in Chaohu Lake |
本研究中涉及的多样性分析包括α-多样性指数即丰富度指数(Chao1指数)、优势度指数(Simpson指数)和多样性指数(Shannon指数)和β多样性.Chao1指数是指群落中含有OTUs数目的指数, 其数值越高代表群落内物种的数目越多.Simpson指数表示在一个群落中随机取样的两个属于不同种个体的概率.Shannon指数主要反映群落种数和各种间个体分配的均匀性;表 2为各样点α-多样性指数由软件Mothur计算所得.由表 2可知, Chao1指数最高的是WH9样点(927.016667), 最低的是SA5(472.807692);Shannon指数最高的是SH4(3.782816), 最低的是SA5(3.246777);Simpson指数最高的WH4(0.125015), 最低的是SH4(0.051590).通过α-多样性指数分析可得SH4样点的物种多样性相对较高, 而SA5样点的物种多样性相对较低.Chao1指数冬季样点远大于夏季样点, 而冬夏季样点的Shannon指数和Simpson指数差异不大, 由此在一定程度上可以反应冬季巢湖表层沉积物中的comammox细菌的物种多样性大于夏季.
| 表 2 巢湖表层沉积物完全氨氧化菌的α-多样性指数 Table 2 α-diversity index of coammmox bacteria in Chaohu Lake surface sediments |
在OTUs水平计算巢湖表层沉积物样品的Bray-Curtis差异度矩阵, 利用Qiime软件做主坐标轴图(Principal Coordinate Axis Analysis, PCoA) (图 5).如图 5所示, 两轴对排序结果的解释度分别为48.44%和15.43%.PcoA结果分析表明, 在巢湖表层沉积物中夏季样品聚集在一起, 冬季样品聚集在一起, 说明comammox细菌的群落结构具有时间异质性;岸边带样品聚集在一起, 湖心区样品聚集在一起, 说明comammox细菌的群落结构具有空间间异质性.
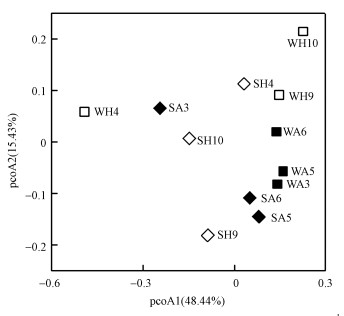 |
| 图 5 巢湖表层沉积物comammox细菌pcoA图 Fig. 5 Principal coordinates analysis of comammox bacteria in Chaohu Lake surface sediments |
通过各样点comammox菌的amoA功能基因的丰度及菌群相对丰度, 并结合其理化性质进行斯皮尔曼相关性分析(表 3)和冗余分析(RDA) (图 6)来探究环境影响因子对巢湖表层沉积物中comammox细菌的群落结构的影响.
| 表 3 巢湖表层沉积物comammox菌群与环境因子相关性分析 Table 3 Correlation analysis between community structure of comammox bacteria and chemical characters of soil (n=12) |
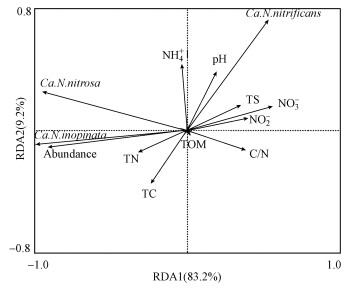 |
| 图 6 巢湖表层沉积物RDA图 Fig. 6 RDA analysis of comammox bacteria community and environmental factors |
由RDA和斯皮尔曼分析可得, comammox的丰度与NO3-呈显著负相关(r=-0.636, p=0.026), 与NH4+、NO2-、TS、TOM、pH和C/N呈负相关, 与TC和TN呈正相关.Ca.N.nitrificans与NO2-呈显著正相关(r=0.587, p=0.045), 与NH4+、NO3-、TS、TOM、pH和C/N呈正相关, 与TC和TN呈负相关.Ca.N.nitrosa与NH4+、NO2-、NO3-和C/N呈负相关, 与TC、TN、TS、TOM和pH呈正相关.Ca.N.inopinata与NH4+、NO2-、NO3-、TS、TOM和pH呈负相关, 与TN和C/N呈正相关.
3.6 comammox菌群间的相互作用基于R语言的psych软件包, 通过斯皮尔曼相关系数, 采用pairwise分析, 对各样点的物种在种水平上的OTUs进行相关性分析, 将ρ>0.8和p-value < 0.05的物种间的相互作用关系用网络图的形式展示(图 7).图中有123个节点, 231条边.按照种属进行划分得出Ca.N.nitrificans占比最高为49.59%, Ca.N.nitrosa占比为10.57%, Ca.N.inopinata占比为1.63%.且Ca.N.nitrificans与其他种属连接最紧密, 是网络图中的关键菌属.图中蓝线代表正相关关系(83.55%), 红线代表负相关关系(16.45%).
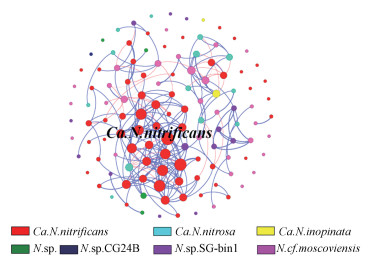 |
| 图 7 巢湖comammox细菌的生态网络分析 Fig. 7 Molecular ecological network analysis of comammox bacteria in Chaohu Lake |
本研究运用高通量测序以及实时荧光定量PCR技术对巢湖冬夏两季的表层沉积物中的完全氨氧化过程进行实验研究.分析了comammox细菌在湖泊湿地巢湖表层沉积物中的分布规律、群落结构、多样性等, 并研究了其影响因素.
4.1 低氨影响commammox细菌的丰度和细胞密度在空间尺度上岸边带中comammox细菌的丰度高于湖心区;岸边带中Ca.N.nitrificans的细胞密度高于湖心区.相邻两个生态系统之间的界面通常包含生物地球化学循环的热区, 长期以来, 岸边带一直被认为是氮循环的生物地球化学热点(McClain et al., 2003), 具有较高的氨氧化速率(Wang.et al. 2012)、耗氧率(Wang et al., 2002)和氮消耗率(Hefting et al., 2006).湖泊岸边带是陆地与水生生态系统之间的过渡边界, 风力、地表径流和人为活动等因素都会对其造成扰动, 使沉积物与上覆水之间存在着频繁的环境变化和物质交换(Alvarez-Cobelas et al., 2005), 相反, 湖盆中心沉积物受到的干扰较小.以往研究发现comammox对底物氨和氧具有更高的亲和力, 能够更好地适应极低氨浓度和低氧或微量氧环境(Costa, 2006; Daims et al., 2015; Kits et al., 2017), 岸边带是氮循环发生的热区, 岸边带沉积物由于水位频繁波动处于富氧-缺氧交替条件下(Cirmo, 1997; McClain, 2003);湖心区沉积物长期处于缺氧条件.在本研究中岸边带土壤中氨浓度显著低于湖心区, 因此在岸边带中comammox细菌的丰度和细胞密度高于湖心区.
4.2 pH影响comammox细菌的丰度本研究中comammox的丰度显著高于AOA和AOB, 样品pH呈弱酸性.comammox细菌的丰度与pH呈负相关关系这与Shi等的研究结果一致(Shi et al., 2018).pH被认为是影响陆地环境中氨氧化微生物的生物地理分布和代谢活性的主要因素(Nicol et al., 2008; He et al., 2012; Prosser and Nicol, 2012; Hu et al., 2013).氨(NH3)而非铵离子(NH4+)被普遍认为是氨氧化剂的直接底物, 酸性土壤由于氨被氢离子电离而具有氨利用率低的特点(Hu et al., 2014).有研究显示酸性土壤中, comammox细菌的丰度最高, 其次是AOA, AOB丰度相对较低(Hu et al., 2017).这些发现表明, comammox细菌也可能是在酸性土壤中硝化的一个重要贡献者, 这挑战了先前的观点, 即AOA而不是AOB对酸性土壤中的硝化贡献很大(He et al., 2012; Hu et al., 2014).Shi等研究中pH和NH4+对comammox细菌的丰度的贡献率最大分别为27.2%和22.7%(Shi et al., 2018).另外有研究显示当沉积物的pH值为7.7~8.0, pH对comammox的分布没有明显影响(Jiang et al., 2019).因此comammox细菌可能更适应于酸性环境.
4.3 Ca.N.nitrificans在该环境中起着重要的作用RDA和斯皮尔曼分析显示对Ca.N.nitrificans的相对丰度影响最大的理化因子是NH4+和pH, 且与其呈正相关.再结合生态网络分析得出Ca.N.nitrificans与其他种属连接最紧密, 是网络图中的关键菌, 表明其在comammox菌群中起着重要作用.有研究显示Ca.N.nitrificans可在pH为6.99±0.1的条件下存在(van Kessel et al., 2015), 而本研究中样品的pH均值为6.47.研究表明pH是影响细菌多样性和群落结构的重要决定因素(Fierer et al., 2006).Kit等最新研究结果显示根据氨氧化动力学分析comammox (Ca.N. inopinata)的Km (μmol·L-1 NH3)值为0.063, 显著低于AOA Km(0.41~0.96 μmol·L-1 NH3), 且比AOB低了4~2500倍.由此我们猜测Ca.N.nitrificans的Km可能低于Ca.N.inopinata, 使其在该环境中起着重要的作用.为获得更有代表性的comammox菌株并测定其氨氧化动力学, 需要进行下一步的研究.
5 结论(Conclusions)本文运用高通量测序技术对巢湖表层沉积物中comammox进行研究发现, 低氨影响commammox细菌的丰度和细胞密度, 岸边带中comammox细菌的丰度和细胞密度高于湖心区;comammox细菌可能更适应于酸性环境.在一定程度上揭示了comammox细菌的分布具有空间异质性, 且在酸性环境中相比于传统氨氧化微生物可能更有优势, 为更好地研究湖泊湿地沉积物中氨氧化过程提供了良好的基础.但comammox细菌各种属和关键理化因子间的生物调节机制, comammox与传统氨氧化过程在氮循环中所占比重以及comammox的活性检验等都有待进一步研究.
Alavrez-Cobelas M, Velasco J L, Valladolid M, et al. 2005. Daily patterns of mixing and nutrient concentrations during early autumn circulation in a small sheltered lake[J]. Freshwater Biology, 50(5): 813-829. |
Annavajhala M K, Kapoor V, Santo-Domingo J, et al. 2018. Comammox functionality identified in diverse engineered biological wastewater treatment systems[J]. Environmental Science and Technology Letters, 5(2): 110-116. |
鲍士旦. 2000. 土壤农化分析(第3版)[M]. 北京: 中国农业出版社.
|
Bartleme R P, Mclellan S L, Newton R J. 2017. Freshwater recirculating aquaculture system operations drive biofilter bacterial community shifts around a stable nitrifying consortium of ammonia oxidizing archaea and comammox nitrospira[J]. Frontiers in Microbiology, 8: 101. |
Caporaso J G, Kuczynski J, Stombaugh J, et al. 2010. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 7(5): 335. |
Chao Y Q, Mao Y P, Yu K, et al. 2016. Novel nitrifiers and comammox in a full-scale hybrid biofilm and activated sludge reactor revealed by metagenomic approach[J]. Applied Microbiology and Biotechnology, 100(18): 8225-8237. |
Chen X, Yang X, Dong X, et al. 2011. Nutrient dynamics linked to hydrological condition and anthropogenic nutrient loading in Chaohu Lake (southeast China)[J]. Hydrobiologia, 661(1): 223-234. |
Cirmo C P, Mcdonnell J J. 1997. Linking the hydrologic and biogeochemical controls of nitrogen transport in near-stream zones of temperate-forested catchments:A review[J]. Journal of Hydrology, 199(1/2): 88-120. |
Costa E, Pérez J, Kreft J U. 2006. Why is metabolic labour divided in nitrification?[J]. Trends in Microbiology, 14(5): 213-219. |
Daims H, Lebedeva E V, Pjevac P, et al. 2015. Complete nitrification by Nitrospira bacteria[J]. Nature, 528(7583): 504509. |
Edgar R C. 2010. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 26(19): 2460. |
Fan X, Gao J, Pan K, et al. 2017. Temporal dynamics of bacterial com munities and predicted nitrogen metabolism genes in a full-scale waste watertreatment plant[J]. RSC Advances, 7(89): 56317-56327. |
Fierer N, Jackson R B. 2006. The diversity and biogeography of soil bacterial communities[J]. Proc Natl Acad Sci USA, 103: 626-631. |
Fowler S J, Palomo A, Dechesne A, et al. 2018. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities[J]. Environmental Microbiology, 20(3): 1002-1015. |
Francis C A, Roberts K J, Beman J M, et al. 2005. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 102: 14683-14688. |
Gonzalez-Martinez A, Rodriguez-Sanchez A, van Loosdrecht M C M, et al. 2016. Detection of comammox bacteria in full-scale wastewater treatment bioreactors using tag-454-pyrosequencing[J]. Environmental Science and Pollution Research, 23(24): 25501-25511. |
Hefting M M, Bobbink R, Janssens M P. 2006. Spatial variation in denitrification and N2O emission in relation to nitrate removal efficiency in a n-stressed riparian buffer zone[J]. Ecosystems, 9: 550-563. |
Heinrich R, Schuster S. 1996. The regulation of cellular systems[M]. Berlin: Springer.
|
He J Z, Hu H W, Zhang L M. 2012. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils[J]. Soil Biol Biochem, 55: 146-154. |
Hu H W, Trivedi P, He J Z, et al. 2017. Microbial nitrous oxide emissions in dryland ecosystems:Mechanisms, microbiome and mitigation[J]. Environmental Microbiology, 19(12): 4808-4828. |
Hu H W, Xu Z H, He J Z. 2014. Ammonia-oxidizing archaea play a predominant role in acid soil nitrification[J]. Adv Agron, 125: 261-302. |
Hu H W, Zhang L M, Dai Y, et al. 2013. pH-dependent distribution of soil ammonia oxidizers across a large geographical scale as revealed by high-throughput pyrosequencing[J]. Journal of Soils Sediments, 13: 1439-1449. |
Hu H W, He J Z. 2017. Comammox a newly discovered nitrification process in the terrestrial nitrogen cycle[J]. Journal of Soils & Sediments, 17(12): 2709-2717. |
Jiang Q, Xia F, Zhu T, et al. 2019. Distribution of comammox and canonical ammonia oxidizing bacteria in tidal flat sediments of the Yangtze River estuary at different depths over four seasons[J]. Journal of Applied Microbiology, 127(2): 533-543. |
Jetten M S, Lv N, Strous M, et al. 2009. Biochemistry and molecular biology of anammox bacteria[J]. Critical Reviews in Biochemistry and Molecular Biology, 44(2/3): 65-84. |
Kits K D, Sedlacek C J, Lebedeva E V, et al. 2017. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature, 549(7671): 269-272. |
Könneke M, Bernhard A E, José R, et al. 2005. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 437(7058): 543. |
Kits K D, Sedlacek C J, Lebededeva E V, et al. 2017. Kinetic Analysis of a finished nitrifier revolutionary styles[J]. Nature, 549(7671): 269-272. |
Luesken F A, Alen T A V, Biezen E V D, et al. 2011. Diversity and enrichment of nitrite-dependent anaerobic methane oxidizing bacteria from wastewater sludge[J]. Applied Microbiology and Biotechnology, 92(4): 845. |
Maixner F, Noguera D R, Anneser B, et al. 2010. Nitrite concentration influences the population structure of Nitrospira-like bacteria[J]. Environmental Microbiology, 8(8): 1487-1495. |
McClain M E, Boyer E W, Dent C L, et al. 2003. Biogeochemical hot spots and hot moments at the interface of terrestrial and aquatic ecosystems[J]. Ecosystems, 6(4): 301-312. |
Naiman R J, Décamps, H. 1997. The ecology of interfaces Riparian zones[J]. Annual Review of Ecology and Systematics, 28(1): 621-658. |
Nicol G W, Leininger S, Schleper C, Prosser J I. 2008. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria[J]. Environmental Microbiology, 10: 2966-2978. |
Palmo A, Jane F S, Gülay A, et al. 2016. Metagenic Analysis of rapid gravity and filter microbiotic communities survey novel physics of Nitrospira.spp[J]. ISME Journal, 10(11): 2569-2581. |
Pan K L, Gao J F, Fan X Y, et al. 2018. The more important role of ar chaea than bacteria in nitrification of wastewater treatment plants in cold season despite their numerical relationships[J]. Water Research, 145: 552. |
Pfeiffer T, Bonhoeffer S. 2004. Evolution of cross feeding in microbial populations[J]. The American Naturalist, 163: 126-135. |
Pinto A J, Marcus D N, Zeeshan I U, et al. 2016. Metagenomic Evidence for the Presence of Comammox Nitrospira-Like Bacteria in a Drinking Water System[J]. Msphere, 1(1): e00054-15. |
Pjevac P, Schauberger C, Poghosyan L, et al. 2017. AmoA-targeted polymerase chain reaction primers for the specific detection and quantification of Comammox Nitrospirain the environment[J]. Frontiers in Microbiology, 8: 1508. |
Prosser J I, Nicol G W. 2012. Archaeal and bacterial ammonia-oxidizers in soil:the quest for niche specialisation and differentiation[J]. Trends Microbiology, 20: 523-531. |
Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63: 4704-4712. |
Schloss P D, Westcott S L, Ryabin T, et al. 2009. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied Environmental Microbiology, 75(23): 7537-7541. |
Shi X, Hu H W, Wang J, et al. 2018. Niche separation of comammox Nitrospira and canonical ammonia oxidizers in an acidic subtropical forest soil under long-term nitrogen deposition[J]. Soil Biology and Biochemistry, 126: 114-122. |
Tatari K, Musovic S, Gülay A, et al. 2017. Density and distribution of nitrifying guilds in rapid sand filters for drinking water production:Dominance of Nitrospira spp[J]. Water Research, 127: 239-248. |
Teske A, Alm E, Regan J M, et al. 1994. Evolutionary relationships among ammonia-and nitrite-oxidizing bacteria[J]. Journal of bacteriology, 176(21): 6623-6630. |
Tobias C, Giblin A, McClelland J, et al. 2003. Sediment DIN fluxes and preferential recycling of benthic microalgal nitrogen in a shallow macrotidal estuary[J]. Marine Ecology Progress Series, 257: 25-36. |
Van Kessel M A, Speth D R, Albertsen M, et al. 2015. Complete nitrification by a single microorganism[J]. Nature, 528(7583): 555. |
Wang S Y, Wang W D. 2018. Microbial nitrogen cycle hotspots in the plant-bed/ditch system of a constructed wetland with N2O mitigation[J]. Environmental Science & Technology, 52(11): 6226-6236. |
Wang W D, Wang D L, Yin C Q. 2002. A field study on the hydrochemistry of land/inland water ecotones with reed domination[J]. Acta Hydrochimica et Hydrobiologica, 30(2/3): 117-127. |
Wang Y, Zhu G, Ye L, et al. 2012. Spatial distribution of archaeal and bacterial ammonia oxidizers in the littoral buffer zone of a nitrogen-rich lake[J]. Journal of Environmental Sciences, 24(5): 790-799. |
Wang Y L, Ma L P, Mao Y P, et al. 2017. Comammox in drinking water systems[J]. Water Research, 116: 332-341. |
Wang Z, Cao Y, Zhu-Barker X, et al. 2019. Comammox Nitrospira clade B contributes to nitrification in soil[J]. Soil Biology and Biochemistry, 135: 392-395. |
Winogradsky S. 1890. The morphology of the contributions of nitrification system[J]. Archives of Biological Sciences, 4: 257-275. |
张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理, 30(3): 137-141. |
Zhu G, Wang S, Wang Y, et al. 2011. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. Isme Journal, 5(12): 1905-1912. |
庄林杰, 夏超, 田晴, 等. 2017. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 37(1): 261-271. |
 2020, Vol. 40
2020, Vol. 40


