抗生素因其良好的抗病原体作用与抑菌效果, 被广泛应用于医药行业、农业与畜牧业.长期以来, 过度使用和滥用抗生素导致了抗生素耐药细菌(ARB)的产生与流行, 降低了其对人类和动物病原体的治疗潜力(Sharma et al., 2016).与抗生素类似, 一些金属离子也被广泛应用于医疗、畜牧等领域, 作为抗菌剂与生长促进剂等, 也引发了相关耐药菌的产生与流行(Hobman et al., 2015).在抗生素与抗菌金属的复合作用下, 导致了一些抗生素与金属协同抗性细菌的产生(Akinbowale et al., 2007;Hu et al., 2007).目前, 抗菌药物耐药性(AMR)已成为公共卫生的主要威胁, 预计到2050年, AMR导致的年死亡人数将增加到1000万(O′Neill, 2016).致病细菌的耐药性已成为公共卫生中的全球性问题, 因而越来越受到人们的关注(Berendonk et al., 2015).
城市水环境与人类生活息息相关, 生活污水、医院废水、工业废水、养殖场废水等都进入城市污水系统, 相应地, 未经人体与动物体吸收的抗生素、抗菌金属药物通过这些途径进入污水处理厂与城市河道中(Huang et al., 2019).污水处理厂、城市污水河道被认为是耐药细菌与抗性基因的潜在来源(Ferro et al., 2017), 而城市污水的再循环将进一步推动耐药菌的持续存在与传播, 进而影响相关的生态系统(Taylor et al., 2011).肠杆菌科细菌在自然界分布广泛, 多数为条件致病菌, 影响着人体的健康, E. coli作为其中的代表菌, 能较好地反映出肠杆菌科细菌的耐药情况与城市水环境受污染状况(Edberg et al., 2000).
黑龙江省地处中国最北端, 属于中国的寒区, 一年中冬季长达5个月左右, 其省会城市哈尔滨市冬季平均气温在-18 ℃左右.一些研究显示, 低温会增加耐药菌株对一些抗菌药物的耐药性, 在冷应激状态下对抗菌药物有更强的适应性(Al-Nabulsi et al., 2015).此外, 低温环境还是抗性基因水平转移的重要驱动因素, 会引起基因转移元件与抗性基因的增殖(Miller et al., 2014;Son et al., 2018).因此, 研究低温环境下细菌的耐药性越发重要.
本研究以肠杆菌科细菌中的E. coli为研究对象, 通过调查哈尔滨市城市污水河道污泥中抗生素抗性和金属耐受性细菌的存在状况, 以期为现有抗性细菌所致危害的减轻及科学认识城市污水与生态健康和公共安全的关系提供参考.
2 材料与方法(Materials and methods) 2.1 样品采集采样区域为哈尔滨市城市污水河道—马家沟河, 该河是松花江干流的南岸支流, 位于黑龙江省南部, 流经哈尔滨市4个区、2个风景区, 全长44.3 km, 其中哈尔滨市区河段长34.7 km, 河道宽30~100 m, 流域面积为240 km2.河水主要来源包括居民生活污水、工厂废水及自然降水, 污水直接排入松花江, 严重影响了松花江水质, 给沿岸人们的生产生活带来了巨大影响(张博, 2013).鉴于污染的严重性, 马家沟河流域一直在持续治理中(解海, 2003).本课题组于2018年6月在马家沟河流域进行河道淤泥的采集, 共选取4个取样点(图 1), 取样点概况如表 1所示.每个取样点采集4份土样, 每份约200 g, 用无菌袋收集于冰上保存, 运回实验室.
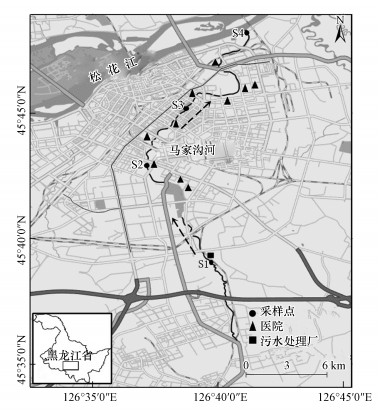 |
| 图 1 采样点位置示意图 Fig. 1 Map of sampling sites |
| 表 1 取样点概况 Table 1 The overview of sampling sites |
每个取样点的4份样品充分混合, 从中取出10 g样品置于90 mL生理盐水(0.9%, m/V)中, 充分振荡混匀, 使土样充分溶解于生理盐水中, 随后进行逐级梯度稀释(10-2、10-3、10-4、10-5、10-6、10-7).利用紫红胆盐葡萄糖琼脂(VRBG)进行肠杆菌的分离(Pourcher et al., 2014), 利用TBX agar、吲哚试验与氧化酶试验从肠杆菌中筛选E. coli, 若TBX agar显蓝色、吲哚阳性、氧化酶阴性, 则判定为E. coli(Ibrahim et al., 2016).对实验中所分离的所有肠杆菌进行16S rDNA的扩增与测序鉴定(Sah et al., 2018), 分离菌株在Genbank的序列登录号为MN416242~MN416279.
2.3 E. coli的抗生素表型检测根据NCCLS与CLSI指南, 参照Ibrahim等(2016)相关研究检测了E. coli关于17种抗生素的表型.试验中使用的所有抗生素列于表 2, 抗生素均由英国Oxoid公司提供, 试验中以E. coli ATCC25922为质控菌株, 其购于北纳创联生物科技有限公司.
| 表 2 用于E. coli抗性表型鉴定的抗生素 Table 2 Antibiotic assay discs and amount of antibiotic contained in each disc |
金属表型的检测参照NCCLS与CLSI指南, 检测E. coli对4种金属(Hg、Ag、Zn、Cu)的抗性, 按照2倍稀释法对药液母液(4096 μg·mL-1)进行逐级梯度稀释, 浓度范围为2048~0.25 μg·mL-1.每个样品3次重复, 试验中以E. coli ATCC25922为质控菌株, 样品于96孔板中培养, 37 ℃恒温培养箱中培养18~20 h, 培养后的96孔板置于酶标仪中观察生长状况, 记录MIC50、MIC90.
2.5 DNA提取从培养18~24 h的LB琼脂平板中挑选1~2个菌落悬浮于5 mL Mueller-Hinton broth中, 然后将悬液在37 ℃下150~200 r·min-1振荡24 h, 取1.5 mL细菌培养液, 12000 r·min-1离心1 min;取沉淀, 用100 μL TE溶液(pH=8.0)重复洗2次;12000 r·min-1离心1 min, 取沉淀, 加100 μL TE溶液悬浮, 98 ℃水浴10 min, 冰上放置5 min;12000 r·min-1离心1 min, 将上清液转移到无菌的微量离心管中, 并保存在-20 ℃条件下直到使用(邵碧英等, 2007).
2.6 抗性基因检测与整合酶基因检测使用PCR检测所有E. coli分离株的ESBLs基因(blaSHV、blaTEM、blaCTX-M)、四环素类抗性基因(tetA、tetM)、磺胺类抗性基因(sul1、sul2)、喹诺酮类抗性基因(qnrB、qnrS)、链霉素抗性基因(strA、strB、addaA)、复方磺胺甲恶唑抗性基因(dfrA1、dfrA5、dfrA12)、汞耐受基因(merC)、银耐受基因(silB、silE)、铜耐受基因(pcoE)和整合酶基因(intI1).靶标基因的引物、PCR体系及反应条件基于前人所述(Liebert et al., 1997;Goldstein et al., 2001;Grape et al., 2007;Dierikx et al., 2012;Titilawo et al., 2015;Asiani, 2016;Hong et al., 2018), 表 3提供了引物序列.PCR结果送由哈尔滨擎科生物科技公司测序, 并通过美国国家生物技术信息中心(NCBI)进行比对分析.
| 表 3 抗性基因与整合酶基因检测所用引物及PCR条件 Table 3 PCR details for resistance genes and integrase genes |
利用软件MEGA 6.0对实验中分离的肠杆菌进行系统发育树分析.
3 结果与讨论(Results and discussion) 3.1 样品分离结果利用VRBG agar从4个样点的河道淤泥中共分离到455株肠杆菌, 具体结果如图 2所示.结果显示, 共分离到Escherichia、Citrobacter、Enterobacter、Klebsiella、Yersinia、Raoultella、Lelliottia、Serratia、Kluyvera、Rahnella、Cronobacter、Hafnia、Moellerella、Pantoea、Cedecea 15个属, 38个种.进一步经TBX培养基、氧化酶试验与吲哚试验从肠杆菌中筛选出110株E. coli, 各个样点肠杆菌、E. coli的分离组成比例如图 3a所示, E. coli的整体检出率约为24.2%.图 3b箱线图的分析结果表明, 4个取样点的E. coli检出率未出现偏离, 所以数值均具代表性.
 |
| 图 2 本研究中所分离细菌的系统发育分析(构建方法为Neighbor-Joining法, 分枝节点数字表示Bootstrap值) Fig. 2 Phylogenetic tree analysis of isolates in this study(Construction method is Neighbor-Joining Tree, the number of branch nodes indicates the bootstrap value) |
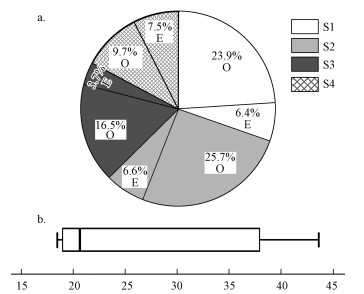 |
| 图 3 不同样点E. coli的分离结果(a.肠杆菌分离比例(E: E. coli, O:others);b. E. coli检出率箱线图) Fig. 3 The isolation results of E. coli at different sampling sites (a.The isolation precentage of Enterobacteriaceae(E:E.coli, O:others); b. The boxplot of E. coli detection) |
在本研究中, 使用标准抗生素药敏纸片法测试了110株E. coli关于17种抗生素的敏感性.没有菌株对所有抗生素敏感, 69.1%的分离株对至少1种抗生素具有抗性, 45.5%的分离株为多耐药菌株(MDR, 显示出对3种或3种以上不同抗生素的抗性).图 4显示了所有菌株的抗性分布范围.
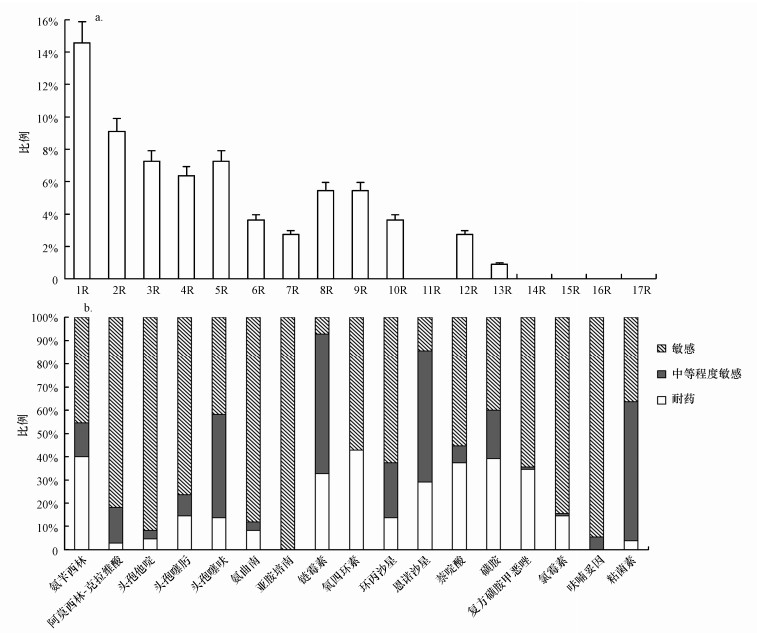 |
| 图 4 分离的E. coli群体中不同抗生素抗性种类E. coli所占比例(a, 横坐标中1R表示对1种抗生素具有抗性, 其余类推)及同种抗生素中E. coli的抗性分布情况(b) Fig. 4 Percentage of E. coli strains with antibiotic resistances(a) and percentage sensitivity to 17 antibiotics for 110 E. coli isolates(b) |
图 4b显示了整个大肠菌群110株E. coli对每种抗生素敏感性情况.在所测试的菌株中, 抗氧四环素的比例最高(42.7%), 其次是氨苄西林(40.0%)、磺胺(39.1%)、萘啶酸(37.3%).该结果与Divya等(2019)和Poma等(2016)对城市水环境中E. coli耐药性的研究结果类似, 这几种药物使用历史较早, 像磺胺1937年引入使用(Davies et al., 2010), 青霉素1942年引入使用(Qiao et al., 2018), 四环素1948年引入使用(Nelson et al., 2011);据Zhang等(2015)报道, 青霉素、四环素、磺胺是我国居民使用量排名前几的抗生素, 较长的使用历史与较大的使用量导致了该结果的产生.分离菌株对头孢菌素家族中第三代头孢菌素的耐药率分别为头孢噻肟14.6%、头孢噻呋13.6%、头孢他啶4.6%, 头孢菌素一直也是我国居民使用较多的抗生素(Zhang et al., 2015), 具有较广的抗菌谱.结果显示, 阿莫西林-克拉维酸与粘菌素对这些菌株具有较大抑制作用, 只有2.7%与3.6%的分离株对其具有抗性.所有的E. coli分离株中, 没有一株菌对亚胺培南和呋喃妥因具有抗性, 这与大多数的研究类似(Sanchez et al., 2016;Ranjbar et al., 2017;Lien et al., 2017;Araujo et al., 2017), 表明硝基呋喃类、碳青霉烯类仍是抵抗E. coli效果良好的抗生素.
表 4显示了各个取样点E. coli对17种抗生素的耐药情况, 取样点S1靠近该流域的源头, 取样点S2、S3位于人为活动密集区域, 取样点S4位于流域尽头区域, 可以发现空间水平上, 除了氨苄西林, E. coli对于其他抗生素的耐药率并不是呈逐渐递增的趋势, 对大多数抗生素的耐药率在前3个样点呈现出逐级递增的趋势, 而在最后一个样点耐药率却下降了.S1区域仅少数菌对几种抗生素呈现出较弱程度的抗性, 该区域位于污水处理厂的上游, 周围人为活动较弱;而人为活动较强的S2、S3区域的分离株对氨苄西林、头孢噻肟、氨曲南、头孢噻呋、链霉素、环丙沙星、恩诺沙星、萘啶酸、复方磺胺甲恶挫、磺胺、氯霉素、氧四环素12种抗生素具有抗性, 且对氨苄西林、链霉素、复方磺胺甲恶唑、磺胺、氧四环素5种抗生素的耐药率都在40%以上, 60%以上的E. coli对2种(含2种)以上的抗生素具有抗性, 抗性最强的抗13种抗生素的菌株DL27就位于S2区域, 该区域另有两株分离株抗12种抗生素, S2、S3区域位于污水处理厂的下游, 如前所述, 污水处理厂可能是抗性细菌的潜在储库(Ferro et al., 2017).与整体结果相同, 各样点中E. coli对氧四环素、氨苄西林、磺胺、萘啶酸的耐药率依然高于其他抗生素.
| 表 4 不同采样点E. coli的抗生素耐药水平 Table 4 Antibiotic resistance levels of E. coli at different sampling sites |
本研究检测了110株E. coli关于金属(Hg、Ag、Zn、Cu)的MIC, 试验中以E. coli ATCC25922为质控菌株.表 5显示了110株E. coli关于4种金属MIC范围的分布, 仅少数分离菌株(3.6%)对Hg具有较弱程度耐受, 分离株的MIC50与MIC90多集中于4 μg·mL-1与8 μg·mL-1;40.9%的分离株关于Ag的MIC90集中于16 μg·mL-1, 呈现出弱耐受性, 40.9%的抗银分离株中超过半数的菌株来源于取样点S1, 而样点S1靠近源头, 人为活动较弱.超过90%的分离株关于Zn的MIC50与MIC90都为512 μg·mL-1, 只有3.6%的分离菌株对Zn具有耐受性.大部分的分离株关于铜的MIC50和MIC90与质控菌株E. coli ATCC25922相同, 没有一株菌的MIC90超过1024 μg·mL-1, 因而所有菌株对Cu都不具有耐受性.
| 表 5 E. coli不同金属耐受性的MIC值 Table 5 The MIC of E. coli for metal resistances |
一直以来, 不管是作为抗菌药剂, 还是充当医疗器械, Hg、Ag在医疗行业都发挥着重要的作用(Hobman et al., 2015).近年来, 纳米银胶粒作为一种新型的抗菌药物用于一些疾病的治疗, 具有良好的治疗效果(Saeb et al., 2017);Cu、Zn在畜牧养殖中扮演着极其重要的角色, 作为抗菌剂治疗动物感染, 作为促生长因子提高动物产量(Deng et al., 2018).本研究中, 没有菌株对铜具有耐受性, 仅3.6%的菌株对Zn具有耐受性, 40.9%的菌株对Ag具有耐受性, 3.6%的菌株具有Hg耐受性.如图 1所示, 该河流沿岸有大小医院十余个, 无畜牧场、养殖场及屠宰场, 这也就解释了分离株的Hg、Ag耐受性、抗生素抗性及不存在的Cu耐受性, Hg、Ag耐受性与抗生素抗性可能就是医院废水、生活污水、污水处理厂处理过的水排放所引起.样点S1中检测的多数E. coli呈现出银耐受性, 根据文献报道, 银耐受性的产生与银化合物在医疗和生活中的抑菌应用有关(Pal et al., 2017).S1附近无医院, 且位于污水处理厂的上游, 对其耐受性形成原因需要进一步解析.
人们最初研究耐汞细菌对金属Hg的耐受性时, 发现其体内含有的质粒R100赋予细菌Hg耐受性的同时, 还赋予其对链霉素、四环素、磺胺和氯霉素的抗性, 后来陆续出现了类似的同时具有金属与抗生素抗性的微生物报道(Pal et al., 2017).近年来, 越来越多具有抗生素与金属协同抗性的微生物被报道.例如, Matyar等(2008)从伊斯肯德伦湾中分离了236株革兰氏阴性细菌, 对16种抗生素和5种金属具有耐受性, 并在海中的虾体内分离出对抗生素和金属具有协同抗性的革兰氏阴性细菌;杨芬等(2017)研究一株土壤分离菌株的抗生素与金属抗性时发现, 低浓度的Zn2+与青霉素钠和链霉素之间表现为协同抗性, Zn2+与庆大霉素、Zn2+与新霉素之间具有交叉抗性;李彬辉等(2012)发现在金属和抗生素交叉环境下, G-菌能激发出更多的抗性.本研究中, 有30株E. coli(27.3%)对至少1种抗生素和1种金属具有抗性, 其中一株分离株E. coli DL35甚至对9种抗生素和2种金属具有抗性, 具体结果如图 5所示.这些结果说明在抗生素与金属的交叉作用或协同作用下会增加耐药菌抗性的可能性.
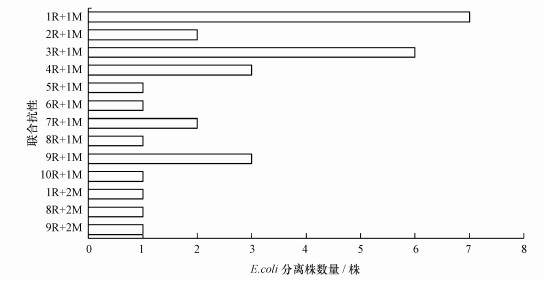 |
| 图 5 E. coli抗生素与金属的联合抗性概况(R.抗生素, M.金属, 1R+1M表示对1种抗生素和1种金属具有抗性, 其余类推) Fig. 5 The combination spectrum of E. coli for antibiotic resistance and metal resistance(R indicates antibiotic species, M indicates mentals) |
本研究共检测了15种ARGs、4种MRGs与整合酶基因intI1, 对110株E. coli进行了所有基因的检测, 具体结果如图 6和图 7所示.图 6反映了110株E. coli中15种ARGs、4种MRGs与整合酶基因intI1在不同样点的分布概况, 四环素抗性基因tetA在4个样点的检出率都很高, 反映了tetA基因在该流域的广泛分布与流行.多数抗生素抗性基因在S2、S3、S4样点的分布明显高于S1样点, 前面提到S2、S3、S4是人为活动相对较强的区域, 朱永官等(2013)报道抗生素的过度使用会引起ARGs的富集, 转座酶会使得抗性基因转移到人类致病菌中, 图 6也反映出人为活动的强弱同时决定着细菌体内抗性基因的存在.抗生素抗性基因blaSHV、qnrB、dfrA1在人为活动较强的S2、S3区域的分布明显低于源头S1区域与人为活动相对较弱的S4区域, 与前面的结果相反, 具体原因需要进一步基因丰度的调查与分析.金属抗性基因的分布集中于S1、S4区域, 研究中超过半数的抗Ag分离株分布于S1区域, 抗Ag分离株中有7株携带铜抗性基因pcoE, S4区域可能呈现出尽头的汇聚作用.抗性基因的分布与细菌的抗性表型相同, 并不是呈现出一种空间水平上递增的趋势, 人为活动的强弱决定了这种趋势.抗性基因空间上的分布与流行及水平遗传元件(质粒、转座子等)在其中所发挥的作用需要进一步的研究与探讨.
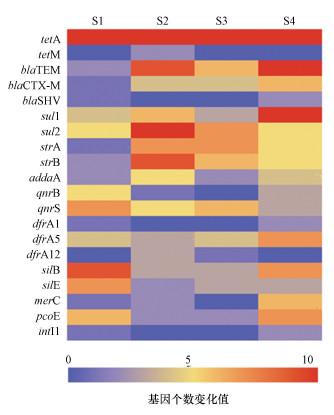 |
| 图 6 不同样点ARGs、MRGs和intI1的检出概况 Fig. 6 The detection of ARGs, HMRGs and intI1 from different sampling sites |
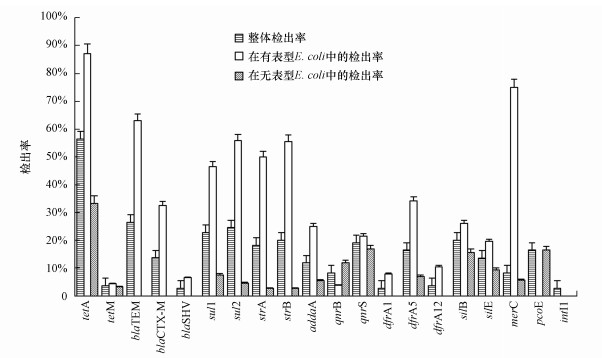 |
| 图 7 E. coli群体中ARGs、MRGs与intI1的检出率 Fig. 7 Detection precentage of ARGs, MRGs and intI1 |
如图 7所示, 在整个E. coli群体中, 四环素抗性基因tetA的检出率最高(56.4%), 其次是ESBLs基因blaTEM(26.4%)、磺胺抗性基因sul2(24.5%)和sul1(22.7%), 这几个基因一直以来也是抗性基因研究中检出频率比较高的基因(Xu et al., 2016;Deng et al., 2018);其他抗生素抗性基因与金属耐受性基因的检出率都在20%以下, 其中, 整合基因intI1的检出率仅为2.7%, 表明intI1对于该流域抗性基因的增殖与流行推动作用较小.在有抗生素抗性与金属耐受性的菌株中, 除了qnrB、pcoE基因, 其他抗性基因的检出率都高于在E. coli整个群体中的检出率.本研究中抗性基因与抗性表型的相关性分析表明, 抗性基因一定程度上决定着菌株对抗生素与金属的表型抗性, 如tetA与四环素耐抗性(r=0.54), blaTEM与ESBL抗性(r=0.64), sul2与磺胺抗性(r=0.58).与整体情况类似, tetA基因、blaTEM基因、sul2基因和sul1基因检出率仍然高于大多数抗性基因.
在无抗生素表型与金属耐受性的菌株中, 仍然有相关抗性基因的检出, 反映出抗性基因与抗性表型并不是一一对应的关系, 其中最典型的就是铜耐受性基因pcoE, 没有一株分离株具有铜耐受性, 但有18株菌中都检测出了该基因, 这也从侧面证实了基因的选择性表达.Hobman等(2015)曾报道一些细菌的质粒或染色体上的同源区段同时存在铜、银耐受性操纵子(pco、sil).本研究中18株具有pcoE基因的菌株, 有7株显示出对Ag的耐受性并具有Ag耐受性基因, 该结果表明可能存在共抗性, 具体的原因有待进一步分子机理方面的解析.
4 结论(Conclusions)本研究调查了中国寒区城市污水河道污泥中耐药菌的现状, 分离鉴定了污水河道中的455株肠杆菌, 检测了其中110株E. coli对17种抗生素、4种金属的耐药性, 结果显示, 69.1%的E. coli分离株对至少1种抗生素具有抗性, 45.5%的E. coli分离株为MDR菌株, 40.9%的E. coli分离株对Ag具有耐受性, 极少数菌株对Hg、Zn具有耐受性, 没有菌株对Cu具有耐受性;同时检测到了对13种抗生素具有抗性的E. coli DL27和对9种抗生素、2种金属具有耐受的E. coli DL35.研究表明, 河道污泥中不仅存在多重抗生素抗性E. coli, 而且存在同时具有抗生素抗性和金属耐受性的E. coli, 河道污泥是抗性细菌和抗性基因的储库, 对人类生产生活构成了潜在的巨大威胁.
Akinbowale A L, Peng H, Grant P, et al. 2007. Antibiotic and heavy metal resistance in motile Aeromonads and Pseudomonads from rainbow trout (Oncorhynchus mykiss) farms in Australia[J]. International Journal of Antimicrobial Agents, 30: 177-182. |
Al-Nabulsi A A, Osaili M, Shaker R R, et al. 2015. Effects of osmotic pressure, acid, or cold stresses on antibiotic susceptibility of Listeria monocytogenes[J]. Food Microbiology, 46: 154-160. |
Araujo S, Silva I A T, Tacalo M, et al. 2017. Characterization of antibiotic resistant and pathogenic Escherichia coli in irrigation water and vegetables in household farms[J]. International Journal of Food Microbiology, 257: 192-200. |
Asiani K.2016.Biochemical and biophysical studies on sile from the sil silver resistance locus[D].Nottingham: the University of Nottingham
|
Berendonk T U, Manaia C M, Merlin C, et al. 2015. Tackling antibiotic resistance:The environmental framework[J]. Nature Reviews Microbiology, 13(5): 310-317. |
Davies J, Davies D. 2010. Origins and evolution of antibiotic resistance[J]. Microbiolgy and Molecular Biolgy Reviews, 74(3): 417-433. |
Deng W W, Quan Y, Yang S Z, et al. 2018. Antibiotic resistance in Salmonella from retail foods of animal origin and its association with disinfectant and heavy metal resistance[J]. Microbial Drug Resistance, 24: 782-791. |
Dierikx C M, Van Duijkeren E, Schoormans A H W, et al. 2012. Occurrence and characteristics of extended-spectrum-lactamase- and AmpC-producing clinical isolates derived from companion animals and horses[J]. Journal of Antimicrobial Chemotherapy, 67(6): 1368-1374. |
Divya S P, Hatha A A M. 2019. Screening of tropical estuarine water in south-west coast of India reveals emergence of ARGs-harboring hypervirulent Escherichia coli of global significance[J]. International Journal of Hygiene and Environmental Health, 222(2): 235-248. |
Edberg S C, Rice E W, Karlin R J, et al. 2000. Escherichia coli:The best biological drinking water indicator forpublic health protection[J]. Journal of Applied Microbiology, 88(S1): 106S-116S. |
Ferro G, Guarino F, Cicatelli A, et al. 2017. β-lactams resistance gene quantification in an antibiotic resistant Escherichia coli water suspension treated by advanced oxidation with UV/H2O2[J]. Journal of Hazardous Materials, 323: 426-433. |
Goldstein C, Lee M D, Sanchez S, et al. 2001. Incidence of class 1 and 2 integrases in clinical and commensal bacteria from livestock, companion animals, and exotics[J]. Antimicrobial Agents and Chemotherapy, 45: 723-726. |
Grape M, Motakefi A, Pavuluri S, et al. 2007. Standard and real-time multiplex PCR methods for detection of trimethoprim resistance dfr genes in large collections of bacteria[J]. Clinical Microbiology and Infection, 13(11): 1112-1118. |
Hobman J L, Crossman L C. 2015. Bacterial antimicrobial metal ion resistance[J]. Journal of Medical Microbiology, 64: 471-497. |
Hong B, Ba Y B, Niu L, et al. 2018. A comprehensive research on antibiotic resistance genes in microbiota of aquatic animals[J]. Frontiers in Microbiology, 9: 1617. |
Huang Y H, Liu Y, Du P P, et al. 2019. Occurrence and distribution of antibiotics and antibiotic resistant genes in water and sediments of urban rivers with black-odor water in Guangzhou, South China[J]. Science of the Total Environment, 670: 170-180. |
Hu Q, Dou M N, Qi H Y, et al. 2017. Detection, isolation, and identification of cadmium-resistant bacteria based on PCR-DGGE[J]. Journal of Environmental Sciences, 19(9): 1114-1119. |
Ibrahim D R, Dodd C E, Hobman J L, et al. 2016. Multidrug resistant, extended spectrum β-lactamase (ESBL)-producing Escherichia coli isolated from a dairy farm[J]. FEMS Microbiology Ecology, 92: fiw013. |
李彬辉, 许燕滨, 赵欣欣, 等. 2012. 养殖废水中抗生素与金属交叉抗性微生物的筛选及其抗性研究[J]. 中国农学通报, 28(11): 103-107. |
Liebert C A, Wireman J, Smith T, et al. 1997. Phylogeny of mercury resistance (mer) operons of gram-negative bacteria isolated from the fecal flora of primates[J]. Applied and Environmental Microbiology, 63(3): 1066-1076. |
Lien T Q, Lan P T, Chuc N T K, et al. 2017. Antibiotic resistance and antibiotic resistance genes in Escherichia coli isolates from hospital wastewater in Vietnam[J]. International Journal of Environmental Research and Public Health, 14(7): 699. |
Matyar F, Kaya A, Dincer S. 2008. Antibacterial agents and heavy metal resistance in Gram-negative bacteria isolated from seawater, shrimp and sediment in Iskenderun Bay, Turkey[J]. Science of the Total Environment, 407(1): 279-285. |
Miller J H, Novak J T, Knocke W R, et al. 2014. Elevation of antibiotic resistance genes at cold temperatures:implications for winter storage of sludge and biosolids[J]. Letters in Applied Microbiology, 59(6): 587-593. |
Nelson M L, Levy S B. 2011. The history of the tetracyclines[J]. Annals of the New York Academy of Sciences, 1241(1): 17-32. |
O'Neill J. 2016. Antimicrobial Resistance:Tackling a crisis for the health and wealth of nations[J]. Review on Antimicrobial Resistance, (12): 1-16. |
Pal C, Asiani K, Hobman J L, et al. 2017. Metal resistance and its association with antibiotic resistance[J]. Advances in Microbial Physiology, 70: 261-313. |
Poma V, Mamani N, Iniguez V. 2016. Impact of urban contamination of the La Paz River basin on thermotolerant coliform density and occurrence of multiple antibiotic resistant enteric pathogens in river water, irrigated soil and fresh vegetables[J]. SpringerPlus, 5(1): 499. |
Pourcher A M, Jadas-Hecart A, Cotinet P, et al. 2014. Effect of land application of manure from enrofloxacin-treated chickens on ciprofloxacin resistance of Enterobacteriaceae in soil[J]. Science of the Total Environment, 482: 269-275. |
Qiao M, Ying G G, Singer A C, et al. 2018. Review of antibiotic resistance in China and its environment[J]. Environment International, 110: 160-172. |
Ranjbar R, Sami M. 2017. Genetic investigation of bata-lactam associated antibiotic resistance among Escherichia coli strains isolated from water sources[J]. The Open Microbiology Journal, 11(1): 203-210. |
Saeb A T M, Al-Rubeaan K A, Abouelhoda M. 2017. Genome sequencing and analysis of the first spontaneous Nanosilver resistant bacterium Proteus mirabilis strain SCDR1[J]. Antimicrobial Resistance and Infection Control, 6(1): 119. |
Sah S, Bordoloi P, Vijaya D, et al. 2018. Simple and economical method for identification and speciation of Staphylococcus epidermidis and other coagulase negative Staphylococci and its validation by molecular methods[J]. Journal of Microbiological Methods, 149: 106-119. |
Sanchez G V, Babiker A, Master R N, et al. 2016. Antibiotic resistance among urinary isolates from female outpatients in the United States in 2003 and 2012[J]. Antimicrobial Agents and Chemotherapy, 60(5): 2680-2683. |
邵碧英, 陈彬, 汤敏英, 等. 2007. 沙门氏菌DNA提取及PCR反应条件的优化[J]. 食品科学, 28(7): 331-334. |
Sharma V K, Johnson N, Cizmas L, et al. 2016. A review of the influence of treatment strategies on antibiotic resistant bacteria and antibiotic resistance genes[J]. Chemosphere, 150: 702-714. |
苏建强, 黄福义, 朱永官. 2013. 环境抗生素抗性基因研究进展[J]. 生物多样性, 21(4): 481-487. |
Son D I, Aleta P, Park M, et al. 2018. Seasonal changes in antibiotic resistance genes in rivers and reservoirs in South Korea[J]. Journal of Environmental Quality, 47(5): 1079-1085. |
Taylor N G, Verner-Jeffreys D W, Baker-Austin C. 2011. Aquatic systems:maintaining, mixing and mobilising antimicrobial resistance?[J]. Trends in Ecology and Evolution, 26(6): 278-84. |
Titilawo Y, Obi L, Okoh A. 2015. Antimicrobial resistance determinants of Escherichia coli isolates recovered from some rivers in Osun State, South-Western Nigeria:Implications for public health[J]. Science of the Total Environment, 523: 82-94. |
解海.2003.马家沟河流域综合治理方案与措施的研究[D].哈尔滨: 哈尔滨工业大学 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y564169
|
Xu Y, Guo C S, Luo Y, et al. 2016. Occurrence and distribution of antibiotics, antibiotic resistance genes in the urban rivers in Beijing, China[J]. Environment Pollutation, 213: 833-840. |
杨芬, 巢波, 杨万红, 等. 2017. 一株抗生素和金属交叉抗性菌的筛选及抗性研究[J]. 环境工程学报, 11(2): 1287-1292. |
张博.2013.城市污水中肠杆菌的抗生素耐药性和多种耐药基因的检测[D].哈尔滨: 东北农业大学 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2295355
|
Zhang Q Q, Ying G G, Pan C G, et al. 2015. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environment Science and Technology, 49(11): 6772-6782. |
 2020, Vol. 40
2020, Vol. 40


