2. 中国科学院生态环境研究中心, 环境模拟与污染控制国家重点联合实验室, 北京 100085;
3. 中国科学院生态环境研究中心, 水污染控制实验室, 北京 100085;
4. 中国科学院大学, 北京 100049;
5. 江西省科学院, 能源研究所, 南昌 330096
2. State Key Joint Laboratory of Environmental Simulation and Pollution Control, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
3. Laboratory of Water Pollution Control, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
4. University of Chinese Academy of Sciences, Beijing 100049;
5. Institute of Energy, Jiangxi Academy of Sciences, Nanchang 330096
厌氧氨氧化以亚硝酸盐为电子受体氧化氨氮生成氮气(Kartal et al., 2011), 这一过程由浮霉菌门厌氧氨氧化细菌完成(Tang et al., 2017), 在处理富含氨氮废水方面具有节约能耗等优势(Lawson et al., 2017).目前已报道的厌氧氨氧化细菌主要有Kuenenia、Brocadia、Anammoxoglobus、Jettenia及Scalindua, 但尚未发现纯培养得到的厌氧氨氧化细菌(Kartal et al., 2013; Lawson et al., 2017).尽管运行方式及水质组成不同, 大多数厌氧氨氧化生物反应器存在一个由Chloroflexi、Chlorobi、Proteobacteria、Acidobacteria及Bacteroidetes组成的核心微生物群落(Speth et al., 2016).以往研究表明, 这个核心微生物群落在底物及代谢方面与厌氧氨氧化细菌存在交互营养共生关系(Zhao et al., 2018), 如在无外碳源的条件下, 异养菌可降解厌氧氨氧化细菌分泌的胞外蛋白、多肽及细胞碎片(Kindaichi et al., 2012), 厌氧氨氧化细菌Brocadia sp.还为异养菌提供B族维生素(Lawson et al., 2017);Septh等研究发现, Chlorobi可以将厌氧氨氧化细菌Brocadia sp.产物硝酸盐还原成亚硝酸盐然后重新被厌氧氨氧化细菌利用, 提高了氮去除效率(Speth et al., 2016);厌氧氨氧化细菌缺乏固定CO2所需的亚钼嘌呤(MOCO)及叶酸合成途径但存在相关的转运体, 微生物群落中Armatimonadetes和Proteobacteria合成分泌MOCO及叶酸(Phelan et al., 2011; Seth et al., 2014);异养菌分泌的胞外多糖有利于厌氧氨氧化细菌的聚集(Jennings et al., 2015; Li et al., 2017).
在厌氧氨氧化细菌富集过程中, 这个核心微生物群落对厌氧氨氧化细菌的活性及生长有很大影响(Zhao et al., 2018).以往研究多关注厌氧氨氧化细菌的生态生理学(Kartal et al., 2013; Ali et al., 2014)或者与主要的自养硝化细菌的相互关系(Park et al., 2015), 对厌氧氨氧化生物反应器中异养菌的作用还缺乏认识.本研究采用实验室规模UASB逐步富集培养厌氧氨氧化细菌, 在培养过程中采集微生物样品, 采用分子生物学方法与传统微生物培养法等手段考察富集启动过程中功能基因、微生物群落组成、共生微生物种类及功能基因丰度等变化, 以菌群演替、共生关系等为突破口, 为厌氧氨氧化细菌快速富集、脱氮性能提升提供科学支持.
2 材料与方法(Materials and methods) 2.1 实验装置与接种污泥课题组前期采用UASB对厌氧氨氧化细菌进行富集培养(陈彦霖等, 2018).该UASB的有效容积为3 L, 试验过程中温度控制在(30±2) ℃, 通过蠕动泵控制连续进水、排水, 进水经过氮吹除氧后, 处于低溶解氧(<0.1 mg · L-1)状态.反应器接种泥为储存超过3个月、脱氮效果恶化(TN去除率小于5%)的厌氧氨氧化污泥, 接种泥的MLSS为2.575 g · L-1, MLVSS为1.63 g · L-1, VSS/SS比值为0.63.
2.2 实验设计与运行效果实验用水为人工合成的模拟废水, 其中, NH4+-N和NO2--N分别由NH4Cl和NaNO2按需配制, 其它组分包括:KH2PO4 25 mg · L-1、CaCl2 120 mg · L-1、MgSO4·7H2O 260 mg · L-1及微量元素Ⅰ、Ⅱ各1 mL · L-1, 微量元素配制参考文献(van de Graaf et al., 1996).
UASB启动过程进、出水浓度与脱氮效果如表 1所示.经过连续55 d的运行, 脱氮效果逐步提升, 运行至第55 d, NH4+-N和NO2--N平均出水浓度分别为9.29 mg · L-1和1.87 mg · L-1, 平均去除率分别为94.8%和97.5%;TN去除负荷从0.02 kg · m-3 · d-1提升至0.89 kg · m-3 · d-1, TN去除率从1.39%提升到79.62%, 脱氮效果可参考本课题组已发表论文(陈彦霖等, 2018).这表明在第55 d UASB反应器成功启动厌氧氨氧化反应, 并达到了稳定运行状态.
| 表 1 UASB反应器启动厌氧氨氧化过程的脱氨效果 Table 1 The performance of nitrogen removal during the start-up of anammox in a UASB reactor |
反应器运行过程中, 分别于第0 d(S1)、第20 d(S2)、第35 d(S3)、第55 d(S4)采集污泥样品.一份样品用于菌种培养与筛选, 一份污泥样品保存在-20 ℃冰箱中待用.
2.4 异养菌筛选、分离与鉴定采用传统培养法培养细菌, 制备R2A培养基, 进行梯度稀释(通过梯度稀释预实验确定最佳稀释倍数), 涂布, 恒温28 ℃倒置培养5 d, 挑取单个健壮的菌落, 进行单菌落菌液培养.
对培养得到的单菌落进行PCR扩增得到16S rRNA, PCR引物为27F(5′-AGAGTTTGATCMTGG CTCAG-3′)和1492R(5′-TACGGYTACCTTGTTACG ACTT-3′).菌落PCR的体系如下:12.5 μL的Taq MIX, 10.5 μL的ddH2O, 1 μL的菌液, 1 μL的上下游引物.菌落PCR程序如下:95 ℃预变性10 min;95 ℃变性45 s, 55 ℃退火30 s, 72 ℃延伸1 min, 共循环35次.
通过琼脂糖凝胶电泳鉴定16S rRNA菌落PCR产物的大小.结果显示, PCR扩增条带大小为1.5 kb左右, 与理论16S rRNA片段大小一致.获得的PCR产物利用传统的Sanger进行测序后, 通过Blast与NCBI数据库比对, 进行菌种鉴定.
2.5 DNA提取、基因扩增及高通量测序污泥总DNA提取采用FastDNA® SPIN Kit for soil(MP Biomedicals, Santa Ana, CA, U.S)试剂盒, 具体按说明书操作;提取得到的DNA通过琼脂凝胶电泳进行定性检测, 经过分光光度计进行浓度、纯度检测, 最后将提取的DNA样品放置于-80 ℃冰箱保存.
针对细菌的16S rRNA的V4区, 使用通用引物515F(5′-GTGCCAGCMGCCGCGGTAA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)(Caporaso et al., 2011)进行PCR扩增, PCR反应条件跟体系与上文相同.PCR扩增产物经2%的琼脂糖凝胶检测, 使用凝胶回收试剂盒进行纯化, 并经过定量分析后, 构建MiSeq文库, 利用Illumina MiSeq测序平台完成测序.本研究测序工作交由上海生工生物测序公司完成.
2.6 定量PCR(qPCR)为了研究氮素循环关键功能基因在培养过程中的丰度变化情况, 使用qPCR定量分析氨单加氧酶基因(ammonium monooxygenase, amoA)、亚硝酸盐氧化还原酶(nitrite oxidoreductase type B, nxrB)、亚硝酸还原酶(nitrite reductase, nirS)和肼脱氢酶(hydrazine dehydrogenase, hzo), 引物序列见表 2.具体实验步骤参照文献(Wang et al., 2015).
| 表 2 定量PCR引物序列 Table 2 List of the primers for qPCR |
高通量测序得到的原始数据经过QC质量控制, 去除嵌合体及非特异性扩增序列.将所得序列与NCBI 16S rRNA数据库进行Blast比对, 将相似度高于97%的序列使用Usearch软件进行OTU聚类.使用mothur计算α-多样性指数Shannon、Simspon、ACE1、Chao1指数和覆盖率(Coverage).采用ggplot2及ggalluvial绘制门水平物种丰度冲积条形图, 采用Circos Table Viewer在线工具绘制属水平物种丰度弦图.相关性分析采用Spearman相关性分析(IBM, USA), 微生物丰度具有显著正相关(p<0.05), 采用网络结构分析并绘图(Gephi 0.9.2).采用PICRUSt2对16S rRNA扩增子进行功能预测, 预测结果基于KEGG数据库, 并采用ggplot2对KEGG第一、第二层级进行绘图.
3 结果与讨论(Results and discussion) 3.1 关键功能基因的定量分析采用qPCR分析启动过程中amoA、nxrB、nirS和hzo的丰度, 结果如图 1所示, 除amoA丰度从接种泥的1.43%下降到第55 d的0.35%之外, nxrB、nirS、hzo丰度分别从接种泥的0.89%、1.01%、2.33%上升到第55 d的1.73%、1.45%、37.65%.amoA丰度下降是由于本研究采用UASB在厌氧条件下富集厌氧氨氧化细菌, 厌氧环境下AOB的生长受到抑制.nxrB基因丰度略有上升, nxrB是Nitrospira代表性功能基因, Nitrospira对DO具有较强的底物亲和力, 这是因为AOB和NOB的溶解氧半饱和常数分别为1.14和0.16 mg · L-1(Regmi et al., 2014).nirS由反硝化细菌、厌氧氨氧化细菌携带, 其基因丰度出现了小幅上升, 主要是由于厌氧氨氧化作用的提升, 同时也表明厌氧氨氧化反应器中存在厌氧氨氧化细菌和其他异养反硝化菌的相互作用.厌氧氨氧化细菌的功能基因hzo丰度显著增加, 这表明厌氧氨氧化细菌在反应器中成功富集, 这与反应器的脱氮效果一致(表 1).如图 2所示, 进水氮负荷的增加是nirS、hzo丰度提高的重要原因之一.nirS丰度随着进水氮负荷对数性增加, hzo丰度随着进水氮负荷指数性增加, 而Pearson相关性分析表明, nirS丰度与氮去除负荷呈线性相关(p<0.05).nirS是决定脱氮效果的关键因素, 反硝化菌在脱氮效果中也发挥着一定的作用.
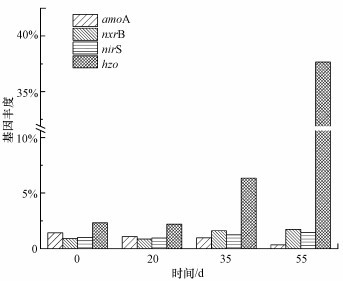 |
| 图 1 功能基因的定量分析 Fig. 1 The quantitative analysis of functional gene |
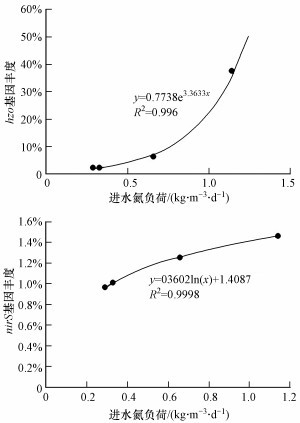 |
| 图 2 进水氮负荷与hzo、nirS功能基因丰度的关系 Fig. 2 Relationship between nitrogen load of influent and abundance of hzo and nirS functional genes |
如图 3所示, 第20 d, 异养菌的数量相较于接种泥出现了轻微下降, 从1×10(6.35±0.51) CFU · mL-1下降到1×10(6.04±0.14) CFU · mL-1, 说明启动初期环境条件的改变不利于一些异养菌的生长.第35 d, 异养菌数量上升到1×10(6.25±0.33) CFU · mL-1, 说明适应环境的异养菌生长得到恢复.第55 d, 异养菌数量上升到1×10(6.28±0.21) CFU · mL-1, 此时已恢复到接种泥时期的异养菌水平.整个富集过程中异养菌的数量未发生明显变化.
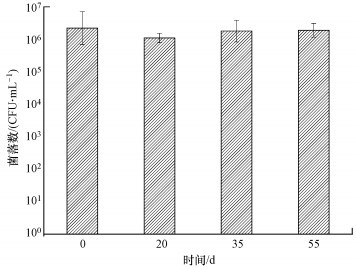 |
| 图 3 异养菌数量变化 Fig. 3 Changes in the number of heterotrophic bacteria |
筛选鉴定的异养菌主要属于变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)(表 4), 它们是厌氧氨氧化系统常见的两大门类.接种污泥鉴定的异养菌与食酸菌属(Acinetobacter sp.)、丛毛单胞菌属(Comamonas sp.)、芽孢杆菌属(Bacillus sp.)等最相似.食酸菌属和芽孢杆菌属是重要的异养硝化-好氧反硝化菌(Xia et al., 2020), 这类细菌能够进行同时硝化反硝化过程, 可以将异养硝化的产物作为好氧反硝化菌的底物(Zhang et al., 2019b; Xia et al., 2020);丛毛单胞菌属(Comamonas sp.)可以利用细菌分泌的胞外多糖进行好氧反硝化(张千等, 2018; Su et al., 2020).从反应器启动到第55 d, 反应器运行稳定, 异养菌的菌群组成过渡到厚壁菌门, 主要有类芽孢杆菌属(Paenibacillus sp.)和芽孢杆菌属(Bacillus sp.).类芽孢杆菌为兼性厌氧菌, 主要是适应环境而留在系统中.芽孢杆菌在剪切力的刺激下向胞外分泌胞外多糖, 促进厌氧氨氧化细菌的团聚过程(Chang et al., 2020), 由胞外多糖形成的凝胶网状结构对微生物聚集至关重要(Li et al., 2017).综上所述, 随着厌氧氨氧化菌的富集成功, 异养菌的数量并未发生明显下降, 但异养菌的菌群从变形菌门过渡到厚壁菌门, 且芽孢杆菌分泌的胞外多糖有利于厌氧氨氧化细菌聚集.
| 表 4 异养菌鉴定结果 Table 4 Identification of heterotrophic bacteria |
微生物群落的α多样性如表 5所示, 污泥样品的覆盖率都为99%, 可确保测序结果代表污泥样品中的微生物群落组成.OTU数目先升高后降低, 推测接种泥起始阶段为环境适应期, 微生物群落发生了较大的变化, 度过环境适应期后淘汰掉一些不适应的微生物, 因此出现OTU先升高后降低的趋势.Chao1和Ace指数反映物种的丰富度, Shannon和Simpson指数反映物种的多样性.它们均呈现先升高后降低的趋势, 这表示启动过程中的α-多样性先升高后降低, 说明启动过程中逐渐富集了功能更加专一性的菌群, 这与以往的研究结果一致.
| 表 5 微生物群落的α多样性分析 Table 5 α diversity analysis of microbial community |
门水平上的微生物群落结构如图 4所示, 变形菌门(Proteobacteria)、浮霉菌门(Planctomycetes)、Ignavibacteriae、拟杆菌门(Bacteroidetes)和绿弯菌门(Chloroflexi)为优势菌门.许多研究表明, 在厌氧氨氧化反应体系, 浮霉菌门、变形菌门、Ignavibacteriae、绿弯菌门、Armatimonadetes存在共存现象(Mardanov et al., 2019; Zhao et al., 2018).
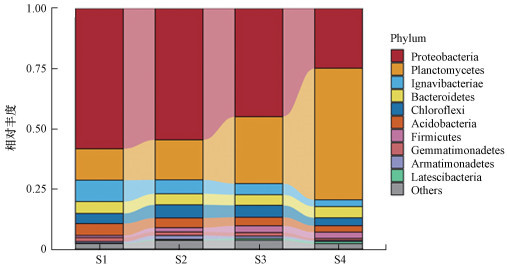 |
| 图 4 门水平群落结构变化 Fig. 4 Evolution of microbial community structure at phylum level |
在启动过程中, 厌氧氨氧化细菌所属的浮霉菌门相对丰度不断升高, 接种泥中相对丰度为13.07%, 第55 d已经上升到54.70%, 成为反应器中第一优势菌门.变形菌门相对丰度从起始的58.31%下降到第55 d的24.81%, 降为反应器中的第二优势菌门, 但仍在系统中占据着重要地位.已有研究表明, 一些合成途径复杂的次生代谢物可由变形菌门合成并转运至厌氧氨氧化细菌内, 这种方式更能节约能量(Phelan et al., 2011; Zhao et al., 2018).第35~55 d, 反应器的脱氮效果并没有出现明显升高, 推测系统中存在一定的变形菌门更有利于厌氧氨氧化细菌的活性.
Ignavibacteriae作为第三优势菌门, 其相对丰度在启动过程中明显减少.Ignavibacteriae在进化关系上与拟杆菌门和绿弯菌门相近, 是一种异养兼性厌氧菌, 广泛存在于厌氧生物反应器, 它能够利用细胞外肽将硝酸盐还原成亚硝酸盐, 被认为是蛋白降解剂(Mardanov et al., 2019; Zhang et al., 2019a).宏基因组分析表明, Ignavibacteriae可以与厌氧氨氧化细菌形成硝酸盐-亚硝酸盐循环, 提高脱氮效率(Mardanov et al., 2019).其丰度减少是因为厌氧氨氧化细菌增多, 厌氧氨氧化细菌代谢缓慢致使反应器中碳源减少不足以维持Ignavibacteriae代谢.
已有研究表明, 绿弯菌门通常占据厌氧氨氧化反应器的主要成分, 且宏基因组和FISH分析结果表明, 大多数丝状菌属于绿弯菌门(Kragelund et al., 2011; Speirs et al., 2017).由于呈丝状结构, 这些丝状菌在厌氧氨氧化细菌中通常充当骨架结构, 参与生物膜的形成(Zhao et al., 2018).绿弯菌门在厌氧氨氧化反应器菌群中还参与死细胞的降解, 或者参与胞外基质的合成与降解(Speth et al., 2016).因此, 含有一定的绿弯菌门对厌氧氨氧化细菌的生长更加有利.
3.3.3 属水平的群落结构变化反应器中属水平上的物种丰度变化如图 5所示, 厌氧氨氧化细菌主要是Candidatus Brocadia和Candidatus Kuenenia两种, 它们都属于浮霉菌门, 分别是r-策略和k-策略厌氧氨氧化细菌(Oshiki et al., 2016).反应器运行至第55 d, Candidatus Brocadia丰度从起始的7.56%下降到3.87%, 而Candidatus Kuenenia丰度则从4.75%上升到48.77%, 取代Candidatus Brocadia成为优势菌属.已有研究发现, Candidatus Kuenenia比Candidatus Brocadia能耐受更高水平的亚硝态氮所带来的抑制作用(Oshiki et al., 2016);也有研究表明, 高浓度底物会激活Candidatus Kuenenia膜表面的呼吸蛋白, 促进Candidatus Kuenenia生长(Zhang et al., 2020).这些都是Candidatus Kuenenia在反应器中大量增殖的原因.
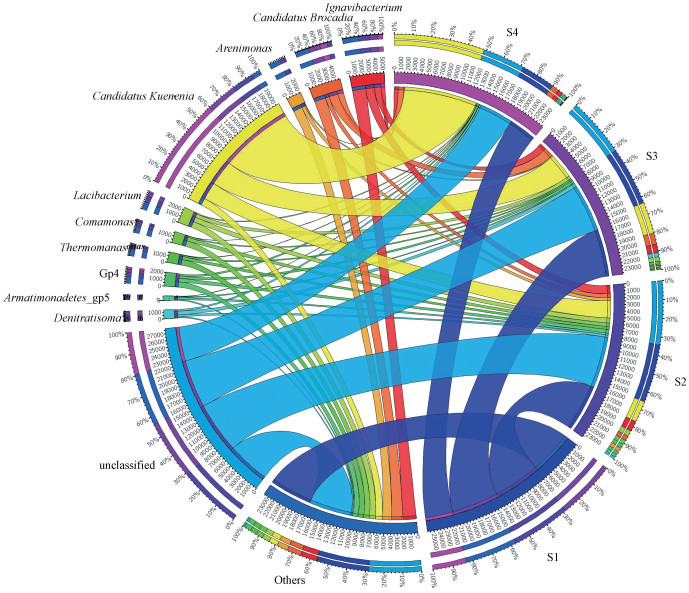 |
| 图 5 属水平群落结构弦图 Fig. 5 Microbial community structure map at genera level |
高通量测序获得的菌属信息基本覆盖了培养法获得的微生物菌种类型, 如Acinetobacter、Comamonas, 大多数无法培养型细菌的变化也可深入分析, 结果如图 5所示.在厌氧氨氧化反应器的菌群中, 大多数异养菌成员都参与N循环过程, 并且可以进行部分反硝化(Speth et al., 2016; Lawson et al., 2017).属于Chlorobi的Ignavibacterium在反应器中丰度一直降低, Chlorobi相关细菌能够将厌氧氨氧化细菌产生的胞外蛋白降解成氨基酸, 从而影响细胞聚集(Zhao et al., 2018).Denitratisoma是典型的反硝化菌, 其丰度从0.72%略上升到1.07%, 说明系统中存在一定的反硝化作用.属于Armatimonadetes的Armatimonadetes_gp5丰度呈现先升高后降低的现象, 它通过合成次级代谢物质影响厌氧氨氧化细菌的活性与生长(Seth et al., 2014; Zhao et al., 2018).其他具有部分反硝化的异养菌丰度一直下降, 这也与异养菌菌种鉴定结果相符.
3.4 微生物网络分析本研究对与厌氧氨氧化细菌存在显著正相关关系的菌属进行了网络结构分析, 其中包含培养法和高通量测序法获得的主要菌种类型.如图 6所示, 与Candidatus Kuenenia存在显著正相关的异养菌菌属包括Ornatilinea、Latescibacteria_genera_incertae_sedis、Ignavibacterium和Gp4, 其中, Ignavibacterium和Gp4与多种异养菌存在显著正相关关系(p<0.05), 这些细菌主要分布在Proteobacteria门.因此, 这些细菌通过直接相关、间接相关与厌氧氨氧化细菌存在共生关系(Zhao et al., 2018).Ignavibacterium和Gp4两个菌属分别属于Ignavibacteriae和Armatimonadetes两个菌门, 这两个菌门在最近的研究中被证明与厌氧氨氧化细菌存在交互营养共生关系(Zhao et al., 2018).网络图中Ornatilinea和Thermomonas是典型丝状菌(Busse et al., 2002; Podosokorskaya et al., 2013), 以往研究表明, 厌氧氨氧化反应器中存在适量丝状菌更有利于厌氧氨氧化细菌的生长, 它们可以充当厌氧氨氧化细菌的骨架及通过部分反硝化参与N循环过程(Chu et al., 2015; Mardanov et al., 2019).
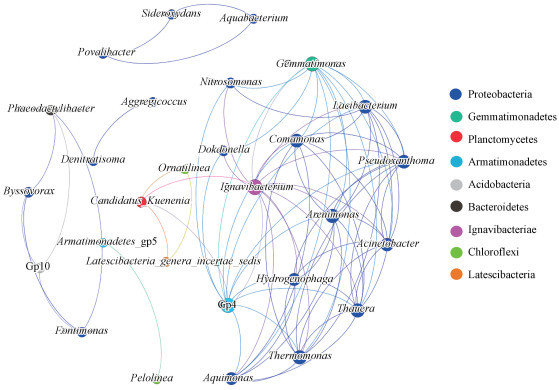 |
| 图 6 微生物网络分析 Fig. 6 Network of microbial community |
基于PICRUSt对微生物群落的功能预测,31条KEGG途径在微生物培养过程中的丰度分布如图 7所示.主要分为细胞过程(Cellular Processes)、环境信息处理(Environmental Information Processing)、遗传信息处理(Genetic Information Processing)、新陈代谢(Metabolism)及生物体系统(Organismal Systems)代谢通路.代谢潜能的变化与微生物群落的变化相关, 与代谢相关的预测路径占比最大, 氨基酸代谢及碳水化合物代谢途径丰度最高.已有研究发现异养菌缺乏氨基酸合成途径但可通过降解厌氧氨氧化细菌分泌的胞外蛋白及多肽得到氨基酸(Ni et al., 2012; Liu et al., 2016), Kindaichi等用碳同位素示踪实验证明了异养菌可利用厌氧氨氧化细菌的细胞碎片(Kindaichi et al., 2012).与物质信息交流的膜运输途径活跃, 说明富集过程中物质运输活跃, 与外界物质交换频繁, 如异养菌通过分泌次生代谢物质MOCO及叶酸并转运至厌氧氨氧化细菌内(Seth et al., 2014; Zhao et al., 2018), 厌氧氨氧化细菌通过分泌胞外蛋白或者直接分泌氨基酸给异养菌(Lawson et al., 2017; Zhao et al., 2018), 厌氧氨氧化细菌为异养菌提供合成多糖的原料UDP-ManNAC、CDP-Glc(Zhao et al., 2018)及NO2--NO3-在异养菌和厌氧氨氧化细菌之间循环(Speth et al., 2016)等过程.这些在群落成员之间发生的膜运输具有节约能量(Seth et al., 2014)、减少反硝化中间物(如NO2-)的毒害作用(Elin et al., 2016)等优点.
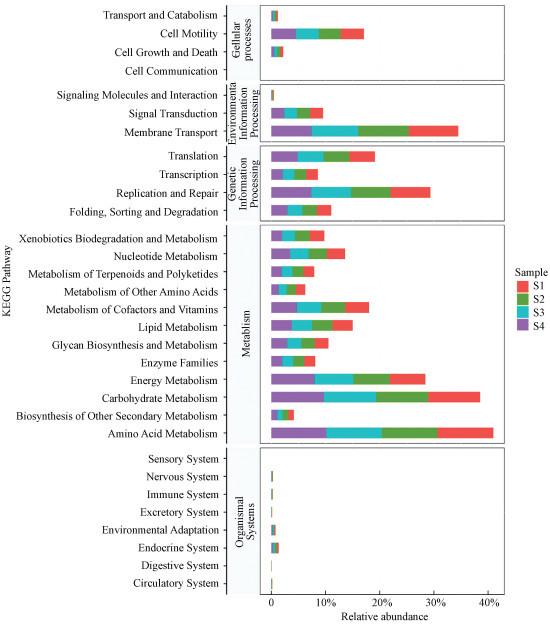 |
| 图 7 基于KEGG通路功能预测的相对丰度 Fig. 7 Relative abundance of functional prediction in KEGG pathway |
1) 厌氧氨氧化富集过程中主要的脱氮功能基因得到富集, hzo丰度达到了37.65%, 异养菌发生演替, 逐渐从变形菌门过渡到厚壁菌门.
2) 厌氧氨氧化富集过程中微生物群落主要由变形菌门(Proteobacteria)、浮霉菌门(Planctomycetes)、Ignavibacteriae、拟杆菌门(Bacteroidetes)和绿弯菌门(Chloroflexi)组成, α-多样性先升高后降低, 厌氧氨氧化细菌Candidatus Kuenenia成功富集, 丰度达到了48.77%, 异养菌多具有部分反硝化、分泌次生代谢物质及促进微生物团聚等功能.
3) 厌氧氨氧化富集过程中异养菌会影响厌氧氨氧化细菌的活性和生长, 异养菌Ignavibacterium、Gp4与厌氧氨氧化细菌Candidatus Kuenenia呈显著正相关(p<0.05), KEGG功能预测表明微生物群落碳代谢、氨基酸代谢及物质转运活跃, 异养菌在某种程度上对厌氧氨氧化细菌进行了补充.
Ali M, Oshiki M, Awata T, et al. 2014. Physiological characterization of anaerobic ammonium oxidizing bacterium "Candidatus Jettenia caeni"[J]. Environmental Microbiology, 17: 2172-2189. |
Busse H J, Kmpfer P, Moore E R B, et al. 2002. Thermomonas haemolytica gen nov., sp.nov., a γ-proteobacterium from kaolin slurry[J]. International Journal of Systematic and Evolutionary Microbiology, 52: 473-483. DOI:10.1099/00207713-52-2-473 |
Caporaso J G, Lauber C L, Walters W A, et al. 2011. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences, 108(S1): 4516-4522. |
陈彦霖, 隋倩雯, 姜黎安, 等. 2018. 厌氧氨氧化菌快速富集培养及微生物机制解析[J]. 中国给水排水, 34(13): 26-31. |
Chang J, He X, Bai X, et al. 2020. The impact of hydrodynamic shear force on adhesion morphology and biofilm conformation of Bacillus sp.[J]. Ocean Engineering, 197: 106860. DOI:10.1016/j.oceaneng.2019.106860 |
Lilja E E, Johnson D R. 2016. Segregating metabolic processes into different microbial cells accelerates the consumption of inhibitory substrates[J]. ISME Journal, 10: 1568-1578. DOI:10.1038/ismej.2015.243 |
Jennings L K, Storek K M, Ledvina H E, et al. 2015. Pel is a cationic exopolysaccharide that cross-links extracellular DNA in the Pseudomonas aeruginosa biofilm matrix[J]. Proceedings of the National Academy of Sciences, 112: 11353. DOI:10.1073/pnas.1503058112 |
Kartal B, De Almeida N M, Maalcke W J, et al. 2013. How to make a living from anaerobic ammonium oxidation[J]. FEMS Microbiology Reviews, 37: 428-461. DOI:10.1111/1574-6976.12014 |
Kartal B, Maalcke W, Almeida N, et al. 2011. Molecular mechanism of anaerobic ammonium oxidation[J]. Nature, 479: 127-130. DOI:10.1038/nature10453 |
Kindaichi T, Yuri S, Ozaki N, et al. 2012. Ecophysiological role and function of uncultured Chloroflexi in an anammox reactor[J]. Water Science and Technology, 66(12): 2556-61. DOI:10.2166/wst.2012.479 |
Kragelund C, Thomsen T R, Mielczarek A T, et al. 2011. Eikelboom's morphotype 0803 in activated sludge belongs to the genus Caldilinea in the phylum Chloroflexi[J]. FEMS Microbiology Ecology, 76: 451-462. DOI:10.1111/j.1574-6941.2011.01065.x |
Lawson C E, Wu S, Bhattacharjee A S, et al. 2017. Metabolic network analysis reveals microbial community interactions in anammox granules[J]. Nature Communications, 8: 15416. DOI:10.1038/ncomms15416 |
Li X, Luo J, Guo G, et al. 2017. Seawater-based wastewater accelerates development of aerobic granular sludge:A laboratory proof-of-concept[J]. Water Research, 115: 210-219. DOI:10.1016/j.watres.2017.03.002 |
Liu Y, Sun J, Peng L, et al. 2016. Assessment of heterotrophic growth supported by soluble microbial products in anammox biofilm using multidimensional modeling[J]. Scientific Reports, 6: 27576. DOI:10.1038/srep27576 |
Lu H, Chandran K, Stensel D. 2014. Microbial ecology of denitrification in biological wastewater treatment[J]. Water Research, 64: 237-254. DOI:10.1016/j.watres.2014.06.042 |
Mardanov A, Beletsky A, Ravin N, et al. 2019. Genome of a novel bacterium "Candidatus Jettenia ecosi" reconstructed from the metagenome of an anammox bioreactor[J]. Frontiers in Microbiology, 10: 02442. DOI:10.3389/fmicb.2019.02442 |
Ni B J, Ruscalleda M, Smets B F. 2012. Evaluation on the microbial interactions of anaerobic ammonium oxidizers and heterotrophs in Anammox biofilm[J]. Water Research, 45: 4645-4652. |
Oshiki M, Satoh H, Okabe S. 2016. Ecology and physiology of anaerobic ammonium oxidizing bacteria[J]. Environmental Microbiology, 18: 2784-2796. DOI:10.1111/1462-2920.13134 |
Ma Y, Sundar S, Park H, et al. 2015. The effect of inorganic carbon on microbial interactions in a biofilm nitritation-anammox process[J]. Water Research, 70: 246-254. DOI:10.1016/j.watres.2014.12.006 |
Phelan V V, Liu W T, Pogliano K, et al. 2011. Microbial metabolic exchange——the chemotype-to-phenotype link[J]. Nature Chemical Biology, 8: 26. |
Podosokorskaya O A, Bonch-Osmolovskaya E A, Novikov A A, et al. 2013. Ornatilinea apprima gen nov., sp.nov., a cellulolytic representative of the class Anaerolineae[J]. International Journal of Systematic and Evolutionary Microbiology, 63: 86-92. DOI:10.1099/ijs.0.041012-0 |
Regmi P, Miller M W, Holgate B, et al. 2014. Control of aeration, aerobic SRT and COD input for mainstream nitritation/denitritation[J]. Water Research, 57: 162-171. DOI:10.1016/j.watres.2014.03.035 |
Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63: 4704. DOI:10.1128/AEM.63.12.4704-4712.1997 |
Schmid M C, Hooper A B, Klotz M G, et al. 2008. Environmental detection of octahaem cytochrome c hydroxylamine/hydrazine oxidoreductase genes of aerobic and anaerobic ammonium-oxidizing bacteria[J]. Environmental Microbiology, 10: 1462-2920. |
Seth E C, Taga M E. 2014. Nutrient cross-feeding in the microbial world[J]. Frontiers in Microbiology, 5: 350. |
Speirs L B M, Dyson Z A, Tucci J, et al. 2017. Eikelboom filamentous morphotypes 0675 and 0041 embrace members of the Chloroflexi:Resolving their phylogeny, and design of fluorescence in situ hybridisation probes for their identification[J]. FEMS Microbiology Ecology, 93(10). DOI:10.1093/femsec/fix115 |
Speth D R, In'T Zandt M H, Guerrero-Cruz S, et al. 2016. Genome-based microbial ecology of anammox granules in a full-scale wastewater treatment system[J]. Nature Communication, 7: 11172. DOI:10.1038/ncomms11172 |
Su J F, Yang S, Huang T L, et al. 2020. Enhancement of the denitrification in low C/N condition and its mechanism by a novel isolated Comamonas sp[J]. Environmental Pollution, 256: 113294. DOI:10.1016/j.envpol.2019.113294 |
Suzuki M, Taylor L, Delong E. 2000. Quantitative analysis of small-subunit rRNA genes in mixed microbial populations via 5'-Nuclease assays[J]. Applied and Environmental Microbiology, 66: 4605-4614. DOI:10.1128/AEM.66.11.4605-4614.2000 |
Tang C J, Duan C S, Yu C, et al. 2017. Removal of nitrogen from wastewaters by anaerobic ammonium oxidation (ANAMMOX) using granules in upflow reactors[J]. Environmental Chemistry Letters, 15: 311-328. DOI:10.1007/s10311-017-0607-5 |
Van De Graaf A A, Bruijn P, Robertson L, et al. 1996. Autotrophic growth of anaerobic ammonium-oxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology, 142: 2187-2196. DOI:10.1099/13500872-142-8-2187 |
Vanparys B, Spieck E, Heylen K, et al. 2007. The phylogeny of the genus Nitrobacter based on comparative rep-PCR, 16S rRNA and nitrite oxidoreductase gene sequence analysis[J]. Systematic and Applied Microbiology, 30: 297-308. |
Wang Y, Wang Y, Wei Y, et al. 2015. In-situ restoring nitrogen removal for the combined partial nitritation-anammox process deteriorated by nitrate build-up[J]. Biochemical Engineering Journal, 98: 127-136. |
Xia L, Li X, Fan W, et al. 2020. Heterotrophic nitrification and aerobic denitrification by a novel Acinetobacter sp ND7 isolated from municipal activated sludge[J]. Bioresource Technology, 301: 122749. |
Zhang D, Xu S, Antwi P, et al. 2019a. Accelerated start-up, long-term performance and microbial community shifts within a novel upflow porous-plated anaerobic reactor treating nitrogen-rich wastewater via ANAMMOX process[J]. RSC Advances, 9: 26263-26275. |
Zhang Q, Chen X, Luo W, et al. 2019b. Effects of temperature on the characteristics of nitrogen removal and microbial community in post solid-phase denitrification biofilter process[J]. International Journal of Environmental Research and Public Health, 16(22): 4466. |
Zhang T, Wei N, Wu G. 2020. Autotrophic nitrogen removal and potential microbial interactions in anammox systems with different ammonia and organic carbon concentrations[J]. Journal of Water Process Engineering, 37: 101493. |
Zhao Y, Liu S, Jiang B, et al. 2018. Genome-centered metagenomics analysis reveals the symbiotic organisms possessing ability to cross-feed with anammox bacteria in anammox consortia[J]. Environmental Science and Technology, 52: 11285-11296. |
张千, 吉芳英, 付旭芳, 等. 2018. 气水比对后置固相反硝化滤池工艺脱氮及微生物群落影响[J]. 环境科学, 39(7): 3297-3305. |
 2021, Vol. 41
2021, Vol. 41


