铜矿的开采及含铜杀虫剂的使用, 造成金属Cu在土壤中大量富集(Chen et al., 2014).作为生命体必需的微量元素, Cu是细胞中多种金属酶和蛋白质的组成部分, 参与了电子传递、氧化还原和其他重要反应(Cervantes et al., 1994; Cai et al., 2019).当土壤中Cu含量到达一定阈值时, 会对土壤生物的功能和质量造成有害且不可逆的影响(赵杰等, 2018; Cai et al., 2019).Choudhary等(2007)研究发现, 当Cu含量达到200 mg·kg-1时, 土壤中脲酶和硝酸还原酶的活性被抑制.微生物是土壤的重要组成部分, 参与了几乎所有的生物化学活动, 是土壤生态系统健康运行的基础(Smith et al., 2015).相较于动植物, 土壤微生物对重金属胁迫更加敏感(Jansen et al., 1994), 当土壤中Cu含量到达一定值时, 会影响土壤微生物的丰度、群落结构及功能, 进而导致土壤质量的下降和生态结构的破坏(Chen et al., 2018; Lin et al., 2019; 施昊坤等, 2020).目前, Cu污染对土壤微生物的影响已引起了人们的广泛关注.
在长期重金属胁迫下, 一些细菌通过表达重金属抗性基因(Heavy metal resistance genes, MRGs)来调控各种抗性机制(细胞壁生物吸附、不溶性盐的沉淀、氧化还原改变价态和细胞膜运输), 从而降低重金属对自身的毒害作用(Bouskill et al., 2007; Denef et al., 2010).Li等(2012)对Cu污染的稻田土壤研究发现, Cu和copA(可以将铜离子从细胞质挤出到周质空间)之间具有显著的正相关关系.MRGs的表达对重金属的迁移和累计具有重要意义, 使得重金属污染土壤中的细菌在土壤污染生物修复中具有极大的潜力(Umrania, 2006).利用细菌对重金属Cu污染土壤修复的前提是了解Cu污染下细菌群落结构和重金属抗性基因的变化, 并建立两者之间的联系.然而目前为止有关细菌重金属Cu抗性的研究主要集中在单一分离菌株上(Albarracín et al., 2005; Cayol et al., 2015), 整体探究重金属Cu污染下细菌群落结构及重金属抗性基因的变化相对较少.
本研究通过高通量测序和荧光定量PCR技术, 分析不同Cu浓度下细菌丰度、群落结构、多样性的变化及抗性基因的分布, 探究Cu浓度与抗性基因的相关性及细菌与抗性基因之间的关系, 从而深入了解随Cu浓度升高土壤中细菌群落结构及抗性基因的变化, 为重金属Cu污染评价及修复提供理论依据.
2 材料与方法(Materials and methods) 2.1 实验设计本研究的土壤样品于2019年6月采集自山西省太原市小店区孙家寨村(37°41.5′N, 112°32.2′E)荒废农田(已知该农田多年未施肥记录)的表层土壤(0~10 cm), 类型为褐土.将所采样品装入聚乙烯塑料保鲜袋中, 24 h内带回实验室, 挑选去除样品中大的植物碎片及石块沙砾, 过2 mm筛后充分混匀, 分为两部分, 一部分储存于4 ℃冰箱用于后续微宇宙实验, 一部分自然风干用于理化性质分析.土壤pH值采用电位法测定(水土比2.5:1), pH为7.4.土壤总氮(TN)采用凯氏定氮法测定, TN为0.94 g·kg-1.有机碳(SOC)采用高温外热重铬酸钾氧化-容量法测定, SOC为15.85 g·kg-1.土壤重金属Cu含量利用电感耦合等离子体质谱仪ICP-MS(Agilent 7700x, Agilent Technologies)测定, Cu含量为29.39 mg·kg-1.
设计微宇宙实验, 将CuCl2粉末与预处理好的土壤充分混匀, 设置7个浓度梯度, 分别为50、100、200、300、500、750和1000 mg·kg-1, 分别标记为Cu50、Cu100、Cu200、Cu300、Cu500、Cu750、Cu1000, 以原始土样为空白对照(CK), 每个浓度3个重复.每个浓度下称取50 g干土置于150 mL尿杯中, 调节土壤样品的充水孔隙度(WFPS)为60%, 培养过程中, 每2 d打开培养杯盖以保证有氧条件, 使用称重法以保持60% WFPS的土壤水分.将土壤样品置于恒温培养箱中, 在25 ℃黑暗条件下培养14 d后取样进行后续分析.
2.2 土壤样品总DNA提取和荧光定量PCR根据E.Z.N.A.Ⓡ soil试剂盒(Omega Bio-tek, Norcross, GA, U.S.)的说明流程提取总DNA, 利用1%的琼脂糖凝胶电泳确认总DNA的提取质量, 同时采用NanoDrop ND-1000微光分光光度计(Thermo Fisher Scientific, USA)测定浓度.采用iCycler iQ5荧光定量PCR仪(Bio-Rad, Herculers, CA, USA)对样本中16S rDNA的V3~V4区、10个MRGs和2个可移动遗传元件(Mobile Genetic Elements, MGEs)分别进行定量, 每个样本重复3次.16S rDNA的引物序列为338F:5′-ACTCCTACGGGAGGCAGCAG-3′和534R:5′-ATTACCGCGGCTGCTGG-3′, 扩增体系(10 μL)含5 μL的SYBR Premix Ex TaqTM(Takara Bio, Inc., Shiga, Japan)、0.5 μL前后引物(10 mmol·L-1)、1 μL稀释模板DNA和3 μL无菌水.扩增条件为:95 ℃预变性5 min;95 ℃预变性3 min, 95 ℃变性30 s, 58 ℃退火30 s, 共40个循环.MRGs包括4个Cu抗性基因(copA、copB、cueO和pcoA)和6个多金属抗性基因(czc、czcA、czcD、czcI、czcN和czcR), MGEs包括一个Ⅰ类整合酶基因(intⅠ1)和一个转座酶基因(tnpA).这些MRGs在荧光定量PCR检测中所用引物序列、目标基因片段大小及退火温度主要依据已发表的文献(Bouskill et al., 2007; Roosa et al., 2014; Chen et al., 2019; Chen et al., 2020), 详细引物序列见表 1.
| 表 1 qPCR引物序列表 Table 1 Target genes and primer sequences |
采用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTW-TCTAAT-3′)引物对16S rRNA基因的V3~V4可变区域进行PCR扩增.扩增体系为20 μL, 扩增条件为:95 ℃预变性3 min;95 ℃变性30 s, 53 ℃退火30 s, 72 ℃延伸45 s, 29个循环;72 ℃终延伸10 min.扩增产物纯化并测定浓度(NanoDrop)后在Illumina MiSeq平台上测序.在97%的相似水平上, 采用RDP classifier贝叶斯算法对获得的高质量序列中的OTU代表序列进行分类学分析, 并与Silva数据库(http://www.arb-silva.de)进行比对, 获得各分类学水平上的群落信息.本研究中, 细菌相对丰度大于1%的类群为优势类群.高通量测序和生信分析在美吉生物医药科技有限公司(上海)完成.
2.4 数据分析采用单因素方差分析(one-way ANOVA)比较不同Cu浓度土壤间细菌16S rDNA、MRGs、MGEs和群落多样性指数的差异. 采用Spearman相关分析确定Cu浓度、MRGs和MGEs之间的相关性(显著性阈值p < 0.05, p < 0.01, p < 0.001).采用Spearman秩相关, 构建相关矩阵分析确定MRGs和MGEs的潜在细菌宿主.数据采用Microsoft Excel 2013和SPSS 24.0进行处理和分析, 采用Microsoft Excel 2018对单因素方差分析可视化, 采用R 3.5.1实现相关系数的可视化, 采用Gephi 0.9.2实现网络分析可视化.
3 结果与分析(Results and analysis) 3.1 细菌丰度本研究通过定量PCR获得不同Cu浓度土壤中细菌的16S rDNA基因拷贝数(copies·g-1), 以此表征土壤中的细菌丰度(图 1).由图 1可知, 随着Cu浓度增加, 细菌丰度呈先升高后降低的趋势, 在Cu浓度为100 mg·kg-1时最高(8.02×109 copies·g-1), 而当Cu浓度≥500 mg·kg-1时, 细菌丰度显著低于CK(p < 0.05), 在Cu浓度为1000 mg·kg-1时最低(2.01×109 copies·g-1).
 |
| 图 1 不同Cu浓度土壤的细菌丰度 (不同小写字母表示各处理之间存在差异性差异, p < 0.05)) Fig. 1 The bacterial abundance in different Cu treatments |
对样品16S rDNA V3~V4可变区域测序, 经过数据过滤去除低质量序列, 进行最小样本序列数抽平, 每个样品平均得到22890个序列, 以97%相似度划分, 得到3137个OTUs, 共30个门、82个纲、213个目、373个科、694个属和1375个种.如图 2所示, 随着序列数目增加, 各处理的稀疏性曲线趋于平缓, 说明扩增序列可以真实反映各个土壤样品的细菌群落结构, 测序深度足够.
 |
| 图 2 测序结果的稀疏性曲线 Fig. 2 Rarefaction curves of high-throughput sequencing |
通过计算不同Cu污染土壤OTUs序列的Alpha多样性指数, 分析Cu污染对土壤细菌丰富度和多样性的影响.Ace指数和Chao1指数为群落丰富度指数, Shannon指数和Simpson指数为群落多样性指数(指数越高, 微生物多样性越低).结果显示(表 2), 当Cu浓度≤100 mg·kg-1时, 多样性指数呈上升的趋势, 在Cu浓度为100 mg·kg-1时多样性指数最大且显著大于CK处理(p < 0.05), 分别为3313.14± 97.62(Ace)、3494.71±123.80(Chao1)、6.27±0.55(Shannon)和0.010±0.001(Simpson). 而当Cu浓度≥500 mg·kg-1时, 多样性指数显著降低(p < 0.05), 在Cu浓度为1000 mg·kg-1时多样性指数最小, 分别为2257.97±85.13(Ace)、2173.28±162.04(Chao1)、4.67±0.45(Shannon)和0.051±0.009(Simpson).
| 表 2 细菌群落Alpha多样性指数 Table 2 Alpha diversity indexes of bacteria community |
细菌群落优势细菌门共有9个(图 3), 大约占细菌群落丰度的95%. 这些优势菌门分别为变形菌门(Proteobacteria, 23.81%~55.42%)、拟杆菌门(Bacteroidetes, 15.26%~33.15%)、放线菌门(Actinobacteria, 8.16%~14.81%)、酸杆菌门(Acidobacteria, 4.06%~17.09%)、绿弯菌门(Chloroflexi, 3.19%~8.61%)、厚壁菌门(Firmicutes, 0.54%~6.85%)、芽单胞菌门(Gemmatimonadetes, 1.69%~6.84%)、浮霉菌门(Planctomycetes, 0.51%~2.01%)和Deinococcus(0.017%~1.56%). 随着Cu浓度的增加, 变形菌门相对丰度呈上升的趋势(p < 0.05), 在Cu浓度为1000 mg·kg-1时相对丰度最大(55.42%).而酸杆菌门和芽单胞菌门却表现出相反的趋势(p < 0.05), 在CK中最大, 其相对丰度分别从17.09%、6.84%降至4.06%、1.69%.
 |
| 图 3 门水平上土壤样品的细菌群落组成 Fig. 3 Bacterial community of soil samples at the level of phylum |
相对丰度≥1%的属共有30个, 本文以热图的形式呈现, 通过颜色的深浅将数据的大小直观地表现出来(图 4).随着Cu浓度的增加, 17个属的相对丰度降低(图 4A), 如芽孢杆菌属(Bacillus)、假单胞菌属(Pseudomonas)和Subgroup_6等;13个属的相对丰度升高(图 4B), 如溶杆菌属(Lysobacter)、黄杆菌属(Flavobacterium)和Brevundimonas等.
 |
| 图 4 属水平上土壤样品的细菌群落变化 (A簇为被Cu降低的属, B簇为被Cu富集的属) Fig. 4 Changes of soil bacterial community in genus |
荧光定量PCR结果表明, 目的基因在所有样品中均有检出.对于MRGs而言(图 5), 随着Cu浓度的增加其丰度呈上升的趋势, 当Cu浓度≥500 mg·kg-1时显著升高(p < 0.05), 在Cu浓度为1000 mg·kg-1时其丰度最大(7.35×106 copies·g-1), 达到CK的2.5倍.copA、copB和czcA这3种基因的丰度较高, 其中, copB占主导优势.copB和czcA的丰度随Cu浓度的升高而增加, 在Cu浓度为1000 mg·kg-1时最大(5.14×106和1.62×106 copies·g-1), 而copA的丰度则表现出相反的趋势, 在Cu浓度为1000 mg·kg-1时最小(4.07×105 copies·g-1).
 |
| 图 5 不同Cu浓度土壤的MRGs丰度 (不同小写字母表示各处理之间存在差异性差异, p < 0.05)) Fig. 5 The MRGs abundance in different Cu treatments |
随着Cu浓度的升高, 整合酶基因intⅠ1的丰度并没有规律性变化(图 6a), 在Cu浓度为750 mg·kg-1时整合酶基因intⅠ1的丰度最大(7.91×106 copies·g-1).而转座酶基因tnpA的丰度(图 6b)随着Cu浓度的升高表现出逐渐上升的趋势, 在Cu浓度为1000 mg·kg-1时转座酶基因tnpA的丰度最大(5.73×103 copies·g-1).
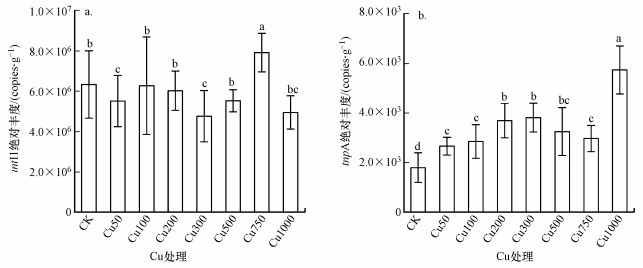 |
| 图 6 不同Cu浓度土壤的MGEs丰度 (不同小写字母表示各处理之间存在差异性差异, p < 0.05)) Fig. 6 The MRGs abundance in different Cu treatments |
Cu浓度与Cu抗性基因均具有显著相关性(图 7), copB(p < 0.001)和cueO(p < 0.05)显著正相关, 而copA与pcoA呈显著负相关(p < 0.001).对于多金属抗性基因, 只有czcA、czcD与Cu浓度表现出显著正相关(p < 0.001).tnpA与Cu浓度之间显著正相关(p < 0.001), 并且与copB、czcA和czcD之间也存在显著正相关关系(p < 0.001).反观intI1与Cu浓度无显著相关性.此外, 本研究还发现copA与pcoA之间存在显著正相关关系(p < 0.001), copB、czcA和czcD之间也存在显著正相关关系(p < 0.001).
 |
| 图 7 Cu、MRGs和MGEs之间的相关性 (*p < 0.05, **p < 0.01, ***p < 0.001) Fig. 7 The correlation analysis between concentration of Cu, MGEs and MRGs |
通过网络分析探究细菌群落和抗性基因之间的关系(图 8), 生成的网络图由39个节点(29个属、9个MRGs和1个MGEs)和123个边组成.在属水平上对抗性基因的潜在宿主进行分析.同一模块中的MRGs、MGEs和细菌群落可能在相同的环境压力下共现.如果MRGs与共现的微生物类群之间存在着强而显著的正相关(p < 0.05), 那么MRGs与微生物类群之间的非随机共现模式可以说明MRGs可能的宿主信息.本研究结果表明, MRGs与不同属之间存在着密切的相关性(p < 0.05), 并且被明确的分为4个模块, 其中主要为MRGs与降低属和MRGs与富集属这两个模块, 这两个模块的属可能是MRGs的潜在宿主.例如, 降低属中的假单胞菌属(Pseudomonas)、芽孢菌属(Bacillus)和鞘脂单孢菌属(Sphingomonas)等可能是copA和pcoA的潜在宿主, 富集属中的溶杆菌属(Lysobacter)、黄杆菌属(Flavobacterium)和地杆菌属(Pedobacter)等可能是copB、czcA和tnpA的潜在宿主.
 |
| 图 8 细菌群落与抗性基因的共现性网络分析 (边缘表示相邻节点之间显著性的正相关关系(p < 0.05);每个节点的大小与节点间显著相关性的数量成正比) Fig. 8 Network analysis of the correlations among the bacterial community, MRGs and MGEs |
随Cu浓度的增加细菌丰度表现出先增后减的单峰趋势(图 1), 在Cu浓度为100 mg·kg-1时达到最大值, 当Cu浓度≥500 mg·kg-1时, 细菌丰度显著小于CK(p < 0.05).这说明低浓度Cu可促进细菌生长, 而高浓度Cu则有抑制作用, 这与杨元根等(2002)的研究结果相似.本研究中Alpha多样性指数同样表现出先增后减的单峰趋势(表 2), 并在Cu浓度为100 mg·kg-1时最大.谢学辉等(2012)的研究也认为中等浓度重金属污染的土壤中微生物多样性最高, 不同重金属浓度对微生物多样性的影响可能并不是简单地线性增长或者降低, 与本研究结果相似.
为进一步探讨Cu污染对土壤细菌的影响, 分析了细菌群落结构的变化.在门水平上, 优势菌门基本相似, 均为变形菌门、拟杆菌门、放线菌门、酸杆菌门和厚壁菌门等, 它们在重金属污染土壤中分布广泛(Harichová et al., 2012;Chodak et al., 2013).随着Cu浓度的升高, 变形菌门的相对丰度呈上升趋势, 而酸杆菌门和芽孢杆菌门却表现出相反的趋势, 这与许多研究结果相似(Gillan et al., 2005; Tipayno et al., 2018; Song et al., 2018).这可能是由于变形菌门作为革兰氏阴性菌, 其特征是细胞壁外存在一层有能力将金属从细胞外分离的脂多糖层, 从而可以适应恶劣的污染环境(Pereira et al., 2006), 而酸杆菌门和芽单胞菌门则缺少这层脂多糖层.在属水平上, 芽孢杆菌属、假单胞杆菌属和Subgroup_6等17个属的相对丰度被降低.相关研究表明, 芽孢杆菌属和假单胞杆菌属对重金属有一定的耐受性(苏少华等, 2011), 而本研究结果与之相反.这可能是因为芽孢杆菌属和假单胞杆菌属在一定的重金属胁迫下具有抗性, 而超过其阈值后重金属也会对其产生毒害作用.在高浓度Cu条件下, 溶杆菌属和黄杆菌属等13个属的相对丰度升高, 表明这些属对Cu具有较好的耐受性, 相关研究也证明溶杆菌属和黄杆菌属对Cu具有一定的耐受性(Li et al., 2015; Bhat et al., 2020).
当Cu浓度≥500 mg·kg-1时, MRGs的丰度显著增加, 在Cu浓度为1000 mg·kg-1时MRGs的丰度最大且显著高于CK(p < 0.05), 这与之前的研究结果相似(Chen et al., 2019), 表明高浓度污染会诱导更多细菌抗性基因的表达.copA、copB和czcA这3种基因的丰度较高, 其中copB占主导优势, 并且与Cu呈显著正相关(p < 0.001).这是因为细胞内Cu浓度升高时, copB参与的外排泵系统可以对细胞进行解毒(Bissig et al., 2001).copA的大量表达有助于提高微生物对Cu的耐受性(Costa et al., 2012).有研究发现高浓度Cu污染下copA丰度最高(师碧玲等, 2019), 同时, 也有研究发现随Cu浓度的升高copA丰度降低(Besaury et al., 2016).本研究结果与后者相似, 这可能是因为Cu浓度的升高对一部分微生物的毒害作用增加, 并导致某些携带copA的微生物消失, 进而导致copA丰度降低.此外发现, 对于多金属抗性基因czcA, 其丰度随Cu浓度的增加而升高, 与Cu浓度呈显著正相关(p < 0.001), 并且czcA与copB和tnpA均呈显著正相关(p < 0.001), 说明在单金属胁迫下, 细菌群落可以同时产生对不同污染物的抗性.这可能是抗性基因位于同一选择单元(质粒或转座子)所造成的(Durrant et al., 2019), 这与Stéphanie等(2014)的研究结果相同.
重金属抗性基因潜在宿主分析发现, 相同模块中, 抗性基因与细菌群落随Cu浓度的升高变化趋势相同(图 8), 其中, 假单胞菌属、芽孢杆菌属、鞘脂单孢菌属可能为copA和pcoA的潜在宿主(Granja-Travez et al., 2018), 溶杆菌属、黄杆菌属和地杆菌属可能是copB和czcA的潜在宿主(Zhao et al., 2021).许多研究表明, 细菌群落结构是细菌抗性基因分布重要的决定因素, 当重金属对细菌产生毒害或是富集作用时, 细菌所携带的抗性基因也会升高或降低(Su et al., 2015; Liu et al., 2019).因此, 本研究进一步证实了copA丰度的减少与其潜在宿主的降低有关.copB、czcA丰度的升高可能是由于溶杆菌属、黄杆菌属和地杆菌属等属的相对丰度的升高.此外, 我们还发现copB、czcA存在多个共同的潜在宿主(Yang et al., 2019), 这进一步证明了Cu污染在引起抗Cu基因变化的同时, 也会引起多抗基因丰度的改变.
5 结论(Conclusions)1) Cu浓度≤100 mg·kg-1时, 细菌丰度和群落多样性升高, Cu浓度≥500 mg·kg-1时细菌丰度和群落多样性显著降低.
2) Cu浓度的增加使变形菌门的相对丰度升高(p < 0.05), 酸杆菌门和芽单胞菌门的相对丰度降低(p < 0.05);在属水平上, 溶杆菌属、黄杆菌属和Brevundimonas等13个属的相对丰度升高, 芽孢杆菌属、假单胞菌属和Subgroup_6等17个属的相对丰度降低.
3) Cu浓度≥500 mg·kg-1时, 微宇宙中重金属抗性基因的丰度显著升高.相关性分析和共现分析表明, Cu可以通过影响其潜在宿主来间接影响抗性基因的丰度变化.
Albarracin V H, Amoroso M J, Abate C M, et al.2005.Isolation and characterization of indigenous copper-resistant actinomycete strains[R].Germany: Friedrich-Schiller Univ.145-156
|
Besaury L, Pawlak B, Quillet L. 2016. Expression of copper-resistance genes in microbial communities under copper stress and oxic/anoxic conditions[J]. Environmental Science and Pollution Research, 23(5): 4013-4023. DOI:10.1007/s11356-014-3254-4 |
Bhat S A, Cui G, Li W, et al. 2020. Effect of heavy metals on the performance and bacterial profiles of activated sludge in a semi-continuous reactor[J]. Chemosphere, 241: 125035. DOI:10.1016/j.chemosphere.2019.125035 |
Bissig K D, Voegelin T C, Solioz M. 2001. Tetrathiomolybdate inhibition of the Enterococcus hirae copB copper ATPase[J]. FEBS Lett, 507(3): 367-370. DOI:10.1016/S0014-5793(01)03009-5 |
Bouskill N J, Barnhart E P, Galloway T S, et al. 2007. Quantification of changing Pseudomonas aeruginosa sodA, htpX and mt gene abundance in response to trace metal toxicity: a potential in situ biomarker of environmental health[J]. FEMS Microbiol Ecol, 60(2): 276-286. DOI:10.1111/j.1574-6941.2007.00296.x |
Cai L, Wang Q, Luo J, et al. 2019. Heavy metal contamination and health risk assessment for children near a large Cu-smelter in central China[J]. Science of the Total Environment, 650: 725-733. DOI:10.1016/j.scitotenv.2018.09.081 |
Cayol J L, Ollivier B, Alazard D, et al. 2015. The Extreme Conditions of Life on the Planet and Exobiology[M]. Netherlands: Springer.
|
Cervantes C, Gutierrez-Corona F. 1994. Copper resistance mechanisms in bacteria and fungi[J]. FEMS Microbiology Reviews, 14(2): 121-137. DOI:10.1111/j.1574-6976.1994.tb00083.x |
Chen J, He F, Zhang X, et al. 2014. Heavy metal pollution decreases microbial abundance, diversity and activity within particle-size fractions of a paddy soil[J]. FEMS Microbiology Ecology, 87(1): 164-181. DOI:10.1111/1574-6941.12212 |
Chen J, Li J, Zhang H, et al. 2019. Bacterial heavy-metal and antibiotic resistance genes in a copper tailing dam area in Northern China[J]. Frontiers in Microbiology. DOI:10.3389/fmicb.2019.01916 |
Chen J, Zhang H, Li J, et al. 2020. The toxic factor of copper should be adjusted during the ecological risk assessment for soil bacterial community[J]. Ecological Indicators, 111: 106072. DOI:10.1016/j.ecolind.2020.106072 |
Chen Y, Jiang Y, Huang H, et al. 2018. Long-term and high-concentration heavy-metal contamination strongly influences the microbiome and functional genes in Yellow River sediments[J]. Science of the Total Environment, 637-638: 1400-1412. DOI:10.1016/j.scitotenv.2018.05.109 |
Chodak M, Gołębiewski M, Morawska-Płoskonka J, et al. 2013. Diversity of microorganisms from forest soils differently polluted with heavy metals[J]. Applied Soil Ecology, 64: 7-14. DOI:10.1016/j.apsoil.2012.11.004 |
Choudhary M, Jetley U K, Abash Khan M, et al. 2007. Effect of heavy metal stress on proline, malondialdehyde, and superoxide dismutase activity in the cyanobacterium Spirulina platensis-S5[J]. Ecotoxicology and Environmental Safety, 66(2): 204-209. DOI:10.1016/j.ecoenv.2006.02.002 |
Costa J S D, Kothe E, Abate C M, et al. 2012. Unraveling the Amycolatopsis tucumanensis copper-resistome[J]. Biometals, 25(5): 905-917. DOI:10.1007/s10534-012-9557-3 |
Denef V J, Mueller R S, Banfield J F. 2010. AMD biofilms: using model communities to study microbial evolution and ecological complexity in nature[J]. The ISME Journal, 4(5): 599-610. DOI:10.1038/ismej.2009.158 |
Durrant M G, Li M M, Siranosian B A, et al. 2019. A bioinformatic analysis of integrative mobile genetic elements highlights their role in bacterial adaptation[J]. Cell Host & Microbe, 27(1): 140-153. |
Gillan D C, Danis B, Pernet P, et al. 2005. Structure of sediment-associated microbial communities along a heavy-metal contamination gradient in the marine environment[J]. Applied and Environmental Microbiology, 2(71): 679-690. |
Granja-Travez R S, Bugg T D H. 2018. Characterization of multicopper oxidase CopA from Pseudomonas putida KT2440 and Pseudomonas fluorescens Pf-5:Involvement in bacterial lignin oxidation[J]. Archives of Biochemistry and Biophysics, 660: 97-107. DOI:10.1016/j.abb.2018.10.012 |
Harichová J, Karelová E, Pangallo D, et al. 2012. Structure analysis of bacterial community and their heavy-metal resistance determinants in the heavy-metal-contaminated soil sample[J]. Biologia, 67(6): 1038-1048. DOI:10.2478/s11756-012-0123-9 |
Jansen E, Michels M, Til M, et al. 1994. Effects of heavy metals in soil on microbial diversity and activity as shown by the sensitivity-resistance index, an ecologically relevant parameter[J]. Biology and Fertility of soils, 3(17): 177-184. |
Li Q, Chen Y, Fu H, et al. 2012. Health risk of heavy metals in food crops grown on reclaimed tidal flat soil in the Pearl River Estuary, China[J]. Journal of Hazardous Materials, 227-228: 148-154. DOI:10.1016/j.jhazmat.2012.05.023 |
Li X, You F, Bond P L, et al. 2015. Establishing microbial diversity and functions in weathered and neutral Cu-Pb-Zn tailings with native soil addition[J]. Geoderma, 247-248: 108-116. DOI:10.1016/j.geoderma.2015.02.010 |
Lin Y, Ye Y, Hu Y, et al. 2019. The variation in microbial community structure under different heavy metal contamination levels in paddy soils[J]. Ecotoxicology and Environmental Safety, 180: 557-564. DOI:10.1016/j.ecoenv.2019.05.057 |
Liu H, Sun H, Zhang M, et al. 2019. Dynamics of microbial community and tetracycline resistance genes in biological nutrient removal process[J]. Journal of Environmental Management, 238(15): 84-91. |
Pereira S I A, Lima A I G, de Almeida Paula Figueira E M. 2006. Screening possible mechanisms mediating cadmium resistance in Rhizobium leguminosarum bv.viciae isolated from contaminated Portuguese soils[J]. Microbial Ecology, 52(2): 176-186. DOI:10.1007/s00248-006-9057-5 |
Roosa S, Wattiez R, Prygiel E, et al. 2014. Bacterial metal resistance genes and metal bioavailability in contaminated sediments[J]. Environmental Pollution, 189: 143-151. DOI:10.1016/j.envpol.2014.02.031 |
师碧玲, 陈建文, 刘勇, 等. 2019. 铜尾矿库区土壤重金属及微生物重金属抗性基因空间分异特征[J]. 环境科学学报, 39(8): 2654-2661. |
施昊坤, 吴次芳, 张茂鑫, 等. 2020. 土地整治对工业区周边土壤微生物多样性和群落结构影响分析[J]. 环境科学学报, 40(1): 212-223. |
Smith M B, Rocha A M, Smillie C S, et al. 2015. Natural bacterial communities serve as quantitative geochemical biosensors[J]. MBIO. DOI:10.1128/mBio.00326-15 |
Song J, Shen Q, Wang L, et al. 2018. Effects of Cd, Cu, Zn and their combined action on microbial biomass and bacterial community structure[J]. Environmental Pollution, 243: 510-518. DOI:10.1016/j.envpol.2018.09.011 |
苏少华, 张玉秀, 朱凌峰, 等. 2011. 近十年我国耐重金属细菌研究文献分析[J]. 农业图书情报学刊, 23(5): 63-67. |
Su J Q, Wei B, OuYang W Y, et al.015.Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J].Environmental Science & Technology, 49(12): 7356-7363
|
Tipayno S C, Truu J, Samaddar S, et al. 2018. The bacterial community structure and functional profile in the heavy metal contaminated paddy soils, surrounding a nonferrous smelter in South Korea[J]. Ecology and Evolution, 8(12): 6157-6168. DOI:10.1002/ece3.4170 |
Umrania V V. 2006. Bioremediation of toxic heavy metals using acidothermophilic autotrophes[J]. Bioresource Technology, 97(10): 1237-1242. DOI:10.1016/j.biortech.2005.04.048 |
谢学辉, 范凤霞, 袁学武, 等. 2012. 德兴铜矿尾矿重金属污染对土壤中微生物多样性的影响[J]. 微生物学通报, 39(5): 624-637. |
杨元根, Paterson E, Campbell C. 2002. 重金属Cu的土壤微生物毒性研究[J]. 土壤通报, (2): 137-141. |
Yang Y, Liu G, Song W, et al. 2019. Plastics in the marine environment are reservoirs for antibiotic and metal resistance genes[J]. Environment International, 123: 79-86. DOI:10.1016/j.envint.2018.11.061 |
赵杰, 罗志军, 赵越, 等. 2018. 环鄱阳湖区农田土壤重金属空间分布及污染评价[J]. 环境科学学报, 38(6): 2475-2485. |
Zhao Y, Gao J, Wang Z, et al. 2021. Responses of bacterial communities and resistance genes on microplastics to antibiotics and heavy metals in sewage environment[J]. Journal of Hazardous Materials, 402: 123550. DOI:10.1016/j.jhazmat.2020.123550 |
 2021, Vol. 41
2021, Vol. 41


